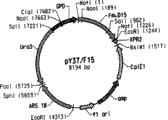
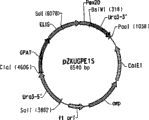
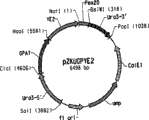
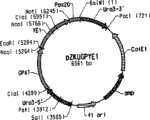
CN101437952A - 解脂耶氏酵母的高花生四烯酸酸生产菌株 - Google Patents
解脂耶氏酵母的高花生四烯酸酸生产菌株 Download PDFInfo
- Publication number
- CN101437952A CN101437952A CNA2005800459100A CN200580045910A CN101437952A CN 101437952 A CN101437952 A CN 101437952A CN A2005800459100 A CNA2005800459100 A CN A2005800459100A CN 200580045910 A CN200580045910 A CN 200580045910A CN 101437952 A CN101437952 A CN 101437952A
- Authority
- CN
- China
- Prior art keywords
- gene
- desaturases
- enzyme
- acid
- seq
- Prior art date
- Legal status (The legal status is an assumption and is not a legal conclusion. Google has not performed a legal analysis and makes no representation as to the accuracy of the status listed.)
- Pending
Links
- 241000235015 Yarrowia lipolytica Species 0.000 title abstract description 39
- 229960005135 eicosapentaenoic acid Drugs 0.000 title description 2
- JAZBEHYOTPTENJ-JLNKQSITSA-N all-cis-5,8,11,14,17-icosapentaenoic acid Chemical compound CC\C=C/C\C=C/C\C=C/C\C=C/C\C=C/CCCC(O)=O JAZBEHYOTPTENJ-JLNKQSITSA-N 0.000 title 1
- JAZBEHYOTPTENJ-UHFFFAOYSA-N eicosapentaenoic acid Natural products CCC=CCC=CCC=CCC=CCC=CCCCC(O)=O JAZBEHYOTPTENJ-UHFFFAOYSA-N 0.000 title 1
- 235000020673 eicosapentaenoic acid Nutrition 0.000 title 1
- 108090000623 proteins and genes Proteins 0.000 claims abstract description 393
- YZXBAPSDXZZRGB-DOFZRALJSA-N arachidonic acid Chemical compound CCCCC\C=C/C\C=C/C\C=C/C\C=C/CCCC(O)=O YZXBAPSDXZZRGB-DOFZRALJSA-N 0.000 claims abstract description 326
- 240000004808 Saccharomyces cerevisiae Species 0.000 claims abstract description 253
- 235000021342 arachidonic acid Nutrition 0.000 claims abstract description 164
- 229940114079 arachidonic acid Drugs 0.000 claims abstract description 164
- 238000000034 method Methods 0.000 claims abstract description 100
- 238000004519 manufacturing process Methods 0.000 claims abstract description 71
- 108700016155 Acyl transferases Proteins 0.000 claims abstract description 60
- 102000057234 Acyl transferases Human genes 0.000 claims abstract description 48
- 235000020665 omega-6 fatty acid Nutrition 0.000 claims abstract description 30
- 102000004190 Enzymes Human genes 0.000 claims description 240
- 108090000790 Enzymes Proteins 0.000 claims description 240
- 235000019197 fats Nutrition 0.000 claims description 195
- 150000002632 lipids Chemical class 0.000 claims description 103
- 235000013305 food Nutrition 0.000 claims description 95
- 239000002253 acid Substances 0.000 claims description 89
- 108010001348 Diacylglycerol O-acyltransferase Proteins 0.000 claims description 79
- 102000002148 Diacylglycerol O-acyltransferase Human genes 0.000 claims description 79
- 239000000047 product Substances 0.000 claims description 75
- 125000002252 acyl group Chemical group 0.000 claims description 61
- 102100034544 Acyl-CoA 6-desaturase Human genes 0.000 claims description 53
- 108010037138 Linoleoyl-CoA Desaturase Proteins 0.000 claims description 53
- 108010022240 delta-8 fatty acid desaturase Proteins 0.000 claims description 50
- 102100034542 Acyl-CoA (8-3)-desaturase Human genes 0.000 claims description 49
- 108010073542 Delta-5 Fatty Acid Desaturase Proteins 0.000 claims description 49
- 235000020660 omega-3 fatty acid Nutrition 0.000 claims description 46
- 239000000194 fatty acid Substances 0.000 claims description 43
- 230000000813 microbial effect Effects 0.000 claims description 42
- 102100038805 Lysophospholipid acyltransferase 2 Human genes 0.000 claims description 39
- VZCCETWTMQHEPK-QNEBEIHSSA-N gamma-linolenic acid Chemical compound CCCCC\C=C/C\C=C/C\C=C/CCCCC(O)=O VZCCETWTMQHEPK-QNEBEIHSSA-N 0.000 claims description 38
- 108090000765 processed proteins & peptides Proteins 0.000 claims description 37
- 230000006696 biosynthetic metabolic pathway Effects 0.000 claims description 36
- 235000020664 gamma-linolenic acid Nutrition 0.000 claims description 36
- 229920001184 polypeptide Polymers 0.000 claims description 32
- 102000004196 processed proteins & peptides Human genes 0.000 claims description 32
- 235000014113 dietary fatty acids Nutrition 0.000 claims description 30
- 229930195729 fatty acid Natural products 0.000 claims description 30
- 239000002773 nucleotide Substances 0.000 claims description 30
- 125000003729 nucleotide group Chemical group 0.000 claims description 30
- 241001465754 Metazoa Species 0.000 claims description 29
- DTOSIQBPPRVQHS-PDBXOOCHSA-N alpha-linolenic acid Chemical compound CC\C=C/C\C=C/C\C=C/CCCCCCCC(O)=O DTOSIQBPPRVQHS-PDBXOOCHSA-N 0.000 claims description 29
- -1 isopropyl ester Chemical class 0.000 claims description 28
- 235000020661 alpha-linolenic acid Nutrition 0.000 claims description 27
- 229960004488 linolenic acid Drugs 0.000 claims description 27
- 239000000126 substance Substances 0.000 claims description 27
- 108010052187 1-Acylglycerophosphocholine O-Acyltransferase Proteins 0.000 claims description 26
- 150000007523 nucleic acids Chemical class 0.000 claims description 24
- 229940012843 omega-3 fatty acid Drugs 0.000 claims description 24
- 125000003275 alpha amino acid group Chemical group 0.000 claims description 23
- 150000004665 fatty acids Chemical class 0.000 claims description 23
- 229940033080 omega-6 fatty acid Drugs 0.000 claims description 23
- 235000005911 diet Nutrition 0.000 claims description 22
- 235000013350 formula milk Nutrition 0.000 claims description 22
- 235000016709 nutrition Nutrition 0.000 claims description 22
- 230000037213 diet Effects 0.000 claims description 21
- 244000144974 aquaculture Species 0.000 claims description 19
- 102100034543 Fatty acid desaturase 3 Human genes 0.000 claims description 17
- 108010087894 Fatty acid desaturases Proteins 0.000 claims description 17
- 238000009360 aquaculture Methods 0.000 claims description 17
- 239000000463 material Substances 0.000 claims description 17
- 238000002360 preparation method Methods 0.000 claims description 17
- 102000039446 nucleic acids Human genes 0.000 claims description 14
- 108020004707 nucleic acids Proteins 0.000 claims description 14
- 150000001720 carbohydrates Chemical class 0.000 claims description 11
- 239000003814 drug Substances 0.000 claims description 11
- 230000000050 nutritive effect Effects 0.000 claims description 11
- 239000013589 supplement Substances 0.000 claims description 11
- 229940088594 vitamin Drugs 0.000 claims description 10
- 235000013343 vitamin Nutrition 0.000 claims description 10
- 239000011782 vitamin Substances 0.000 claims description 10
- 229930003231 vitamin Natural products 0.000 claims description 10
- 150000003722 vitamin derivatives Chemical class 0.000 claims description 10
- 230000035764 nutrition Effects 0.000 claims description 9
- 101710091951 Glycerol-3-phosphate acyltransferase Proteins 0.000 claims description 8
- 229910052500 inorganic mineral Inorganic materials 0.000 claims description 8
- 235000013622 meat product Nutrition 0.000 claims description 8
- 239000011707 mineral Substances 0.000 claims description 8
- 102000014384 Type C Phospholipases Human genes 0.000 claims description 5
- 108010079194 Type C Phospholipases Proteins 0.000 claims description 5
- 239000012620 biological material Substances 0.000 claims description 5
- VZCCETWTMQHEPK-UHFFFAOYSA-N gamma-Linolensaeure Natural products CCCCCC=CCC=CCC=CCCCCC(O)=O VZCCETWTMQHEPK-UHFFFAOYSA-N 0.000 claims description 5
- 101710088194 Dehydrogenase Proteins 0.000 claims description 4
- 108010055012 Orotidine-5'-phosphate decarboxylase Proteins 0.000 claims description 4
- 235000013410 fast food Nutrition 0.000 claims description 4
- 229940098330 gamma linoleic acid Drugs 0.000 claims description 4
- 238000002156 mixing Methods 0.000 claims description 4
- ZIIUUSVHCHPIQD-UHFFFAOYSA-N 2,4,6-trimethyl-N-[3-(trifluoromethyl)phenyl]benzenesulfonamide Chemical compound CC1=CC(C)=CC(C)=C1S(=O)(=O)NC1=CC=CC(C(F)(F)F)=C1 ZIIUUSVHCHPIQD-UHFFFAOYSA-N 0.000 claims description 3
- OYHQOLUKZRVURQ-HZJYTTRNSA-N Linoleic acid Chemical group CCCCC\C=C/C\C=C/CCCCCCCC(O)=O OYHQOLUKZRVURQ-HZJYTTRNSA-N 0.000 claims description 3
- 102100037214 Orotidine 5'-phosphate decarboxylase Human genes 0.000 claims description 3
- 102000015439 Phospholipases Human genes 0.000 claims description 3
- 108010064785 Phospholipases Proteins 0.000 claims description 3
- 229960002733 gamolenic acid Drugs 0.000 claims description 3
- 235000011868 grain product Nutrition 0.000 claims description 3
- 229960004232 linoleic acid Drugs 0.000 claims description 3
- 244000144977 poultry Species 0.000 claims description 3
- XSXIVVZCUAHUJO-AVQMFFATSA-N (11e,14e)-icosa-11,14-dienoic acid Chemical compound CCCCC\C=C\C\C=C\CCCCCCCCCC(O)=O XSXIVVZCUAHUJO-AVQMFFATSA-N 0.000 claims description 2
- 235000021297 Eicosadienoic acid Nutrition 0.000 claims description 2
- 101100161758 Yarrowia lipolytica (strain CLIB 122 / E 150) POX3 gene Proteins 0.000 claims description 2
- IQLUYYHUNSSHIY-HZUMYPAESA-N eicosatetraenoic acid Chemical compound CCCCCCCCCCC\C=C\C=C\C=C\C=C\C(O)=O IQLUYYHUNSSHIY-HZUMYPAESA-N 0.000 claims description 2
- 238000004064 recycling Methods 0.000 claims description 2
- BBWMTEYXFFWPIF-CJBMEHDJSA-N (2e,4e,6e)-icosa-2,4,6-trienoic acid Chemical compound CCCCCCCCCCCCC\C=C\C=C\C=C\C(O)=O BBWMTEYXFFWPIF-CJBMEHDJSA-N 0.000 claims 1
- PIFPCDRPHCQLSJ-WYIJOVFWSA-N 4,8,12,15,19-Docosapentaenoic acid Chemical compound CC\C=C\CC\C=C\C\C=C\CC\C=C\CC\C=C\CCC(O)=O PIFPCDRPHCQLSJ-WYIJOVFWSA-N 0.000 claims 1
- 101150102441 ACO3 gene Proteins 0.000 claims 1
- PIFPCDRPHCQLSJ-UHFFFAOYSA-N Clupanodonic acid Natural products CCC=CCCC=CCC=CCCC=CCCC=CCCC(O)=O PIFPCDRPHCQLSJ-UHFFFAOYSA-N 0.000 claims 1
- 101710084373 Lipase 1 Proteins 0.000 claims 1
- 101710084378 Lipase 2 Proteins 0.000 claims 1
- 241000282849 Ruminantia Species 0.000 claims 1
- 101100433922 Solanum lycopersicum ACO4 gene Proteins 0.000 claims 1
- DVSZKTAMJJTWFG-UHFFFAOYSA-N docosa-2,4,6,8,10,12-hexaenoic acid Chemical compound CCCCCCCCCC=CC=CC=CC=CC=CC=CC(O)=O DVSZKTAMJJTWFG-UHFFFAOYSA-N 0.000 claims 1
- MBMBGCFOFBJSGT-KUBAVDMBSA-N docosahexaenoic acid Natural products CC\C=C/C\C=C/C\C=C/C\C=C/C\C=C/C\C=C/CCC(O)=O MBMBGCFOFBJSGT-KUBAVDMBSA-N 0.000 claims 1
- 239000006052 feed supplement Substances 0.000 claims 1
- 235000021290 n-3 DPA Nutrition 0.000 claims 1
- 238000009825 accumulation Methods 0.000 abstract description 19
- 230000015572 biosynthetic process Effects 0.000 abstract description 8
- 238000003786 synthesis reaction Methods 0.000 abstract description 5
- 102000045404 acyltransferase activity proteins Human genes 0.000 abstract description 4
- 108700014220 acyltransferase activity proteins Proteins 0.000 abstract description 4
- 235000014680 Saccharomyces cerevisiae Nutrition 0.000 description 231
- 239000003925 fat Substances 0.000 description 194
- 230000014509 gene expression Effects 0.000 description 150
- 210000004027 cell Anatomy 0.000 description 114
- 230000000694 effects Effects 0.000 description 102
- 239000003921 oil Substances 0.000 description 88
- 235000019198 oils Nutrition 0.000 description 88
- 241000907999 Mortierella alpina Species 0.000 description 69
- 235000020777 polyunsaturated fatty acids Nutrition 0.000 description 68
- 108020004705 Codon Proteins 0.000 description 65
- 239000013612 plasmid Substances 0.000 description 53
- 238000013459 approach Methods 0.000 description 51
- 239000000758 substrate Substances 0.000 description 49
- 239000000203 mixture Substances 0.000 description 48
- 238000009482 thermal adhesion granulation Methods 0.000 description 47
- 102100039239 Amidophosphoribosyltransferase Human genes 0.000 description 43
- 108010039224 Amidophosphoribosyltransferase Proteins 0.000 description 43
- 108020004414 DNA Proteins 0.000 description 43
- 230000008859 change Effects 0.000 description 41
- 230000001580 bacterial effect Effects 0.000 description 40
- 238000006243 chemical reaction Methods 0.000 description 37
- 102000053187 Glucuronidase Human genes 0.000 description 35
- 108010060309 Glucuronidase Proteins 0.000 description 35
- 238000006555 catalytic reaction Methods 0.000 description 31
- 244000005700 microbiome Species 0.000 description 31
- 241000235070 Saccharomyces Species 0.000 description 26
- 230000001851 biosynthetic effect Effects 0.000 description 25
- 230000012010 growth Effects 0.000 description 25
- KQQKGWQCNNTQJW-UHFFFAOYSA-N linolenic acid Natural products CC=CCCC=CCC=CCCCCCCCC(O)=O KQQKGWQCNNTQJW-UHFFFAOYSA-N 0.000 description 25
- 108091032973 (ribonucleotides)n+m Proteins 0.000 description 24
- 229910052799 carbon Inorganic materials 0.000 description 24
- WTJKGGKOPKCXLL-RRHRGVEJSA-N phosphatidylcholine Chemical compound CCCCCCCCCCCCCCCC(=O)OC[C@H](COP([O-])(=O)OCC[N+](C)(C)C)OC(=O)CCCCCCCC=CCCCCCCCC WTJKGGKOPKCXLL-RRHRGVEJSA-N 0.000 description 24
- 108020004999 messenger RNA Proteins 0.000 description 23
- 102000004169 proteins and genes Human genes 0.000 description 23
- 230000003570 biosynthesizing effect Effects 0.000 description 21
- 102000006602 glyceraldehyde-3-phosphate dehydrogenase Human genes 0.000 description 21
- 108020004445 glyceraldehyde-3-phosphate dehydrogenase Proteins 0.000 description 21
- 235000018102 proteins Nutrition 0.000 description 21
- HOBAELRKJCKHQD-UHFFFAOYSA-N (8Z,11Z,14Z)-8,11,14-eicosatrienoic acid Natural products CCCCCC=CCC=CCC=CCCCCCCC(O)=O HOBAELRKJCKHQD-UHFFFAOYSA-N 0.000 description 20
- 229940024606 amino acid Drugs 0.000 description 20
- 235000001014 amino acid Nutrition 0.000 description 20
- 150000001413 amino acids Chemical class 0.000 description 20
- HOBAELRKJCKHQD-QNEBEIHSSA-N dihomo-γ-linolenic acid Chemical compound CCCCC\C=C/C\C=C/C\C=C/CCCCCCC(O)=O HOBAELRKJCKHQD-QNEBEIHSSA-N 0.000 description 20
- 239000012634 fragment Substances 0.000 description 20
- 108700039691 Genetic Promoter Regions Proteins 0.000 description 19
- 108091028043 Nucleic acid sequence Proteins 0.000 description 19
- 238000004458 analytical method Methods 0.000 description 19
- 239000000523 sample Substances 0.000 description 19
- OKTJSMMVPCPJKN-UHFFFAOYSA-N Carbon Chemical compound [C] OKTJSMMVPCPJKN-UHFFFAOYSA-N 0.000 description 18
- 241000233866 Fungi Species 0.000 description 18
- 230000035508 accumulation Effects 0.000 description 18
- 108010011713 delta-15 desaturase Proteins 0.000 description 18
- 230000009483 enzymatic pathway Effects 0.000 description 18
- 230000008569 process Effects 0.000 description 18
- 230000014621 translational initiation Effects 0.000 description 18
- 108091026890 Coding region Proteins 0.000 description 17
- 238000013519 translation Methods 0.000 description 17
- 230000014616 translation Effects 0.000 description 17
- IJGRMHOSHXDMSA-UHFFFAOYSA-N Atomic nitrogen Chemical compound N#N IJGRMHOSHXDMSA-UHFFFAOYSA-N 0.000 description 16
- 108700026244 Open Reading Frames Proteins 0.000 description 16
- 238000013016 damping Methods 0.000 description 16
- 238000005516 engineering process Methods 0.000 description 16
- 230000002018 overexpression Effects 0.000 description 16
- 101710097496 Lysophospholipid acyltransferase Proteins 0.000 description 15
- 101710163746 Lysophospholipid acyltransferase 2 Proteins 0.000 description 15
- 239000012530 fluid Substances 0.000 description 15
- WRIDQFICGBMAFQ-UHFFFAOYSA-N (E)-8-Octadecenoic acid Natural products CCCCCCCCCC=CCCCCCCC(O)=O WRIDQFICGBMAFQ-UHFFFAOYSA-N 0.000 description 14
- 101710124165 1-acyl-sn-glycerol-3-phosphate acyltransferase Proteins 0.000 description 14
- LQJBNNIYVWPHFW-UHFFFAOYSA-N 20:1omega9c fatty acid Natural products CCCCCCCCCCC=CCCCCCCCC(O)=O LQJBNNIYVWPHFW-UHFFFAOYSA-N 0.000 description 14
- QSBYPNXLFMSGKH-UHFFFAOYSA-N 9-Heptadecensaeure Natural products CCCCCCCC=CCCCCCCCC(O)=O QSBYPNXLFMSGKH-UHFFFAOYSA-N 0.000 description 14
- 241000251468 Actinopterygii Species 0.000 description 14
- 239000005642 Oleic acid Substances 0.000 description 14
- ZQPPMHVWECSIRJ-UHFFFAOYSA-N Oleic acid Natural products CCCCCCCCC=CCCCCCCCC(O)=O ZQPPMHVWECSIRJ-UHFFFAOYSA-N 0.000 description 14
- 101710172946 Probable 1-acyl-sn-glycerol-3-phosphate acyltransferase Proteins 0.000 description 14
- 235000019688 fish Nutrition 0.000 description 14
- 230000002068 genetic effect Effects 0.000 description 14
- QXJSBBXBKPUZAA-UHFFFAOYSA-N isooleic acid Natural products CCCCCCCC=CCCCCCCCCC(O)=O QXJSBBXBKPUZAA-UHFFFAOYSA-N 0.000 description 14
- ZQPPMHVWECSIRJ-KTKRTIGZSA-N oleic acid Chemical compound CCCCCCCC\C=C/CCCCCCCC(O)=O ZQPPMHVWECSIRJ-KTKRTIGZSA-N 0.000 description 14
- PEDCQBHIVMGVHV-UHFFFAOYSA-N Glycerine Chemical compound OCC(O)CO PEDCQBHIVMGVHV-UHFFFAOYSA-N 0.000 description 13
- 108091081024 Start codon Proteins 0.000 description 13
- 230000001965 increasing effect Effects 0.000 description 13
- 239000003550 marker Substances 0.000 description 13
- 241001501873 Isochrysis galbana Species 0.000 description 12
- 239000000284 extract Substances 0.000 description 12
- IPCSVZSSVZVIGE-UHFFFAOYSA-N hexadecanoic acid Chemical compound CCCCCCCCCCCCCCCC(O)=O IPCSVZSSVZVIGE-UHFFFAOYSA-N 0.000 description 12
- 238000012545 processing Methods 0.000 description 12
- 238000011144 upstream manufacturing Methods 0.000 description 12
- 241000196324 Embryophyta Species 0.000 description 11
- 235000013351 cheese Nutrition 0.000 description 11
- 239000007788 liquid Substances 0.000 description 11
- 235000013372 meat Nutrition 0.000 description 11
- WQZGKKKJIJFFOK-GASJEMHNSA-N Glucose Natural products OC[C@H]1OC(O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-GASJEMHNSA-N 0.000 description 10
- 235000013339 cereals Nutrition 0.000 description 10
- 238000013461 design Methods 0.000 description 10
- 230000036541 health Effects 0.000 description 10
- 238000005984 hydrogenation reaction Methods 0.000 description 10
- 230000001105 regulatory effect Effects 0.000 description 10
- 229940081969 saccharomyces cerevisiae Drugs 0.000 description 10
- XLYOFNOQVPJJNP-UHFFFAOYSA-N water Substances O XLYOFNOQVPJJNP-UHFFFAOYSA-N 0.000 description 10
- SEHFUALWMUWDKS-UHFFFAOYSA-N 5-fluoroorotic acid Chemical compound OC(=O)C=1NC(=O)NC(=O)C=1F SEHFUALWMUWDKS-UHFFFAOYSA-N 0.000 description 9
- 229930182558 Sterol Natural products 0.000 description 9
- 230000000692 anti-sense effect Effects 0.000 description 9
- 239000002585 base Substances 0.000 description 9
- 210000000481 breast Anatomy 0.000 description 9
- 235000014633 carbohydrates Nutrition 0.000 description 9
- 238000011156 evaluation Methods 0.000 description 9
- 230000004927 fusion Effects 0.000 description 9
- 230000008676 import Effects 0.000 description 9
- 230000010354 integration Effects 0.000 description 9
- 230000002503 metabolic effect Effects 0.000 description 9
- 235000003702 sterols Nutrition 0.000 description 9
- 238000012360 testing method Methods 0.000 description 9
- 230000009466 transformation Effects 0.000 description 9
- 241000351920 Aspergillus nidulans Species 0.000 description 8
- 101150099000 EXPA1 gene Proteins 0.000 description 8
- 102100029095 Exportin-1 Human genes 0.000 description 8
- 102000001390 Fructose-Bisphosphate Aldolase Human genes 0.000 description 8
- 108010068561 Fructose-Bisphosphate Aldolase Proteins 0.000 description 8
- 102000004157 Hydrolases Human genes 0.000 description 8
- 108090000604 Hydrolases Proteins 0.000 description 8
- 241001344131 Magnaporthe grisea Species 0.000 description 8
- 108010091086 Recombinases Proteins 0.000 description 8
- 102000018120 Recombinases Human genes 0.000 description 8
- 101100119348 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) EXP1 gene Proteins 0.000 description 8
- 101100269618 Streptococcus pneumoniae serotype 4 (strain ATCC BAA-334 / TIGR4) aliA gene Proteins 0.000 description 8
- 230000003321 amplification Effects 0.000 description 8
- 239000002299 complementary DNA Substances 0.000 description 8
- 230000001066 destructive effect Effects 0.000 description 8
- 108700002148 exportin 1 Proteins 0.000 description 8
- 239000008103 glucose Substances 0.000 description 8
- 229910052757 nitrogen Inorganic materials 0.000 description 8
- 238000003199 nucleic acid amplification method Methods 0.000 description 8
- 230000006798 recombination Effects 0.000 description 8
- 238000005215 recombination Methods 0.000 description 8
- 238000011160 research Methods 0.000 description 8
- 229920002498 Beta-glucan Polymers 0.000 description 7
- 102000036181 Fatty Acid Elongases Human genes 0.000 description 7
- 108010058732 Fatty Acid Elongases Proteins 0.000 description 7
- 241000223195 Fusarium graminearum Species 0.000 description 7
- 241000235087 Lachancea kluyveri Species 0.000 description 7
- 241000221961 Neurospora crassa Species 0.000 description 7
- 108091034117 Oligonucleotide Proteins 0.000 description 7
- 238000012408 PCR amplification Methods 0.000 description 7
- 230000000295 complement effect Effects 0.000 description 7
- 230000032050 esterification Effects 0.000 description 7
- 238000005886 esterification reaction Methods 0.000 description 7
- 230000007246 mechanism Effects 0.000 description 7
- 239000002609 medium Substances 0.000 description 7
- 230000004060 metabolic process Effects 0.000 description 7
- 230000007935 neutral effect Effects 0.000 description 7
- 235000008935 nutritious Nutrition 0.000 description 7
- 230000008521 reorganization Effects 0.000 description 7
- 239000000243 solution Substances 0.000 description 7
- 241000894007 species Species 0.000 description 7
- 238000013518 transcription Methods 0.000 description 7
- 230000035897 transcription Effects 0.000 description 7
- 235000021122 unsaturated fatty acids Nutrition 0.000 description 7
- 150000004670 unsaturated fatty acids Chemical class 0.000 description 7
- 210000005253 yeast cell Anatomy 0.000 description 7
- FYGDTMLNYKFZSV-URKRLVJHSA-N (2s,3r,4s,5s,6r)-2-[(2r,4r,5r,6s)-4,5-dihydroxy-2-(hydroxymethyl)-6-[(2r,4r,5r,6s)-4,5,6-trihydroxy-2-(hydroxymethyl)oxan-3-yl]oxyoxan-3-yl]oxy-6-(hydroxymethyl)oxane-3,4,5-triol Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@H]1OC1[C@@H](CO)O[C@@H](OC2[C@H](O[C@H](O)[C@H](O)[C@H]2O)CO)[C@H](O)[C@H]1O FYGDTMLNYKFZSV-URKRLVJHSA-N 0.000 description 6
- PORPENFLTBBHSG-MGBGTMOVSA-N 1,2-dihexadecanoyl-sn-glycerol-3-phosphate Chemical compound CCCCCCCCCCCCCCCC(=O)OC[C@H](COP(O)(O)=O)OC(=O)CCCCCCCCCCCCCCC PORPENFLTBBHSG-MGBGTMOVSA-N 0.000 description 6
- 101150069620 ARE2 gene Proteins 0.000 description 6
- 241001225321 Aspergillus fumigatus Species 0.000 description 6
- 241000894006 Bacteria Species 0.000 description 6
- 102000053602 DNA Human genes 0.000 description 6
- 102000013444 Diacylglycerol Cholinephosphotransferase Human genes 0.000 description 6
- 108010051225 Diacylglycerol cholinephosphotransferase Proteins 0.000 description 6
- 241000195619 Euglena gracilis Species 0.000 description 6
- OKKJLVBELUTLKV-UHFFFAOYSA-N Methanol Chemical compound OC OKKJLVBELUTLKV-UHFFFAOYSA-N 0.000 description 6
- 241000235575 Mortierella Species 0.000 description 6
- 241000700157 Rattus norvegicus Species 0.000 description 6
- 235000021355 Stearic acid Nutrition 0.000 description 6
- 229940091771 aspergillus fumigatus Drugs 0.000 description 6
- 230000008901 benefit Effects 0.000 description 6
- 238000004422 calculation algorithm Methods 0.000 description 6
- 150000001721 carbon Chemical group 0.000 description 6
- KRKNYBCHXYNGOX-UHFFFAOYSA-N citric acid Chemical compound OC(=O)CC(O)(C(O)=O)CC(O)=O KRKNYBCHXYNGOX-UHFFFAOYSA-N 0.000 description 6
- 230000000052 comparative effect Effects 0.000 description 6
- 235000015872 dietary supplement Nutrition 0.000 description 6
- 238000006911 enzymatic reaction Methods 0.000 description 6
- 238000006062 fragmentation reaction Methods 0.000 description 6
- 239000001963 growth medium Substances 0.000 description 6
- 235000020256 human milk Nutrition 0.000 description 6
- 238000011534 incubation Methods 0.000 description 6
- YAFQFNOUYXZVPZ-UHFFFAOYSA-N liproxstatin-1 Chemical compound ClC1=CC=CC(CNC=2C3(CCNCC3)NC3=CC=CC=C3N=2)=C1 YAFQFNOUYXZVPZ-UHFFFAOYSA-N 0.000 description 6
- 235000013336 milk Nutrition 0.000 description 6
- 239000008267 milk Substances 0.000 description 6
- 210000004080 milk Anatomy 0.000 description 6
- 235000010755 mineral Nutrition 0.000 description 6
- QIQXTHQIDYTFRH-UHFFFAOYSA-N octadecanoic acid Chemical compound CCCCCCCCCCCCCCCCCC(O)=O QIQXTHQIDYTFRH-UHFFFAOYSA-N 0.000 description 6
- OQCDKBAXFALNLD-UHFFFAOYSA-N octadecanoic acid Natural products CCCCCCCC(C)CCCCCCCCC(O)=O OQCDKBAXFALNLD-UHFFFAOYSA-N 0.000 description 6
- 239000000843 powder Substances 0.000 description 6
- 239000002243 precursor Substances 0.000 description 6
- 238000003753 real-time PCR Methods 0.000 description 6
- 229920006395 saturated elastomer Polymers 0.000 description 6
- 150000004671 saturated fatty acids Chemical class 0.000 description 6
- 238000002741 site-directed mutagenesis Methods 0.000 description 6
- 239000008117 stearic acid Substances 0.000 description 6
- UFTFJSFQGQCHQW-UHFFFAOYSA-N triformin Chemical compound O=COCC(OC=O)COC=O UFTFJSFQGQCHQW-UHFFFAOYSA-N 0.000 description 6
- HSHNITRMYYLLCV-UHFFFAOYSA-N 4-methylumbelliferone Chemical compound C1=C(O)C=CC2=C1OC(=O)C=C2C HSHNITRMYYLLCV-UHFFFAOYSA-N 0.000 description 5
- 102100036869 Diacylglycerol O-acyltransferase 1 Human genes 0.000 description 5
- 241000588724 Escherichia coli Species 0.000 description 5
- 102000002754 Glycerol-3-Phosphate O-Acyltransferase Human genes 0.000 description 5
- 108010018837 Glycerol-3-Phosphate O-Acyltransferase Proteins 0.000 description 5
- 108090001060 Lipase Proteins 0.000 description 5
- 241000255640 Loa loa Species 0.000 description 5
- 241000223252 Rhodotorula Species 0.000 description 5
- 108010073771 Soybean Proteins Proteins 0.000 description 5
- 102000001494 Sterol O-Acyltransferase Human genes 0.000 description 5
- 108010054082 Sterol O-acyltransferase Proteins 0.000 description 5
- 229940100389 Sulfonylurea Drugs 0.000 description 5
- 230000033228 biological regulation Effects 0.000 description 5
- 235000009508 confectionery Nutrition 0.000 description 5
- 230000006378 damage Effects 0.000 description 5
- 230000001419 dependent effect Effects 0.000 description 5
- 230000029087 digestion Effects 0.000 description 5
- 201000010099 disease Diseases 0.000 description 5
- 208000037265 diseases, disorders, signs and symptoms Diseases 0.000 description 5
- 238000000855 fermentation Methods 0.000 description 5
- 230000004151 fermentation Effects 0.000 description 5
- 238000007421 fluorometric assay Methods 0.000 description 5
- 238000013467 fragmentation Methods 0.000 description 5
- 235000021588 free fatty acids Nutrition 0.000 description 5
- 238000012239 gene modification Methods 0.000 description 5
- 230000005017 genetic modification Effects 0.000 description 5
- 235000013617 genetically modified food Nutrition 0.000 description 5
- 235000011187 glycerol Nutrition 0.000 description 5
- 238000002744 homologous recombination Methods 0.000 description 5
- 230000006801 homologous recombination Effects 0.000 description 5
- 210000004251 human milk Anatomy 0.000 description 5
- 239000000543 intermediate Substances 0.000 description 5
- 230000004048 modification Effects 0.000 description 5
- 238000012986 modification Methods 0.000 description 5
- 238000002703 mutagenesis Methods 0.000 description 5
- 231100000350 mutagenesis Toxicity 0.000 description 5
- 238000000926 separation method Methods 0.000 description 5
- 150000003432 sterols Chemical class 0.000 description 5
- YROXIXLRRCOBKF-UHFFFAOYSA-N sulfonylurea Chemical class OC(=N)N=S(=O)=O YROXIXLRRCOBKF-UHFFFAOYSA-N 0.000 description 5
- 239000006228 supernatant Substances 0.000 description 5
- 238000012546 transfer Methods 0.000 description 5
- 229920001817 Agar Polymers 0.000 description 4
- QGZKDVFQNNGYKY-UHFFFAOYSA-O Ammonium Chemical compound [NH4+] QGZKDVFQNNGYKY-UHFFFAOYSA-O 0.000 description 4
- 241000244202 Caenorhabditis Species 0.000 description 4
- 108050004099 Diacylglycerol O-acyltransferase 1 Proteins 0.000 description 4
- 241000235395 Mucor Species 0.000 description 4
- NBIIXXVUZAFLBC-UHFFFAOYSA-N Phosphoric acid Chemical compound OP(O)(O)=O NBIIXXVUZAFLBC-UHFFFAOYSA-N 0.000 description 4
- 101000912235 Rebecca salina Acyl-lipid (7-3)-desaturase Proteins 0.000 description 4
- 101000877236 Siganus canaliculatus Acyl-CoA Delta-4 desaturase Proteins 0.000 description 4
- WQDUMFSSJAZKTM-UHFFFAOYSA-N Sodium methoxide Chemical compound [Na+].[O-]C WQDUMFSSJAZKTM-UHFFFAOYSA-N 0.000 description 4
- DRTQHJPVMGBUCF-XVFCMESISA-N Uridine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-XVFCMESISA-N 0.000 description 4
- JLCPHMBAVCMARE-UHFFFAOYSA-N [3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[3-[[3-[[3-[[3-[[3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-[[5-(2-amino-6-oxo-1H-purin-9-yl)-3-hydroxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxyoxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(5-methyl-2,4-dioxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(6-aminopurin-9-yl)oxolan-2-yl]methoxy-hydroxyphosphoryl]oxy-5-(4-amino-2-oxopyrimidin-1-yl)oxolan-2-yl]methyl [5-(6-aminopurin-9-yl)-2-(hydroxymethyl)oxolan-3-yl] hydrogen phosphate Polymers Cc1cn(C2CC(OP(O)(=O)OCC3OC(CC3OP(O)(=O)OCC3OC(CC3O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c3nc(N)[nH]c4=O)C(COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3COP(O)(=O)OC3CC(OC3CO)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3ccc(N)nc3=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cc(C)c(=O)[nH]c3=O)n3cc(C)c(=O)[nH]c3=O)n3ccc(N)nc3=O)n3cc(C)c(=O)[nH]c3=O)n3cnc4c3nc(N)[nH]c4=O)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)n3cnc4c(N)ncnc34)O2)c(=O)[nH]c1=O JLCPHMBAVCMARE-UHFFFAOYSA-N 0.000 description 4
- 239000008272 agar Substances 0.000 description 4
- 235000010419 agar Nutrition 0.000 description 4
- BFNBIHQBYMNNAN-UHFFFAOYSA-N ammonium sulfate Chemical compound N.N.OS(O)(=O)=O BFNBIHQBYMNNAN-UHFFFAOYSA-N 0.000 description 4
- 229910052921 ammonium sulfate Inorganic materials 0.000 description 4
- 235000011130 ammonium sulphate Nutrition 0.000 description 4
- 239000006227 byproduct Substances 0.000 description 4
- 210000004899 c-terminal region Anatomy 0.000 description 4
- 239000007795 chemical reaction product Substances 0.000 description 4
- 239000003153 chemical reaction reagent Substances 0.000 description 4
- 239000003795 chemical substances by application Substances 0.000 description 4
- 150000001875 compounds Chemical class 0.000 description 4
- 238000005520 cutting process Methods 0.000 description 4
- OPTASPLRGRRNAP-UHFFFAOYSA-N cytosine Chemical compound NC=1C=CNC(=O)N=1 OPTASPLRGRRNAP-UHFFFAOYSA-N 0.000 description 4
- 238000001514 detection method Methods 0.000 description 4
- 238000011161 development Methods 0.000 description 4
- 230000018109 developmental process Effects 0.000 description 4
- 238000010586 diagram Methods 0.000 description 4
- 229940079593 drug Drugs 0.000 description 4
- 230000002708 enhancing effect Effects 0.000 description 4
- 238000009472 formulation Methods 0.000 description 4
- 230000001744 histochemical effect Effects 0.000 description 4
- 230000001939 inductive effect Effects 0.000 description 4
- 230000000968 intestinal effect Effects 0.000 description 4
- QJGQUHMNIGDVPM-UHFFFAOYSA-N nitrogen group Chemical group [N] QJGQUHMNIGDVPM-UHFFFAOYSA-N 0.000 description 4
- 239000001301 oxygen Substances 0.000 description 4
- 229910052760 oxygen Inorganic materials 0.000 description 4
- SECPZKHBENQXJG-FPLPWBNLSA-N palmitoleic acid Chemical compound CCCCCC\C=C/CCCCCCCC(O)=O SECPZKHBENQXJG-FPLPWBNLSA-N 0.000 description 4
- 230000008488 polyadenylation Effects 0.000 description 4
- 238000003375 selectivity assay Methods 0.000 description 4
- 238000004088 simulation Methods 0.000 description 4
- 239000007787 solid Substances 0.000 description 4
- 238000001228 spectrum Methods 0.000 description 4
- DCXXMTOCNZCJGO-UHFFFAOYSA-N tristearoylglycerol Chemical compound CCCCCCCCCCCCCCCCCC(=O)OCC(OC(=O)CCCCCCCCCCCCCCCCC)COC(=O)CCCCCCCCCCCCCCCCC DCXXMTOCNZCJGO-UHFFFAOYSA-N 0.000 description 4
- FFRBMBIXVSCUFS-UHFFFAOYSA-N 2,4-dinitro-1-naphthol Chemical compound C1=CC=C2C(O)=C([N+]([O-])=O)C=C([N+]([O-])=O)C2=C1 FFRBMBIXVSCUFS-UHFFFAOYSA-N 0.000 description 3
- TWJNQYPJQDRXPH-UHFFFAOYSA-N 2-cyanobenzohydrazide Chemical compound NNC(=O)C1=CC=CC=C1C#N TWJNQYPJQDRXPH-UHFFFAOYSA-N 0.000 description 3
- NKDFYOWSKOHCCO-YPVLXUMRSA-N 20-hydroxyecdysone Chemical compound C1[C@@H](O)[C@@H](O)C[C@]2(C)[C@@H](CC[C@@]3([C@@H]([C@@](C)(O)[C@H](O)CCC(C)(O)C)CC[C@]33O)C)C3=CC(=O)[C@@H]21 NKDFYOWSKOHCCO-YPVLXUMRSA-N 0.000 description 3
- 101150040074 Aco2 gene Proteins 0.000 description 3
- 102100039338 Aminomethyltransferase, mitochondrial Human genes 0.000 description 3
- 108050001492 Ammonium transporters Proteins 0.000 description 3
- 101100011518 Arabidopsis thaliana ELP1 gene Proteins 0.000 description 3
- 241000972773 Aulopiformes Species 0.000 description 3
- 108010029692 Bisphosphoglycerate mutase Proteins 0.000 description 3
- 241000282472 Canis lupus familiaris Species 0.000 description 3
- 244000025254 Cannabis sativa Species 0.000 description 3
- CURLTUGMZLYLDI-UHFFFAOYSA-N Carbon dioxide Chemical compound O=C=O CURLTUGMZLYLDI-UHFFFAOYSA-N 0.000 description 3
- 102100023441 Centromere protein J Human genes 0.000 description 3
- 229920002101 Chitin Polymers 0.000 description 3
- 108700010070 Codon Usage Proteins 0.000 description 3
- 241000195493 Cryptophyta Species 0.000 description 3
- 108010042407 Endonucleases Proteins 0.000 description 3
- 102000004533 Endonucleases Human genes 0.000 description 3
- LFQSCWFLJHTTHZ-UHFFFAOYSA-N Ethanol Chemical compound CCO LFQSCWFLJHTTHZ-UHFFFAOYSA-N 0.000 description 3
- 101150095274 FBA1 gene Proteins 0.000 description 3
- JZNWSCPGTDBMEW-UHFFFAOYSA-N Glycerophosphorylethanolamin Natural products NCCOP(O)(=O)OCC(O)CO JZNWSCPGTDBMEW-UHFFFAOYSA-N 0.000 description 3
- 101000907924 Homo sapiens Centromere protein J Proteins 0.000 description 3
- 101000600189 Homo sapiens Peroxisomal membrane protein PEX16 Proteins 0.000 description 3
- 101000693082 Homo sapiens Serine/threonine-protein kinase 11-interacting protein Proteins 0.000 description 3
- ROHFNLRQFUQHCH-YFKPBYRVSA-N L-leucine Chemical compound CC(C)C[C@H](N)C(O)=O ROHFNLRQFUQHCH-YFKPBYRVSA-N 0.000 description 3
- ROHFNLRQFUQHCH-UHFFFAOYSA-N Leucine Natural products CC(C)CC(N)C(O)=O ROHFNLRQFUQHCH-UHFFFAOYSA-N 0.000 description 3
- 102000004882 Lipase Human genes 0.000 description 3
- 239000004367 Lipase Substances 0.000 description 3
- 108020002496 Lysophospholipase Proteins 0.000 description 3
- 101100005882 Mus musculus Cel gene Proteins 0.000 description 3
- 101100289046 Mus musculus Lias gene Proteins 0.000 description 3
- 235000021360 Myristic acid Nutrition 0.000 description 3
- TUNFSRHWOTWDNC-UHFFFAOYSA-N Myristic acid Natural products CCCCCCCCCCCCCC(O)=O TUNFSRHWOTWDNC-UHFFFAOYSA-N 0.000 description 3
- XUYPXLNMDZIRQH-LURJTMIESA-N N-acetyl-L-methionine Chemical compound CSCC[C@@H](C(O)=O)NC(C)=O XUYPXLNMDZIRQH-LURJTMIESA-N 0.000 description 3
- 108091092724 Noncoding DNA Proteins 0.000 description 3
- 108010068204 Peptide Elongation Factors Proteins 0.000 description 3
- 102000002508 Peptide Elongation Factors Human genes 0.000 description 3
- 102100037479 Peroxisomal membrane protein PEX16 Human genes 0.000 description 3
- 102100031538 Phosphatidylcholine-sterol acyltransferase Human genes 0.000 description 3
- 102000011025 Phosphoglycerate Mutase Human genes 0.000 description 3
- 102100037883 Phospholipase B1, membrane-associated Human genes 0.000 description 3
- 108090000553 Phospholipase D Proteins 0.000 description 3
- 101710133727 Phospholipid:diacylglycerol acyltransferase Proteins 0.000 description 3
- 241001494715 Porphyridium purpureum Species 0.000 description 3
- 108700008625 Reporter Genes Proteins 0.000 description 3
- 101100501248 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) ELO2 gene Proteins 0.000 description 3
- 108010052160 Site-specific recombinase Proteins 0.000 description 3
- 241001467333 Thraustochytriaceae Species 0.000 description 3
- 241000223230 Trichosporon Species 0.000 description 3
- 101150050575 URA3 gene Proteins 0.000 description 3
- CKUAXEQHGKSLHN-UHFFFAOYSA-N [C].[N] Chemical compound [C].[N] CKUAXEQHGKSLHN-UHFFFAOYSA-N 0.000 description 3
- 239000003513 alkali Substances 0.000 description 3
- 235000013361 beverage Nutrition 0.000 description 3
- 239000002775 capsule Substances 0.000 description 3
- 125000004432 carbon atom Chemical group C* 0.000 description 3
- 235000011089 carbon dioxide Nutrition 0.000 description 3
- 238000010835 comparative analysis Methods 0.000 description 3
- 230000001186 cumulative effect Effects 0.000 description 3
- 235000013365 dairy product Nutrition 0.000 description 3
- 230000009849 deactivation Effects 0.000 description 3
- 239000008121 dextrose Substances 0.000 description 3
- 238000004043 dyeing Methods 0.000 description 3
- 238000002474 experimental method Methods 0.000 description 3
- 235000011194 food seasoning agent Nutrition 0.000 description 3
- 230000002538 fungal effect Effects 0.000 description 3
- 238000007429 general method Methods 0.000 description 3
- 238000006460 hydrolysis reaction Methods 0.000 description 3
- 230000001976 improved effect Effects 0.000 description 3
- 230000006872 improvement Effects 0.000 description 3
- 235000021125 infant nutrition Nutrition 0.000 description 3
- 239000004615 ingredient Substances 0.000 description 3
- 230000005764 inhibitory process Effects 0.000 description 3
- 101150091094 lipA gene Proteins 0.000 description 3
- 235000019421 lipase Nutrition 0.000 description 3
- 230000006372 lipid accumulation Effects 0.000 description 3
- 239000002502 liposome Substances 0.000 description 3
- 235000020978 long-chain polyunsaturated fatty acids Nutrition 0.000 description 3
- 235000012054 meals Nutrition 0.000 description 3
- 239000012528 membrane Substances 0.000 description 3
- 229930182817 methionine Natural products 0.000 description 3
- 238000010369 molecular cloning Methods 0.000 description 3
- 235000015097 nutrients Nutrition 0.000 description 3
- 238000005457 optimization Methods 0.000 description 3
- 150000008104 phosphatidylethanolamines Chemical class 0.000 description 3
- 238000001556 precipitation Methods 0.000 description 3
- 238000004321 preservation Methods 0.000 description 3
- 150000003254 radicals Chemical class 0.000 description 3
- 108700012830 rat Lip2 Proteins 0.000 description 3
- 230000002829 reductive effect Effects 0.000 description 3
- 108091008146 restriction endonucleases Proteins 0.000 description 3
- 235000019515 salmon Nutrition 0.000 description 3
- 235000003441 saturated fatty acids Nutrition 0.000 description 3
- 238000004904 shortening Methods 0.000 description 3
- 229940001941 soy protein Drugs 0.000 description 3
- 238000003756 stirring Methods 0.000 description 3
- 238000003860 storage Methods 0.000 description 3
- 230000009469 supplementation Effects 0.000 description 3
- 239000003826 tablet Substances 0.000 description 3
- 231100000419 toxicity Toxicity 0.000 description 3
- 230000001988 toxicity Effects 0.000 description 3
- 230000002103 transcriptional effect Effects 0.000 description 3
- 238000005406 washing Methods 0.000 description 3
- GVJHHUAWPYXKBD-UHFFFAOYSA-N (±)-α-Tocopherol Chemical compound OC1=C(C)C(C)=C2OC(CCCC(C)CCCC(C)CCCC(C)C)(C)CCC2=C1C GVJHHUAWPYXKBD-UHFFFAOYSA-N 0.000 description 2
- LDVVTQMJQSCDMK-UHFFFAOYSA-N 1,3-dihydroxypropan-2-yl formate Chemical compound OCC(CO)OC=O LDVVTQMJQSCDMK-UHFFFAOYSA-N 0.000 description 2
- HVCOBJNICQPDBP-UHFFFAOYSA-N 3-[3-[3,5-dihydroxy-6-methyl-4-(3,4,5-trihydroxy-6-methyloxan-2-yl)oxyoxan-2-yl]oxydecanoyloxy]decanoic acid;hydrate Chemical group O.OC1C(OC(CC(=O)OC(CCCCCCC)CC(O)=O)CCCCCCC)OC(C)C(O)C1OC1C(O)C(O)C(O)C(C)O1 HVCOBJNICQPDBP-UHFFFAOYSA-N 0.000 description 2
- LODRRYMGPWQCTR-UHFFFAOYSA-N 5-fluoro-2,4-dioxo-1h-pyrimidine-6-carboxylic acid;hydrate Chemical compound O.OC(=O)C=1NC(=O)NC(=O)C=1F LODRRYMGPWQCTR-UHFFFAOYSA-N 0.000 description 2
- 108010000700 Acetolactate synthase Proteins 0.000 description 2
- 101100225658 Arabidopsis thaliana ELP4 gene Proteins 0.000 description 2
- 235000007319 Avena orientalis Nutrition 0.000 description 2
- 235000007558 Avena sp Nutrition 0.000 description 2
- 101000583086 Bunodosoma granuliferum Delta-actitoxin-Bgr2b Proteins 0.000 description 2
- MIMFUSIYQURABA-UHFFFAOYSA-N C=CCCC.[C] Chemical compound C=CCCC.[C] MIMFUSIYQURABA-UHFFFAOYSA-N 0.000 description 2
- 101100289888 Caenorhabditis elegans lys-5 gene Proteins 0.000 description 2
- 241000222122 Candida albicans Species 0.000 description 2
- 108091035707 Consensus sequence Proteins 0.000 description 2
- 241000238424 Crustacea Species 0.000 description 2
- 241001527609 Cryptococcus Species 0.000 description 2
- 101150042222 DGAT1 gene Proteins 0.000 description 2
- 108010014303 DNA-directed DNA polymerase Proteins 0.000 description 2
- 102000016928 DNA-directed DNA polymerase Human genes 0.000 description 2
- 241000238557 Decapoda Species 0.000 description 2
- 108091027757 Deoxyribozyme Proteins 0.000 description 2
- 101150102653 Dgat2 gene Proteins 0.000 description 2
- 239000004278 EU approved seasoning Substances 0.000 description 2
- 241001480508 Entomophthora Species 0.000 description 2
- 241000321428 Epinephelus guttatus Species 0.000 description 2
- 241000333045 Fusarium graminearum PH-1 Species 0.000 description 2
- 241000233732 Fusarium verticillioides Species 0.000 description 2
- 230000005526 G1 to G0 transition Effects 0.000 description 2
- 241000287828 Gallus gallus Species 0.000 description 2
- 108010010803 Gelatin Proteins 0.000 description 2
- 235000010469 Glycine max Nutrition 0.000 description 2
- 244000068988 Glycine max Species 0.000 description 2
- 101001130226 Homo sapiens Phosphatidylcholine-sterol acyltransferase Proteins 0.000 description 2
- 101000639970 Homo sapiens Sodium- and chloride-dependent GABA transporter 1 Proteins 0.000 description 2
- 108010044467 Isoenzymes Proteins 0.000 description 2
- WHUUTDBJXJRKMK-VKHMYHEASA-N L-glutamic acid Chemical compound OC(=O)[C@@H](N)CCC(O)=O WHUUTDBJXJRKMK-VKHMYHEASA-N 0.000 description 2
- 102100037611 Lysophospholipase Human genes 0.000 description 2
- PXHVJJICTQNCMI-UHFFFAOYSA-N Nickel Chemical compound [Ni] PXHVJJICTQNCMI-UHFFFAOYSA-N 0.000 description 2
- 101150059359 POX2 gene Proteins 0.000 description 2
- 102000011420 Phospholipase D Human genes 0.000 description 2
- OAICVXFJPJFONN-UHFFFAOYSA-N Phosphorus Chemical compound [P] OAICVXFJPJFONN-UHFFFAOYSA-N 0.000 description 2
- WCUXLLCKKVVCTQ-UHFFFAOYSA-M Potassium chloride Chemical compound [Cl-].[K+] WCUXLLCKKVVCTQ-UHFFFAOYSA-M 0.000 description 2
- ONIBWKKTOPOVIA-UHFFFAOYSA-N Proline Natural products OC(=O)C1CCCN1 ONIBWKKTOPOVIA-UHFFFAOYSA-N 0.000 description 2
- LCTONWCANYUPML-UHFFFAOYSA-N Pyruvic acid Chemical compound CC(=O)C(O)=O LCTONWCANYUPML-UHFFFAOYSA-N 0.000 description 2
- 241000233639 Pythium Species 0.000 description 2
- 108020004511 Recombinant DNA Proteins 0.000 description 2
- 101100118655 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) ELO1 gene Proteins 0.000 description 2
- FAPWRFPIFSIZLT-UHFFFAOYSA-M Sodium chloride Chemical compound [Na+].[Cl-] FAPWRFPIFSIZLT-UHFFFAOYSA-M 0.000 description 2
- 102100033927 Sodium- and chloride-dependent GABA transporter 1 Human genes 0.000 description 2
- 238000002105 Southern blotting Methods 0.000 description 2
- 241000269809 Sparus aurata Species 0.000 description 2
- 229920002472 Starch Polymers 0.000 description 2
- 229930006000 Sucrose Natural products 0.000 description 2
- CZMRCDWAGMRECN-UGDNZRGBSA-N Sucrose Chemical compound O[C@H]1[C@H](O)[C@@H](CO)O[C@@]1(CO)O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 CZMRCDWAGMRECN-UGDNZRGBSA-N 0.000 description 2
- NINIDFKCEFEMDL-UHFFFAOYSA-N Sulfur Chemical compound [S] NINIDFKCEFEMDL-UHFFFAOYSA-N 0.000 description 2
- 108700005078 Synthetic Genes Proteins 0.000 description 2
- 241000144181 Thraustochytrium aureum Species 0.000 description 2
- 241000306282 Umbelopsis isabellina Species 0.000 description 2
- XSQUKJJJFZCRTK-UHFFFAOYSA-N Urea Chemical compound NC(N)=O XSQUKJJJFZCRTK-UHFFFAOYSA-N 0.000 description 2
- 101900060519 Yarrowia lipolytica Glyceraldehyde-3-phosphate dehydrogenase Proteins 0.000 description 2
- 240000008042 Zea mays Species 0.000 description 2
- 235000005824 Zea mays ssp. parviglumis Nutrition 0.000 description 2
- 235000002017 Zea mays subsp mays Nutrition 0.000 description 2
- LPQOADBMXVRBNX-UHFFFAOYSA-N ac1ldcw0 Chemical compound Cl.C1CN(C)CCN1C1=C(F)C=C2C(=O)C(C(O)=O)=CN3CCSC1=C32 LPQOADBMXVRBNX-UHFFFAOYSA-N 0.000 description 2
- 150000001335 aliphatic alkanes Chemical class 0.000 description 2
- 229910000147 aluminium phosphate Inorganic materials 0.000 description 2
- 150000001412 amines Chemical class 0.000 description 2
- 230000003698 anagen phase Effects 0.000 description 2
- 239000003242 anti bacterial agent Substances 0.000 description 2
- 229940088710 antibiotic agent Drugs 0.000 description 2
- 235000021405 artificial diet Nutrition 0.000 description 2
- QVGXLLKOCUKJST-UHFFFAOYSA-N atomic oxygen Chemical compound [O] QVGXLLKOCUKJST-UHFFFAOYSA-N 0.000 description 2
- 238000002869 basic local alignment search tool Methods 0.000 description 2
- DRTQHJPVMGBUCF-PSQAKQOGSA-N beta-L-uridine Natural products O[C@H]1[C@@H](O)[C@H](CO)O[C@@H]1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-PSQAKQOGSA-N 0.000 description 2
- 230000008238 biochemical pathway Effects 0.000 description 2
- 238000009835 boiling Methods 0.000 description 2
- 235000008429 bread Nutrition 0.000 description 2
- 235000014121 butter Nutrition 0.000 description 2
- 229940095731 candida albicans Drugs 0.000 description 2
- 229940041514 candida albicans extract Drugs 0.000 description 2
- 210000002421 cell wall Anatomy 0.000 description 2
- 239000001913 cellulose Substances 0.000 description 2
- 229920002678 cellulose Polymers 0.000 description 2
- 235000010980 cellulose Nutrition 0.000 description 2
- 229960001231 choline Drugs 0.000 description 2
- OEYIOHPDSNJKLS-UHFFFAOYSA-N choline Chemical compound C[N+](C)(C)CCO OEYIOHPDSNJKLS-UHFFFAOYSA-N 0.000 description 2
- 230000002759 chromosomal effect Effects 0.000 description 2
- 238000010367 cloning Methods 0.000 description 2
- 238000010276 construction Methods 0.000 description 2
- 230000001276 controlling effect Effects 0.000 description 2
- 235000005822 corn Nutrition 0.000 description 2
- GHVNFZFCNZKVNT-UHFFFAOYSA-N decanoic acid Chemical compound CCCCCCCCCC(O)=O GHVNFZFCNZKVNT-UHFFFAOYSA-N 0.000 description 2
- 230000007423 decrease Effects 0.000 description 2
- CYQFCXCEBYINGO-IAGOWNOFSA-N delta1-THC Chemical compound C1=C(C)CC[C@H]2C(C)(C)OC3=CC(CCCCC)=CC(O)=C3[C@@H]21 CYQFCXCEBYINGO-IAGOWNOFSA-N 0.000 description 2
- XBDQKXXYIPTUBI-UHFFFAOYSA-N dimethylselenoniopropionate Natural products CCC(O)=O XBDQKXXYIPTUBI-UHFFFAOYSA-N 0.000 description 2
- 230000008034 disappearance Effects 0.000 description 2
- POULHZVOKOAJMA-UHFFFAOYSA-N dodecanoic acid Chemical compound CCCCCCCCCCCC(O)=O POULHZVOKOAJMA-UHFFFAOYSA-N 0.000 description 2
- 239000012636 effector Substances 0.000 description 2
- 150000002066 eicosanoids Chemical class 0.000 description 2
- 210000002472 endoplasmic reticulum Anatomy 0.000 description 2
- 150000002148 esters Chemical class 0.000 description 2
- 238000000605 extraction Methods 0.000 description 2
- 230000002349 favourable effect Effects 0.000 description 2
- 101150108901 fbaA gene Proteins 0.000 description 2
- 239000004459 forage Substances 0.000 description 2
- 239000013505 freshwater Substances 0.000 description 2
- IFYYFLINQYPWGJ-UHFFFAOYSA-N gamma-decalactone Chemical compound CCCCCCC1CCC(=O)O1 IFYYFLINQYPWGJ-UHFFFAOYSA-N 0.000 description 2
- 239000000499 gel Substances 0.000 description 2
- 239000008273 gelatin Substances 0.000 description 2
- 229920000159 gelatin Polymers 0.000 description 2
- 235000019322 gelatine Nutrition 0.000 description 2
- 235000011852 gelatine desserts Nutrition 0.000 description 2
- 239000007897 gelcap Substances 0.000 description 2
- 239000011521 glass Substances 0.000 description 2
- JFCQEDHGNNZCLN-UHFFFAOYSA-N glutaric acid Chemical compound OC(=O)CCCC(O)=O JFCQEDHGNNZCLN-UHFFFAOYSA-N 0.000 description 2
- 150000004676 glycans Chemical class 0.000 description 2
- UYTPUPDQBNUYGX-UHFFFAOYSA-N guanine Chemical compound O=C1NC(N)=NC2=C1N=CN2 UYTPUPDQBNUYGX-UHFFFAOYSA-N 0.000 description 2
- 241000411851 herbal medicine Species 0.000 description 2
- 230000007062 hydrolysis Effects 0.000 description 2
- VKOBVWXKNCXXDE-UHFFFAOYSA-N icosanoic acid Chemical compound CCCCCCCCCCCCCCCCCCCC(O)=O VKOBVWXKNCXXDE-UHFFFAOYSA-N 0.000 description 2
- 230000036039 immunity Effects 0.000 description 2
- 230000003308 immunostimulating effect Effects 0.000 description 2
- 238000000338 in vitro Methods 0.000 description 2
- 235000021129 infant diet Nutrition 0.000 description 2
- 229960000367 inositol Drugs 0.000 description 2
- 239000013067 intermediate product Substances 0.000 description 2
- 238000002955 isolation Methods 0.000 description 2
- 235000014666 liquid concentrate Nutrition 0.000 description 2
- XIXADJRWDQXREU-UHFFFAOYSA-M lithium acetate Chemical compound [Li+].CC([O-])=O XIXADJRWDQXREU-UHFFFAOYSA-M 0.000 description 2
- LTYOQGRJFJAKNA-VFLPNFFSSA-N malonyl-coa Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)C(O)C(=O)NCCC(=O)NCCSC(=O)CC(O)=O)O[C@H]1N1C2=NC=NC(N)=C2N=C1 LTYOQGRJFJAKNA-VFLPNFFSSA-N 0.000 description 2
- 238000005259 measurement Methods 0.000 description 2
- BDAGIHXWWSANSR-UHFFFAOYSA-N methanoic acid Natural products OC=O BDAGIHXWWSANSR-UHFFFAOYSA-N 0.000 description 2
- WSFSSNUMVMOOMR-NJFSPNSNSA-N methanone Chemical compound O=[14CH2] WSFSSNUMVMOOMR-NJFSPNSNSA-N 0.000 description 2
- VLKZOEOYAKHREP-UHFFFAOYSA-N n-Hexane Chemical class CCCCCC VLKZOEOYAKHREP-UHFFFAOYSA-N 0.000 description 2
- UTOPWMOLSKOLTQ-UHFFFAOYSA-N octacosanoic acid Chemical compound CCCCCCCCCCCCCCCCCCCCCCCCCCCC(O)=O UTOPWMOLSKOLTQ-UHFFFAOYSA-N 0.000 description 2
- 229920001542 oligosaccharide Polymers 0.000 description 2
- 150000002482 oligosaccharides Chemical class 0.000 description 2
- 235000015927 pasta Nutrition 0.000 description 2
- 230000037361 pathway Effects 0.000 description 2
- 125000002467 phosphate group Chemical group [H]OP(=O)(O[H])O[*] 0.000 description 2
- 229910052698 phosphorus Inorganic materials 0.000 description 2
- 239000011574 phosphorus Substances 0.000 description 2
- 230000000704 physical effect Effects 0.000 description 2
- 229920001282 polysaccharide Polymers 0.000 description 2
- 239000005017 polysaccharide Substances 0.000 description 2
- 239000001103 potassium chloride Substances 0.000 description 2
- 235000011164 potassium chloride Nutrition 0.000 description 2
- 230000002265 prevention Effects 0.000 description 2
- 150000003180 prostaglandins Chemical class 0.000 description 2
- 238000011002 quantification Methods 0.000 description 2
- 230000010076 replication Effects 0.000 description 2
- 230000003362 replicative effect Effects 0.000 description 2
- 238000012552 review Methods 0.000 description 2
- 235000013580 sausages Nutrition 0.000 description 2
- 238000012216 screening Methods 0.000 description 2
- AWUCVROLDVIAJX-GSVOUGTGSA-N sn-glycerol 3-phosphate Chemical compound OC[C@@H](O)COP(O)(O)=O AWUCVROLDVIAJX-GSVOUGTGSA-N 0.000 description 2
- 238000010561 standard procedure Methods 0.000 description 2
- 235000019698 starch Nutrition 0.000 description 2
- 239000008107 starch Substances 0.000 description 2
- 210000000130 stem cell Anatomy 0.000 description 2
- 229910052717 sulfur Inorganic materials 0.000 description 2
- 239000011593 sulfur Substances 0.000 description 2
- 150000007970 thio esters Chemical class 0.000 description 2
- 210000001519 tissue Anatomy 0.000 description 2
- 230000005026 transcription initiation Effects 0.000 description 2
- 239000001226 triphosphate Substances 0.000 description 2
- 235000011178 triphosphate Nutrition 0.000 description 2
- DRTQHJPVMGBUCF-UHFFFAOYSA-N uracil arabinoside Natural products OC1C(O)C(CO)OC1N1C(=O)NC(=O)C=C1 DRTQHJPVMGBUCF-UHFFFAOYSA-N 0.000 description 2
- 229940045145 uridine Drugs 0.000 description 2
- 235000013311 vegetables Nutrition 0.000 description 2
- 239000012138 yeast extract Substances 0.000 description 2
- DGVVWUTYPXICAM-UHFFFAOYSA-N β‐Mercaptoethanol Chemical compound OCCS DGVVWUTYPXICAM-UHFFFAOYSA-N 0.000 description 2
- HDTRYLNUVZCQOY-UHFFFAOYSA-N α-D-glucopyranosyl-α-D-glucopyranoside Natural products OC1C(O)C(O)C(CO)OC1OC1C(O)C(O)C(O)C(CO)O1 HDTRYLNUVZCQOY-UHFFFAOYSA-N 0.000 description 1
- GWHCXVQVJPWHRF-KTKRTIGZSA-N (15Z)-tetracosenoic acid Chemical compound CCCCCCCC\C=C/CCCCCCCCCCCCCC(O)=O GWHCXVQVJPWHRF-KTKRTIGZSA-N 0.000 description 1
- DIGQNXIGRZPYDK-WKSCXVIASA-N (2R)-6-amino-2-[[2-[[(2S)-2-[[2-[[(2R)-2-[[(2S)-2-[[(2R,3S)-2-[[2-[[(2S)-2-[[2-[[(2S)-2-[[(2S)-2-[[(2R)-2-[[(2S,3S)-2-[[(2R)-2-[[(2S)-2-[[(2S)-2-[[(2S)-2-[[2-[[(2S)-2-[[(2R)-2-[[2-[[2-[[2-[(2-amino-1-hydroxyethylidene)amino]-3-carboxy-1-hydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxypropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1,5-dihydroxy-5-iminopentylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxybutylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1,3-dihydroxypropylidene]amino]-1-hydroxyethylidene]amino]-1-hydroxy-3-sulfanylpropylidene]amino]-1-hydroxyethylidene]amino]hexanoic acid Chemical compound C[C@@H]([C@@H](C(=N[C@@H](CS)C(=N[C@@H](C)C(=N[C@@H](CO)C(=NCC(=N[C@@H](CCC(=N)O)C(=NC(CS)C(=N[C@H]([C@H](C)O)C(=N[C@H](CS)C(=N[C@H](CO)C(=NCC(=N[C@H](CS)C(=NCC(=N[C@H](CCCCN)C(=O)O)O)O)O)O)O)O)O)O)O)O)O)O)O)N=C([C@H](CS)N=C([C@H](CO)N=C([C@H](CO)N=C([C@H](C)N=C(CN=C([C@H](CO)N=C([C@H](CS)N=C(CN=C(C(CS)N=C(C(CC(=O)O)N=C(CN)O)O)O)O)O)O)O)O)O)O)O)O DIGQNXIGRZPYDK-WKSCXVIASA-N 0.000 description 1
- CUXYLFPMQMFGPL-UHFFFAOYSA-N (9Z,11E,13E)-9,11,13-Octadecatrienoic acid Natural products CCCCC=CC=CC=CCCCCCCCC(O)=O CUXYLFPMQMFGPL-UHFFFAOYSA-N 0.000 description 1
- LXJXRIRHZLFYRP-VKHMYHEASA-L (R)-2-Hydroxy-3-(phosphonooxy)-propanal Natural products O=C[C@H](O)COP([O-])([O-])=O LXJXRIRHZLFYRP-VKHMYHEASA-L 0.000 description 1
- 102000040650 (ribonucleotides)n+m Human genes 0.000 description 1
- RSWGJHLUYNHPMX-UHFFFAOYSA-N 1,4a-dimethyl-7-propan-2-yl-2,3,4,4b,5,6,10,10a-octahydrophenanthrene-1-carboxylic acid Chemical compound C12CCC(C(C)C)=CC2=CCC2C1(C)CCCC2(C)C(O)=O RSWGJHLUYNHPMX-UHFFFAOYSA-N 0.000 description 1
- 102000018659 1-Acylglycerophosphocholine O-Acyltransferase Human genes 0.000 description 1
- 108010069159 1-acylglycerol-3-phosphate O-acyltransferase Proteins 0.000 description 1
- 102100031251 1-acylglycerol-3-phosphate O-acyltransferase PNPLA3 Human genes 0.000 description 1
- GZCWLCBFPRFLKL-UHFFFAOYSA-N 1-prop-2-ynoxypropan-2-ol Chemical compound CC(O)COCC#C GZCWLCBFPRFLKL-UHFFFAOYSA-N 0.000 description 1
- 108020004465 16S ribosomal RNA Proteins 0.000 description 1
- 101710103125 2,3-bisphosphoglycerate-dependent phosphoglycerate mutase 1 Proteins 0.000 description 1
- 101710162666 2,3-bisphosphoglycerate-independent phosphoglycerate mutase 1 Proteins 0.000 description 1
- QKNYBSVHEMOAJP-UHFFFAOYSA-N 2-amino-2-(hydroxymethyl)propane-1,3-diol;hydron;chloride Chemical compound Cl.OCC(N)(CO)CO QKNYBSVHEMOAJP-UHFFFAOYSA-N 0.000 description 1
- 101710161460 3-oxoacyl-[acyl-carrier-protein] synthase Proteins 0.000 description 1
- 101150033839 4 gene Proteins 0.000 description 1
- OSWFIVFLDKOXQC-UHFFFAOYSA-N 4-(3-methoxyphenyl)aniline Chemical compound COC1=CC=CC(C=2C=CC(N)=CC=2)=C1 OSWFIVFLDKOXQC-UHFFFAOYSA-N 0.000 description 1
- 102100024088 40S ribosomal protein S7 Human genes 0.000 description 1
- UHPMCKVQTMMPCG-UHFFFAOYSA-N 5,8-dihydroxy-2-methoxy-6-methyl-7-(2-oxopropyl)naphthalene-1,4-dione Chemical compound CC1=C(CC(C)=O)C(O)=C2C(=O)C(OC)=CC(=O)C2=C1O UHPMCKVQTMMPCG-UHFFFAOYSA-N 0.000 description 1
- WGPCZPLRVAWXPW-NSHDSACASA-N 5-octyloxolan-2-one Chemical compound CCCCCCCC[C@H]1CCC(=O)O1 WGPCZPLRVAWXPW-NSHDSACASA-N 0.000 description 1
- JXCKZXHCJOVIAV-UHFFFAOYSA-N 6-[(5-bromo-4-chloro-1h-indol-3-yl)oxy]-3,4,5-trihydroxyoxane-2-carboxylic acid;cyclohexanamine Chemical compound [NH3+]C1CCCCC1.O1C(C([O-])=O)C(O)C(O)C(O)C1OC1=CNC2=CC=C(Br)C(Cl)=C12 JXCKZXHCJOVIAV-UHFFFAOYSA-N 0.000 description 1
- FHVDTGUDJYJELY-UHFFFAOYSA-N 6-{[2-carboxy-4,5-dihydroxy-6-(phosphanyloxy)oxan-3-yl]oxy}-4,5-dihydroxy-3-phosphanyloxane-2-carboxylic acid Chemical compound O1C(C(O)=O)C(P)C(O)C(O)C1OC1C(C(O)=O)OC(OP)C(O)C1O FHVDTGUDJYJELY-UHFFFAOYSA-N 0.000 description 1
- 101150093595 ACO1 gene Proteins 0.000 description 1
- 244000215068 Acacia senegal Species 0.000 description 1
- 235000006491 Acacia senegal Nutrition 0.000 description 1
- 108010051457 Acid Phosphatase Proteins 0.000 description 1
- 102000013563 Acid Phosphatase Human genes 0.000 description 1
- 102000004539 Acyl-CoA Oxidase Human genes 0.000 description 1
- 108020001558 Acyl-CoA oxidase Proteins 0.000 description 1
- 229920000936 Agarose Polymers 0.000 description 1
- 102000007698 Alcohol dehydrogenase Human genes 0.000 description 1
- 108010021809 Alcohol dehydrogenase Proteins 0.000 description 1
- GUBGYTABKSRVRQ-XLOQQCSPSA-N Alpha-Lactose Chemical compound O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@H]1O[C@@H]1[C@@H](CO)O[C@H](O)[C@H](O)[C@H]1O GUBGYTABKSRVRQ-XLOQQCSPSA-N 0.000 description 1
- 241000143060 Americamysis bahia Species 0.000 description 1
- 108091093088 Amplicon Proteins 0.000 description 1
- 241000272525 Anas platyrhynchos Species 0.000 description 1
- 241000024188 Andala Species 0.000 description 1
- 102100034613 Annexin A2 Human genes 0.000 description 1
- 244000030795 Annona lutescens Species 0.000 description 1
- 235000005288 Annona lutescens Nutrition 0.000 description 1
- 241000272814 Anser sp. Species 0.000 description 1
- 101100499137 Arabidopsis thaliana DGAT1 gene Proteins 0.000 description 1
- 241000228212 Aspergillus Species 0.000 description 1
- 241000228197 Aspergillus flavus Species 0.000 description 1
- 244000075850 Avena orientalis Species 0.000 description 1
- 241000271566 Aves Species 0.000 description 1
- 101100434663 Bacillus subtilis (strain 168) fbaA gene Proteins 0.000 description 1
- 108010023063 Bacto-peptone Proteins 0.000 description 1
- 241000219310 Beta vulgaris subsp. vulgaris Species 0.000 description 1
- 239000002028 Biomass Substances 0.000 description 1
- 241000209202 Bromus secalinus Species 0.000 description 1
- 239000004322 Butylated hydroxytoluene Substances 0.000 description 1
- NLZUEZXRPGMBCV-UHFFFAOYSA-N Butylhydroxytoluene Chemical compound CC1=CC(C(C)(C)C)=C(O)C(C(C)(C)C)=C1 NLZUEZXRPGMBCV-UHFFFAOYSA-N 0.000 description 1
- 101150098791 CPT1 gene Proteins 0.000 description 1
- OYPRJOBELJOOCE-UHFFFAOYSA-N Calcium Chemical compound [Ca] OYPRJOBELJOOCE-UHFFFAOYSA-N 0.000 description 1
- 241000222178 Candida tropicalis Species 0.000 description 1
- 101100161762 Candida tropicalis POX5 gene Proteins 0.000 description 1
- 239000005632 Capric acid (CAS 334-48-5) Substances 0.000 description 1
- 108090000489 Carboxy-Lyases Proteins 0.000 description 1
- 241000238366 Cephalopoda Species 0.000 description 1
- 240000008886 Ceratonia siliqua Species 0.000 description 1
- 235000013912 Ceratonia siliqua Nutrition 0.000 description 1
- 240000006122 Chenopodium album Species 0.000 description 1
- 235000009344 Chenopodium album Nutrition 0.000 description 1
- 244000033714 Clidemia hirta Species 0.000 description 1
- 235000014277 Clidemia hirta Nutrition 0.000 description 1
- RGJOEKWQDUBAIZ-IBOSZNHHSA-N CoASH Chemical compound O[C@@H]1[C@H](OP(O)(O)=O)[C@@H](COP(O)(=O)OP(O)(=O)OCC(C)(C)[C@@H](O)C(=O)NCCC(=O)NCCS)O[C@H]1N1C2=NC=NC(N)=C2N=C1 RGJOEKWQDUBAIZ-IBOSZNHHSA-N 0.000 description 1
- 241001480517 Conidiobolus Species 0.000 description 1
- 241000938605 Crocodylia Species 0.000 description 1
- 241000235646 Cyberlindnera jadinii Species 0.000 description 1
- 241000252233 Cyprinus carpio Species 0.000 description 1
- LXJXRIRHZLFYRP-VKHMYHEASA-N D-glyceraldehyde 3-phosphate Chemical compound O=C[C@H](O)COP(O)(O)=O LXJXRIRHZLFYRP-VKHMYHEASA-N 0.000 description 1
- 101150023395 DGA1 gene Proteins 0.000 description 1
- 108090000626 DNA-directed RNA polymerases Proteins 0.000 description 1
- 102000004163 DNA-directed RNA polymerases Human genes 0.000 description 1
- 241000235035 Debaryomyces Species 0.000 description 1
- 101000729758 Dehalococcoides mccartyi (strain ATCC BAA-2266 / KCTC 15142 / 195) Glycerol-3-phosphate acyltransferase 5 Proteins 0.000 description 1
- 102000007260 Deoxyribonuclease I Human genes 0.000 description 1
- 108010008532 Deoxyribonuclease I Proteins 0.000 description 1
- 229940123980 Desaturase inhibitor Drugs 0.000 description 1
- 229920002307 Dextran Polymers 0.000 description 1
- 241000723298 Dicentrarchus labrax Species 0.000 description 1
- 101000925662 Enterobacteria phage PRD1 Endolysin Proteins 0.000 description 1
- 239000004386 Erythritol Substances 0.000 description 1
- UNXHWFMMPAWVPI-UHFFFAOYSA-N Erythritol Natural products OCC(O)C(O)CO UNXHWFMMPAWVPI-UHFFFAOYSA-N 0.000 description 1
- 108090000371 Esterases Proteins 0.000 description 1
- KGWDUNBJIMUFAP-KVVVOXFISA-N Ethanolamine Oleate Chemical compound NCCO.CCCCCCCC\C=C/CCCCCCCC(O)=O KGWDUNBJIMUFAP-KVVVOXFISA-N 0.000 description 1
- 239000004258 Ethoxyquin Substances 0.000 description 1
- 241000195620 Euglena Species 0.000 description 1
- 241000206602 Eukaryota Species 0.000 description 1
- 108091029865 Exogenous DNA Proteins 0.000 description 1
- 101710089384 Extracellular protease Proteins 0.000 description 1
- 241000282326 Felis catus Species 0.000 description 1
- 235000019733 Fish meal Nutrition 0.000 description 1
- BDAGIHXWWSANSR-UHFFFAOYSA-M Formate Chemical compound [O-]C=O BDAGIHXWWSANSR-UHFFFAOYSA-M 0.000 description 1
- 229930091371 Fructose Natural products 0.000 description 1
- 239000005715 Fructose Substances 0.000 description 1
- RFSUNEUAIZKAJO-ARQDHWQXSA-N Fructose Chemical compound OC[C@H]1O[C@](O)(CO)[C@@H](O)[C@@H]1O RFSUNEUAIZKAJO-ARQDHWQXSA-N 0.000 description 1
- 241000223218 Fusarium Species 0.000 description 1
- IAJILQKETJEXLJ-UHFFFAOYSA-N Galacturonsaeure Natural products O=CC(O)C(O)C(O)C(O)C(O)=O IAJILQKETJEXLJ-UHFFFAOYSA-N 0.000 description 1
- 108700007698 Genetic Terminator Regions Proteins 0.000 description 1
- 108010073178 Glucan 1,4-alpha-Glucosidase Proteins 0.000 description 1
- 108010033128 Glucan Endo-1,3-beta-D-Glucosidase Proteins 0.000 description 1
- 102100022624 Glucoamylase Human genes 0.000 description 1
- 102000005731 Glucose-6-phosphate isomerase Human genes 0.000 description 1
- 108010070600 Glucose-6-phosphate isomerase Proteins 0.000 description 1
- 102100024017 Glycerol-3-phosphate acyltransferase 3 Human genes 0.000 description 1
- 101710199764 Glycerol-3-phosphate acyltransferase 3 Proteins 0.000 description 1
- 102100040870 Glycine amidinotransferase, mitochondrial Human genes 0.000 description 1
- 229930186217 Glycolipid Natural products 0.000 description 1
- 101100246753 Halobacterium salinarum (strain ATCC 700922 / JCM 11081 / NRC-1) pyrF gene Proteins 0.000 description 1
- SQUHHTBVTRBESD-UHFFFAOYSA-N Hexa-Ac-myo-Inositol Natural products CC(=O)OC1C(OC(C)=O)C(OC(C)=O)C(OC(C)=O)C(OC(C)=O)C1OC(C)=O SQUHHTBVTRBESD-UHFFFAOYSA-N 0.000 description 1
- 101000924474 Homo sapiens Annexin A2 Proteins 0.000 description 1
- 101000911390 Homo sapiens Coagulation factor VIII Proteins 0.000 description 1
- 101000893303 Homo sapiens Glycine amidinotransferase, mitochondrial Proteins 0.000 description 1
- 101100084403 Homo sapiens PRODH gene Proteins 0.000 description 1
- 101000955959 Homo sapiens Vacuolar protein sorting-associated protein 52 homolog Proteins 0.000 description 1
- 240000005979 Hordeum vulgare Species 0.000 description 1
- 235000007340 Hordeum vulgare Nutrition 0.000 description 1
- UFHFLCQGNIYNRP-UHFFFAOYSA-N Hydrogen Chemical compound [H][H] UFHFLCQGNIYNRP-UHFFFAOYSA-N 0.000 description 1
- 102000008070 Interferon-gamma Human genes 0.000 description 1
- 108010074328 Interferon-gamma Proteins 0.000 description 1
- 102000014150 Interferons Human genes 0.000 description 1
- 108010050904 Interferons Proteins 0.000 description 1
- 108020003285 Isocitrate lyase Proteins 0.000 description 1
- 241000235649 Kluyveromyces Species 0.000 description 1
- QNAYBMKLOCPYGJ-REOHCLBHSA-N L-alanine Chemical compound C[C@H](N)C(O)=O QNAYBMKLOCPYGJ-REOHCLBHSA-N 0.000 description 1
- HNDVDQJCIGZPNO-YFKPBYRVSA-N L-histidine Chemical compound OC(=O)[C@@H](N)CC1=CN=CN1 HNDVDQJCIGZPNO-YFKPBYRVSA-N 0.000 description 1
- QIVBCDIJIAJPQS-VIFPVBQESA-N L-tryptophane Chemical compound C1=CC=C2C(C[C@H](N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-VIFPVBQESA-N 0.000 description 1
- 101150007280 LEU2 gene Proteins 0.000 description 1
- GUBGYTABKSRVRQ-QKKXKWKRSA-N Lactose Natural products OC[C@H]1O[C@@H](O[C@H]2[C@H](O)[C@@H](O)C(O)O[C@@H]2CO)[C@H](O)[C@@H](O)[C@H]1O GUBGYTABKSRVRQ-QKKXKWKRSA-N 0.000 description 1
- 241001596950 Larimichthys crocea Species 0.000 description 1
- 239000005639 Lauric acid Substances 0.000 description 1
- 108090000364 Ligases Proteins 0.000 description 1
- 102000003960 Ligases Human genes 0.000 description 1
- 235000021353 Lignoceric acid Nutrition 0.000 description 1
- CQXMAMUUWHYSIY-UHFFFAOYSA-N Lignoceric acid Natural products CCCCCCCCCCCCCCCCCCCCCCCC(=O)OCCC1=CC=C(O)C=C1 CQXMAMUUWHYSIY-UHFFFAOYSA-N 0.000 description 1
- 239000006142 Luria-Bertani Agar Substances 0.000 description 1
- 101710163717 Lysophospholipid acyltransferase 5 Proteins 0.000 description 1
- 208000002720 Malnutrition Diseases 0.000 description 1
- 241000124008 Mammalia Species 0.000 description 1
- 240000004658 Medicago sativa Species 0.000 description 1
- 235000017587 Medicago sativa ssp. sativa Nutrition 0.000 description 1
- 102000003792 Metallothionein Human genes 0.000 description 1
- 108090000157 Metallothionein Proteins 0.000 description 1
- 241001123676 Metschnikowia pulcherrima Species 0.000 description 1
- 108700005443 Microbial Genes Proteins 0.000 description 1
- 241000498617 Mucor javanicus Species 0.000 description 1
- ZMXDDKWLCZADIW-UHFFFAOYSA-N N,N-Dimethylformamide Chemical class CN(C)C=O ZMXDDKWLCZADIW-UHFFFAOYSA-N 0.000 description 1
- 241001460678 Napo <wasp> Species 0.000 description 1
- 241000244206 Nematoda Species 0.000 description 1
- 206010028980 Neoplasm Diseases 0.000 description 1
- 241000272458 Numididae Species 0.000 description 1
- 108020005187 Oligonucleotide Probes Proteins 0.000 description 1
- 241000283973 Oryctolagus cuniculus Species 0.000 description 1
- 240000007594 Oryza sativa Species 0.000 description 1
- 235000007164 Oryza sativa Nutrition 0.000 description 1
- 101100008883 Oryza sativa subsp. japonica DGAT1-1 gene Proteins 0.000 description 1
- 229910019142 PO4 Inorganic materials 0.000 description 1
- 101150105372 POX1 gene Proteins 0.000 description 1
- 101150114097 POX3 gene Proteins 0.000 description 1
- 101150053659 POX4 gene Proteins 0.000 description 1
- 101150004239 POX5 gene Proteins 0.000 description 1
- 208000030852 Parasitic disease Diseases 0.000 description 1
- 241001494479 Pecora Species 0.000 description 1
- 241000228143 Penicillium Species 0.000 description 1
- 235000010627 Phaseolus vulgaris Nutrition 0.000 description 1
- 244000046052 Phaseolus vulgaris Species 0.000 description 1
- 108030002254 Phosphate acyltransferases Proteins 0.000 description 1
- 108010069394 Phosphatidate Phosphatase Proteins 0.000 description 1
- 102000001107 Phosphatidate Phosphatase Human genes 0.000 description 1
- 108010011964 Phosphatidylcholine-sterol O-acyltransferase Proteins 0.000 description 1
- 102100035200 Phospholipase A and acyltransferase 4 Human genes 0.000 description 1
- 102100032967 Phospholipase D1 Human genes 0.000 description 1
- 108030002650 Phospholipid:diacylglycerol acyltransferases Proteins 0.000 description 1
- 108091000080 Phosphotransferase Proteins 0.000 description 1
- 241000233614 Phytophthora Species 0.000 description 1
- DLRVVLDZNNYCBX-UHFFFAOYSA-N Polydextrose Polymers OC1C(O)C(O)C(CO)OC1OCC1C(O)C(O)C(O)C(O)O1 DLRVVLDZNNYCBX-UHFFFAOYSA-N 0.000 description 1
- 241000206618 Porphyridium Species 0.000 description 1
- 102100028772 Proline dehydrogenase 1, mitochondrial Human genes 0.000 description 1
- 102000001253 Protein Kinase Human genes 0.000 description 1
- 229940096437 Protein S Drugs 0.000 description 1
- 108010066124 Protein S Proteins 0.000 description 1
- 108010076504 Protein Sorting Signals Proteins 0.000 description 1
- CZPWVGJYEJSRLH-UHFFFAOYSA-N Pyrimidine Chemical compound C1=CN=CN=C1 CZPWVGJYEJSRLH-UHFFFAOYSA-N 0.000 description 1
- 108091034057 RNA (poly(A)) Proteins 0.000 description 1
- 101000813109 Rattus norvegicus Elongation of very long chain fatty acids protein 6 Proteins 0.000 description 1
- 241000206572 Rhodophyta Species 0.000 description 1
- 241000283984 Rodentia Species 0.000 description 1
- 241000700141 Rotifera Species 0.000 description 1
- 101100501251 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) ELO3 gene Proteins 0.000 description 1
- 101100351254 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) LRO1 gene Proteins 0.000 description 1
- 101100313649 Saccharomyces cerevisiae (strain ATCC 204508 / S288c) POT1 gene Proteins 0.000 description 1
- 241000277331 Salmonidae Species 0.000 description 1
- 241000235346 Schizosaccharomyces Species 0.000 description 1
- 235000007238 Secale cereale Nutrition 0.000 description 1
- 244000082988 Secale cereale Species 0.000 description 1
- 244000062793 Sorghum vulgare Species 0.000 description 1
- 244000046109 Sorghum vulgare var. nervosum Species 0.000 description 1
- 235000021536 Sugar beet Nutrition 0.000 description 1
- 241000282887 Suidae Species 0.000 description 1
- 241001634922 Tausonia pullulans Species 0.000 description 1
- 108060008225 Thiolase Proteins 0.000 description 1
- 102000002932 Thiolase Human genes 0.000 description 1
- 241000276707 Tilapia Species 0.000 description 1
- 102000004357 Transferases Human genes 0.000 description 1
- 108090000992 Transferases Proteins 0.000 description 1
- 108010020764 Transposases Proteins 0.000 description 1
- 102000008579 Transposases Human genes 0.000 description 1
- HDTRYLNUVZCQOY-WSWWMNSNSA-N Trehalose Natural products O[C@@H]1[C@@H](O)[C@@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@H](O)[C@@H](O)[C@@H](O)[C@@H](CO)O1 HDTRYLNUVZCQOY-WSWWMNSNSA-N 0.000 description 1
- 241000005601 Trisetum Species 0.000 description 1
- 235000019714 Triticale Nutrition 0.000 description 1
- 235000021307 Triticum Nutrition 0.000 description 1
- 244000098338 Triticum aestivum Species 0.000 description 1
- 229920004890 Triton X-100 Polymers 0.000 description 1
- 239000013504 Triton X-100 Substances 0.000 description 1
- QIVBCDIJIAJPQS-UHFFFAOYSA-N Tryptophan Natural products C1=CC=C2C(CC(N)C(O)=O)=CNC2=C1 QIVBCDIJIAJPQS-UHFFFAOYSA-N 0.000 description 1
- 239000006035 Tryptophane Substances 0.000 description 1
- 208000034953 Twin anemia-polycythemia sequence Diseases 0.000 description 1
- 241000134363 Umbelopsis ramanniana Species 0.000 description 1
- 102100038937 Vacuolar protein sorting-associated protein 52 homolog Human genes 0.000 description 1
- 229930003427 Vitamin E Natural products 0.000 description 1
- 102100028885 Vitamin K-dependent protein S Human genes 0.000 description 1
- 239000005862 Whey Substances 0.000 description 1
- 102000007544 Whey Proteins Human genes 0.000 description 1
- 108010046377 Whey Proteins Proteins 0.000 description 1
- 241001532014 Xanthorrhoea Species 0.000 description 1
- 101100029251 Zea mays PER2 gene Proteins 0.000 description 1
- 241000746966 Zizania Species 0.000 description 1
- 235000002636 Zizania aquatica Nutrition 0.000 description 1
- 230000009102 absorption Effects 0.000 description 1
- 238000010521 absorption reaction Methods 0.000 description 1
- 150000007513 acids Chemical class 0.000 description 1
- 239000000654 additive Substances 0.000 description 1
- 230000000996 additive effect Effects 0.000 description 1
- 238000004026 adhesive bonding Methods 0.000 description 1
- 230000000240 adjuvant effect Effects 0.000 description 1
- 230000002411 adverse Effects 0.000 description 1
- 229940072056 alginate Drugs 0.000 description 1
- 235000010443 alginic acid Nutrition 0.000 description 1
- 229920000615 alginic acid Polymers 0.000 description 1
- 101150099105 alien gene Proteins 0.000 description 1
- 125000001931 aliphatic group Chemical group 0.000 description 1
- AHANXAKGNAKFSK-PDBXOOCHSA-N all-cis-icosa-11,14,17-trienoic acid Chemical compound CC\C=C/C\C=C/C\C=C/CCCCCCCCCC(O)=O AHANXAKGNAKFSK-PDBXOOCHSA-N 0.000 description 1
- CUXYLFPMQMFGPL-SUTYWZMXSA-N all-trans-octadeca-9,11,13-trienoic acid Chemical compound CCCC\C=C\C=C\C=C\CCCCCCCC(O)=O CUXYLFPMQMFGPL-SUTYWZMXSA-N 0.000 description 1
- 230000000735 allogeneic effect Effects 0.000 description 1
- HDTRYLNUVZCQOY-LIZSDCNHSA-N alpha,alpha-trehalose Chemical compound O[C@@H]1[C@@H](O)[C@H](O)[C@@H](CO)O[C@@H]1O[C@@H]1[C@H](O)[C@@H](O)[C@H](O)[C@@H](CO)O1 HDTRYLNUVZCQOY-LIZSDCNHSA-N 0.000 description 1
- 125000000266 alpha-aminoacyl group Chemical group 0.000 description 1
- 125000000539 amino acid group Chemical group 0.000 description 1
- 229940126575 aminoglycoside Drugs 0.000 description 1
- 150000003863 ammonium salts Chemical class 0.000 description 1
- AVKUERGKIZMTKX-NJBDSQKTSA-N ampicillin Chemical compound C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C(O)=O)(C)C)=CC=CC=C1 AVKUERGKIZMTKX-NJBDSQKTSA-N 0.000 description 1
- 229960000723 ampicillin Drugs 0.000 description 1
- KLOHDWPABZXLGI-YWUHCJSESA-M ampicillin sodium Chemical compound [Na+].C1([C@@H](N)C(=O)N[C@H]2[C@H]3SC([C@@H](N3C2=O)C([O-])=O)(C)C)=CC=CC=C1 KLOHDWPABZXLGI-YWUHCJSESA-M 0.000 description 1
- 229940121363 anti-inflammatory agent Drugs 0.000 description 1
- 239000002260 anti-inflammatory agent Substances 0.000 description 1
- 230000000840 anti-viral effect Effects 0.000 description 1
- 239000003529 anticholesteremic agent Substances 0.000 description 1
- 229940127226 anticholesterol agent Drugs 0.000 description 1
- 239000000427 antigen Substances 0.000 description 1
- 108091007433 antigens Proteins 0.000 description 1
- 102000036639 antigens Human genes 0.000 description 1
- 239000003963 antioxidant agent Substances 0.000 description 1
- 230000003078 antioxidant effect Effects 0.000 description 1
- 235000006708 antioxidants Nutrition 0.000 description 1
- 239000007864 aqueous solution Substances 0.000 description 1
- 238000003556 assay Methods 0.000 description 1
- 125000004429 atom Chemical group 0.000 description 1
- 235000015241 bacon Nutrition 0.000 description 1
- 235000015173 baked goods and baking mixes Nutrition 0.000 description 1
- 235000013527 bean curd Nutrition 0.000 description 1
- WPYMKLBDIGXBTP-UHFFFAOYSA-N benzoic acid Chemical compound OC(=O)C1=CC=CC=C1 WPYMKLBDIGXBTP-UHFFFAOYSA-N 0.000 description 1
- WQZGKKKJIJFFOK-VFUOTHLCSA-N beta-D-glucose Chemical compound OC[C@H]1O[C@@H](O)[C@H](O)[C@@H](O)[C@@H]1O WQZGKKKJIJFFOK-VFUOTHLCSA-N 0.000 description 1
- AEMOLEFTQBMNLQ-QIUUJYRFSA-N beta-D-glucuronic acid Chemical compound O[C@@H]1O[C@H](C(O)=O)[C@@H](O)[C@H](O)[C@H]1O AEMOLEFTQBMNLQ-QIUUJYRFSA-N 0.000 description 1
- 238000004166 bioassay Methods 0.000 description 1
- 230000003115 biocidal effect Effects 0.000 description 1
- 239000003139 biocide Substances 0.000 description 1
- 235000015895 biscuits Nutrition 0.000 description 1
- 239000012152 bradford reagent Substances 0.000 description 1
- 210000004556 brain Anatomy 0.000 description 1
- 150000005693 branched-chain amino acids Chemical class 0.000 description 1
- 235000015496 breakfast cereal Nutrition 0.000 description 1
- 238000009395 breeding Methods 0.000 description 1
- 230000001488 breeding effect Effects 0.000 description 1
- 239000000872 buffer Substances 0.000 description 1
- 235000010354 butylated hydroxytoluene Nutrition 0.000 description 1
- 229940095259 butylated hydroxytoluene Drugs 0.000 description 1
- 239000011575 calcium Substances 0.000 description 1
- 229910052791 calcium Inorganic materials 0.000 description 1
- 239000004202 carbamide Substances 0.000 description 1
- 235000021466 carotenoid Nutrition 0.000 description 1
- 150000001747 carotenoids Chemical class 0.000 description 1
- 239000000969 carrier Substances 0.000 description 1
- 230000006652 catabolic pathway Effects 0.000 description 1
- 239000003054 catalyst Substances 0.000 description 1
- 230000003197 catalytic effect Effects 0.000 description 1
- 238000009903 catalytic hydrogenation reaction Methods 0.000 description 1
- 241001233037 catfish Species 0.000 description 1
- 238000004113 cell culture Methods 0.000 description 1
- 239000008004 cell lysis buffer Substances 0.000 description 1
- 239000006285 cell suspension Substances 0.000 description 1
- 230000001413 cellular effect Effects 0.000 description 1
- 238000012512 characterization method Methods 0.000 description 1
- 239000002975 chemoattractant Substances 0.000 description 1
- 235000015218 chewing gum Nutrition 0.000 description 1
- 229940112822 chewing gum Drugs 0.000 description 1
- 210000000349 chromosome Anatomy 0.000 description 1
- 239000013599 cloning vector Substances 0.000 description 1
- RGJOEKWQDUBAIZ-UHFFFAOYSA-N coenzime A Natural products OC1C(OP(O)(O)=O)C(COP(O)(=O)OP(O)(=O)OCC(C)(C)C(O)C(=O)NCCC(=O)NCCS)OC1N1C2=NC=NC(N)=C2N=C1 RGJOEKWQDUBAIZ-UHFFFAOYSA-N 0.000 description 1
- 239000005516 coenzyme A Substances 0.000 description 1
- 229940093530 coenzyme a Drugs 0.000 description 1
- 238000009833 condensation Methods 0.000 description 1
- 230000005494 condensation Effects 0.000 description 1
- 238000006482 condensation reaction Methods 0.000 description 1
- 235000020186 condensed milk Nutrition 0.000 description 1
- 235000013409 condiments Nutrition 0.000 description 1
- 230000003750 conditioning effect Effects 0.000 description 1
- 238000012790 confirmation Methods 0.000 description 1
- 235000014510 cooky Nutrition 0.000 description 1
- 208000029078 coronary artery disease Diseases 0.000 description 1
- 239000002537 cosmetic Substances 0.000 description 1
- 230000008878 coupling Effects 0.000 description 1
- 238000010168 coupling process Methods 0.000 description 1
- 238000005859 coupling reaction Methods 0.000 description 1
- 238000005336 cracking Methods 0.000 description 1
- 235000012777 crisp bread Nutrition 0.000 description 1
- 235000015140 cultured milk Nutrition 0.000 description 1
- 125000004122 cyclic group Chemical group 0.000 description 1
- 229940104302 cytosine Drugs 0.000 description 1
- 238000005034 decoration Methods 0.000 description 1
- 230000007547 defect Effects 0.000 description 1
- 230000007812 deficiency Effects 0.000 description 1
- 230000002950 deficient Effects 0.000 description 1
- 230000018044 dehydration Effects 0.000 description 1
- 238000006297 dehydration reaction Methods 0.000 description 1
- KDTSHFARGAKYJN-UHFFFAOYSA-N dephosphocoenzyme A Natural products OC1C(O)C(COP(O)(=O)OP(O)(=O)OCC(C)(C)C(O)C(=O)NCCC(=O)NCCS)OC1N1C2=NC=NC(N)=C2N=C1 KDTSHFARGAKYJN-UHFFFAOYSA-N 0.000 description 1
- 235000021185 dessert Nutrition 0.000 description 1
- 235000021373 diet component Nutrition 0.000 description 1
- 230000000378 dietary effect Effects 0.000 description 1
- 230000004069 differentiation Effects 0.000 description 1
- 239000003085 diluting agent Substances 0.000 description 1
- 150000002016 disaccharides Chemical class 0.000 description 1
- 239000012153 distilled water Substances 0.000 description 1
- 235000012489 doughnuts Nutrition 0.000 description 1
- 238000011143 downstream manufacturing Methods 0.000 description 1
- 238000001647 drug administration Methods 0.000 description 1
- 238000001035 drying Methods 0.000 description 1
- 239000000975 dye Substances 0.000 description 1
- 230000004064 dysfunction Effects 0.000 description 1
- PRHHYVQTPBEDFE-UHFFFAOYSA-N eicosatrienoic acid Natural products CCCCCC=CCC=CCCCCC=CCCCC(O)=O PRHHYVQTPBEDFE-UHFFFAOYSA-N 0.000 description 1
- 229920001971 elastomer Polymers 0.000 description 1
- 238000004520 electroporation Methods 0.000 description 1
- 101150019586 elo-3 gene Proteins 0.000 description 1
- 239000002158 endotoxin Substances 0.000 description 1
- 238000012407 engineering method Methods 0.000 description 1
- 230000007613 environmental effect Effects 0.000 description 1
- 230000002255 enzymatic effect Effects 0.000 description 1
- 238000001976 enzyme digestion Methods 0.000 description 1
- KAQKFAOMNZTLHT-VVUHWYTRSA-N epoprostenol Chemical compound O1C(=CCCCC(O)=O)C[C@@H]2[C@@H](/C=C/[C@@H](O)CCCCC)[C@H](O)C[C@@H]21 KAQKFAOMNZTLHT-VVUHWYTRSA-N 0.000 description 1
- 229960001123 epoprostenol Drugs 0.000 description 1
- 229940009714 erythritol Drugs 0.000 description 1
- UNXHWFMMPAWVPI-ZXZARUISSA-N erythritol Chemical compound OC[C@H](O)[C@H](O)CO UNXHWFMMPAWVPI-ZXZARUISSA-N 0.000 description 1
- 235000019414 erythritol Nutrition 0.000 description 1
- 235000004626 essential fatty acids Nutrition 0.000 description 1
- BEFDCLMNVWHSGT-UHFFFAOYSA-N ethenylcyclopentane Chemical compound C=CC1CCCC1 BEFDCLMNVWHSGT-UHFFFAOYSA-N 0.000 description 1
- ZMMJGEGLRURXTF-UHFFFAOYSA-N ethidium bromide Chemical compound [Br-].C12=CC(N)=CC=C2C2=CC=C(N)C=C2[N+](CC)=C1C1=CC=CC=C1 ZMMJGEGLRURXTF-UHFFFAOYSA-N 0.000 description 1
- 229960005542 ethidium bromide Drugs 0.000 description 1
- DECIPOUIJURFOJ-UHFFFAOYSA-N ethoxyquin Chemical compound N1C(C)(C)C=C(C)C2=CC(OCC)=CC=C21 DECIPOUIJURFOJ-UHFFFAOYSA-N 0.000 description 1
- 229940093500 ethoxyquin Drugs 0.000 description 1
- 235000019285 ethoxyquin Nutrition 0.000 description 1
- 230000005284 excitation Effects 0.000 description 1
- 101150017244 exp-1 gene Proteins 0.000 description 1
- 239000013604 expression vector Substances 0.000 description 1
- 238000009313 farming Methods 0.000 description 1
- 235000019387 fatty acid methyl ester Nutrition 0.000 description 1
- 150000002190 fatty acyls Chemical group 0.000 description 1
- 125000001924 fatty-acyl group Chemical group 0.000 description 1
- 101150035621 fen1 gene Proteins 0.000 description 1
- 239000000835 fiber Substances 0.000 description 1
- 239000000945 filler Substances 0.000 description 1
- 239000004467 fishmeal Substances 0.000 description 1
- 235000013312 flour Nutrition 0.000 description 1
- 239000007850 fluorescent dye Substances 0.000 description 1
- 235000019253 formic acid Nutrition 0.000 description 1
- 238000007710 freezing Methods 0.000 description 1
- 230000008014 freezing Effects 0.000 description 1
- 235000015203 fruit juice Nutrition 0.000 description 1
- 208000024386 fungal infectious disease Diseases 0.000 description 1
- IFYYFLINQYPWGJ-VIFPVBQESA-N gamma-Decalactone Natural products CCCCCC[C@H]1CCC(=O)O1 IFYYFLINQYPWGJ-VIFPVBQESA-N 0.000 description 1
- WGPCZPLRVAWXPW-LLVKDONJSA-N gamma-Dodecalactone Natural products CCCCCCCC[C@@H]1CCC(=O)O1 WGPCZPLRVAWXPW-LLVKDONJSA-N 0.000 description 1
- 229940044627 gamma-interferon Drugs 0.000 description 1
- WIGCFUFOHFEKBI-UHFFFAOYSA-N gamma-tocopherol Natural products CC(C)CCCC(C)CCCC(C)CCCC1CCC2C(C)C(O)C(C)C(C)C2O1 WIGCFUFOHFEKBI-UHFFFAOYSA-N 0.000 description 1
- 238000004817 gas chromatography Methods 0.000 description 1
- 238000001502 gel electrophoresis Methods 0.000 description 1
- 235000021474 generally recognized As safe (food) Nutrition 0.000 description 1
- 235000021473 generally recognized as safe (food ingredients) Nutrition 0.000 description 1
- 238000010353 genetic engineering Methods 0.000 description 1
- 210000004907 gland Anatomy 0.000 description 1
- 229940097043 glucuronic acid Drugs 0.000 description 1
- 229930182480 glucuronide Natural products 0.000 description 1
- 150000008134 glucuronides Chemical class 0.000 description 1
- 229960002989 glutamic acid Drugs 0.000 description 1
- 125000003976 glyceryl group Chemical group [H]C([*])([H])C(O[H])([H])C(O[H])([H])[H] 0.000 description 1
- 230000034659 glycolysis Effects 0.000 description 1
- 229930182470 glycoside Natural products 0.000 description 1
- 150000002338 glycosides Chemical class 0.000 description 1
- 239000008187 granular material Substances 0.000 description 1
- LHGVFZTZFXWLCP-UHFFFAOYSA-N guaiacol Chemical class COC1=CC=CC=C1O LHGVFZTZFXWLCP-UHFFFAOYSA-N 0.000 description 1
- 101150054900 gus gene Proteins 0.000 description 1
- 238000003306 harvesting Methods 0.000 description 1
- 230000002949 hemolytic effect Effects 0.000 description 1
- 230000002363 herbicidal effect Effects 0.000 description 1
- 239000004009 herbicide Substances 0.000 description 1
- XMHIUKTWLZUKEX-UHFFFAOYSA-N hexacosanoic acid Chemical compound CCCCCCCCCCCCCCCCCCCCCCCCCC(O)=O XMHIUKTWLZUKEX-UHFFFAOYSA-N 0.000 description 1
- KYYWBEYKBLQSFW-UHFFFAOYSA-N hexadecanoic acid Chemical compound CCCCCCCCCCCCCCCC(O)=O.CCCCCCCCCCCCCCCC(O)=O KYYWBEYKBLQSFW-UHFFFAOYSA-N 0.000 description 1
- HNDVDQJCIGZPNO-UHFFFAOYSA-N histidine Natural products OC(=O)C(N)CC1=CN=CN1 HNDVDQJCIGZPNO-UHFFFAOYSA-N 0.000 description 1
- 235000019692 hotdogs Nutrition 0.000 description 1
- 102000057593 human F8 Human genes 0.000 description 1
- 230000036571 hydration Effects 0.000 description 1
- 238000006703 hydration reaction Methods 0.000 description 1
- 239000001257 hydrogen Substances 0.000 description 1
- 229910052739 hydrogen Inorganic materials 0.000 description 1
- 230000003301 hydrolyzing effect Effects 0.000 description 1
- 125000004356 hydroxy functional group Chemical group O* 0.000 description 1
- YQYJSBFKSSDGFO-FWAVGLHBSA-N hygromycin A Chemical compound O[C@H]1[C@H](O)[C@H](C(=O)C)O[C@@H]1Oc1ccc(\C=C(/C)C(=O)N[C@@H]2[C@@H]([C@H]3OCO[C@H]3[C@@H](O)[C@@H]2O)O)cc1O YQYJSBFKSSDGFO-FWAVGLHBSA-N 0.000 description 1
- 230000000774 hypoallergenic effect Effects 0.000 description 1
- 235000015243 ice cream Nutrition 0.000 description 1
- YAMHXTCMCPHKLN-UHFFFAOYSA-N imidazolidin-2-one Chemical compound O=C1NCCN1 YAMHXTCMCPHKLN-UHFFFAOYSA-N 0.000 description 1
- 230000007365 immunoregulation Effects 0.000 description 1
- 229960001438 immunostimulant agent Drugs 0.000 description 1
- 239000003022 immunostimulating agent Substances 0.000 description 1
- 230000001771 impaired effect Effects 0.000 description 1
- 238000007901 in situ hybridization Methods 0.000 description 1
- 238000001727 in vivo Methods 0.000 description 1
- 230000006698 induction Effects 0.000 description 1
- 239000003112 inhibitor Substances 0.000 description 1
- 239000003999 initiator Substances 0.000 description 1
- CDAISMWEOUEBRE-GPIVLXJGSA-N inositol Chemical compound O[C@H]1[C@H](O)[C@@H](O)[C@H](O)[C@H](O)[C@@H]1O CDAISMWEOUEBRE-GPIVLXJGSA-N 0.000 description 1
- 238000003780 insertion Methods 0.000 description 1
- 230000037431 insertion Effects 0.000 description 1
- 238000007689 inspection Methods 0.000 description 1
- 230000003993 interaction Effects 0.000 description 1
- 229940079322 interferon Drugs 0.000 description 1
- 238000007918 intramuscular administration Methods 0.000 description 1
- 235000015094 jam Nutrition 0.000 description 1
- 235000015110 jellies Nutrition 0.000 description 1
- 239000008274 jelly Substances 0.000 description 1
- 230000006651 lactation Effects 0.000 description 1
- 239000008101 lactose Substances 0.000 description 1
- 230000000670 limiting effect Effects 0.000 description 1
- 210000004185 liver Anatomy 0.000 description 1
- 244000144972 livestock Species 0.000 description 1
- 230000007774 longterm Effects 0.000 description 1
- 239000006210 lotion Substances 0.000 description 1
- 239000006166 lysate Substances 0.000 description 1
- 230000002101 lytic effect Effects 0.000 description 1
- 230000001071 malnutrition Effects 0.000 description 1
- 235000000824 malnutrition Nutrition 0.000 description 1
- 230000035800 maturation Effects 0.000 description 1
- 238000012269 metabolic engineering Methods 0.000 description 1
- 239000002207 metabolite Substances 0.000 description 1
- 229910021645 metal ion Inorganic materials 0.000 description 1
- 238000000520 microinjection Methods 0.000 description 1
- 235000019713 millet Nutrition 0.000 description 1
- 235000013379 molasses Nutrition 0.000 description 1
- 210000001616 monocyte Anatomy 0.000 description 1
- 235000021281 monounsaturated fatty acids Nutrition 0.000 description 1
- 230000002969 morbid Effects 0.000 description 1
- 239000003471 mutagenic agent Substances 0.000 description 1
- 230000035772 mutation Effects 0.000 description 1
- 230000001537 neural effect Effects 0.000 description 1
- 210000004493 neutrocyte Anatomy 0.000 description 1
- 229910052759 nickel Inorganic materials 0.000 description 1
- 230000001937 non-anti-biotic effect Effects 0.000 description 1
- 235000021590 normal diet Nutrition 0.000 description 1
- 235000021048 nutrient requirements Nutrition 0.000 description 1
- 208000015380 nutritional deficiency disease Diseases 0.000 description 1
- 235000003715 nutritional status Nutrition 0.000 description 1
- RQFLGKYCYMMRMC-UHFFFAOYSA-N octadecanoic acid Chemical compound CCCCCCCCCCCCCCCCCC(O)=O.CCCCCCCCCCCCCCCCCC(O)=O RQFLGKYCYMMRMC-UHFFFAOYSA-N 0.000 description 1
- 239000002674 ointment Substances 0.000 description 1
- JRZJOMJEPLMPRA-UHFFFAOYSA-N olefin Natural products CCCCCCCC=C JRZJOMJEPLMPRA-UHFFFAOYSA-N 0.000 description 1
- 239000002751 oligonucleotide probe Substances 0.000 description 1
- 239000003960 organic solvent Substances 0.000 description 1
- 230000008520 organization Effects 0.000 description 1
- 210000002220 organoid Anatomy 0.000 description 1
- FKCRAVPPBFWEJD-XVFCMESISA-N orotidine Chemical compound O[C@@H]1[C@H](O)[C@@H](CO)O[C@H]1N1C(=O)NC(=O)C=C1C(O)=O FKCRAVPPBFWEJD-XVFCMESISA-N 0.000 description 1
- FKCRAVPPBFWEJD-UHFFFAOYSA-N orotidine Natural products OC1C(O)C(CO)OC1N1C(=O)NC(=O)C=C1C(O)=O FKCRAVPPBFWEJD-UHFFFAOYSA-N 0.000 description 1
- 229940000673 orphan drug Drugs 0.000 description 1
- 239000002859 orphan drug Substances 0.000 description 1
- 230000003647 oxidation Effects 0.000 description 1
- 238000007254 oxidation reaction Methods 0.000 description 1
- 239000002245 particle Substances 0.000 description 1
- 230000001717 pathogenic effect Effects 0.000 description 1
- 239000001814 pectin Substances 0.000 description 1
- 229920001277 pectin Polymers 0.000 description 1
- 235000010987 pectin Nutrition 0.000 description 1
- NBIIXXVUZAFLBC-UHFFFAOYSA-K phosphate Chemical compound [O-]P([O-])([O-])=O NBIIXXVUZAFLBC-UHFFFAOYSA-K 0.000 description 1
- 239000010452 phosphate Substances 0.000 description 1
- 150000003904 phospholipids Chemical class 0.000 description 1
- 229950004354 phosphorylcholine Drugs 0.000 description 1
- PYJNAPOPMIJKJZ-UHFFFAOYSA-N phosphorylcholine chloride Chemical compound [Cl-].C[N+](C)(C)CCOP(O)(O)=O PYJNAPOPMIJKJZ-UHFFFAOYSA-N 0.000 description 1
- 102000020233 phosphotransferase Human genes 0.000 description 1
- 238000000053 physical method Methods 0.000 description 1
- 230000004962 physiological condition Effects 0.000 description 1
- 230000035479 physiological effects, processes and functions Effects 0.000 description 1
- 239000006187 pill Substances 0.000 description 1
- 239000013600 plasmid vector Substances 0.000 description 1
- 239000001259 polydextrose Substances 0.000 description 1
- 229940035035 polydextrose Drugs 0.000 description 1
- 229920001223 polyethylene glycol Polymers 0.000 description 1
- 238000003752 polymerase chain reaction Methods 0.000 description 1
- 230000002035 prolonged effect Effects 0.000 description 1
- 230000001737 promoting effect Effects 0.000 description 1
- 235000019260 propionic acid Nutrition 0.000 description 1
- 229940095574 propionic acid Drugs 0.000 description 1
- 108060006633 protein kinase Proteins 0.000 description 1
- 235000011962 puddings Nutrition 0.000 description 1
- 238000000746 purification Methods 0.000 description 1
- 229940107700 pyruvic acid Drugs 0.000 description 1
- 238000012797 qualification Methods 0.000 description 1
- 238000004445 quantitative analysis Methods 0.000 description 1
- IUVKMZGDUIUOCP-BTNSXGMBSA-N quinbolone Chemical compound O([C@H]1CC[C@H]2[C@H]3[C@@H]([C@]4(C=CC(=O)C=C4CC3)C)CC[C@@]21C)C1=CCCC1 IUVKMZGDUIUOCP-BTNSXGMBSA-N 0.000 description 1
- 230000005855 radiation Effects 0.000 description 1
- 239000002516 radical scavenger Substances 0.000 description 1
- 239000002994 raw material Substances 0.000 description 1
- 239000011541 reaction mixture Substances 0.000 description 1
- 238000010223 real-time analysis Methods 0.000 description 1
- 230000000384 rearing effect Effects 0.000 description 1
- 229940047431 recombinate Drugs 0.000 description 1
- 210000000664 rectum Anatomy 0.000 description 1
- 235000020989 red meat Nutrition 0.000 description 1
- 230000009467 reduction Effects 0.000 description 1
- 238000007670 refining Methods 0.000 description 1
- 230000022532 regulation of transcription, DNA-dependent Effects 0.000 description 1
- 230000002787 reinforcement Effects 0.000 description 1
- 230000004044 response Effects 0.000 description 1
- 230000001177 retroviral effect Effects 0.000 description 1
- 230000002441 reversible effect Effects 0.000 description 1
- 108010033405 ribosomal protein S7 Proteins 0.000 description 1
- 108091092562 ribozyme Proteins 0.000 description 1
- 235000009566 rice Nutrition 0.000 description 1
- 101150116497 sacm1l gene Proteins 0.000 description 1
- 150000003839 salts Chemical class 0.000 description 1
- 238000007127 saponification reaction Methods 0.000 description 1
- CDAISMWEOUEBRE-UHFFFAOYSA-N scyllo-inosotol Natural products OC1C(O)C(O)C(O)C(O)C1O CDAISMWEOUEBRE-UHFFFAOYSA-N 0.000 description 1
- 239000013535 sea water Substances 0.000 description 1
- 230000028327 secretion Effects 0.000 description 1
- 238000012163 sequencing technique Methods 0.000 description 1
- 238000010008 shearing Methods 0.000 description 1
- 235000015170 shellfish Nutrition 0.000 description 1
- 150000004666 short chain fatty acids Chemical class 0.000 description 1
- 239000002002 slurry Substances 0.000 description 1
- 235000011888 snacks Nutrition 0.000 description 1
- 239000011734 sodium Substances 0.000 description 1
- 239000011780 sodium chloride Substances 0.000 description 1
- 159000000000 sodium salts Chemical class 0.000 description 1
- YWAFNFGRBBBSPD-OCMLZEEQSA-M sodium;[[(2r,3s,4r,5r)-5-(4-amino-2-oxopyrimidin-1-yl)-3,4-dihydroxyoxolan-2-yl]methoxy-oxidophosphoryl] 2-(trimethylazaniumyl)ethyl phosphate Chemical compound [Na+].O[C@@H]1[C@H](O)[C@@H](COP([O-])(=O)OP([O-])(=O)OCC[N+](C)(C)C)O[C@H]1N1C(=O)N=C(N)C=C1 YWAFNFGRBBBSPD-OCMLZEEQSA-M 0.000 description 1
- 238000000638 solvent extraction Methods 0.000 description 1
- 235000010199 sorbic acid Nutrition 0.000 description 1
- 239000004334 sorbic acid Substances 0.000 description 1
- 229940075582 sorbic acid Drugs 0.000 description 1
- 235000021262 sour milk Nutrition 0.000 description 1
- 235000012424 soybean oil Nutrition 0.000 description 1
- 239000003549 soybean oil Substances 0.000 description 1
- 229940033504 soybean preparation Drugs 0.000 description 1
- 235000019710 soybean protein Nutrition 0.000 description 1
- 230000002269 spontaneous effect Effects 0.000 description 1
- 230000010473 stable expression Effects 0.000 description 1
- 108010016092 sterol O-acyltransferase 2 Proteins 0.000 description 1
- 239000000021 stimulant Substances 0.000 description 1
- 230000004936 stimulating effect Effects 0.000 description 1
- 210000002784 stomach Anatomy 0.000 description 1
- 210000000352 storage cell Anatomy 0.000 description 1
- 238000007920 subcutaneous administration Methods 0.000 description 1
- 238000006467 substitution reaction Methods 0.000 description 1
- 239000005720 sucrose Substances 0.000 description 1
- 235000000346 sugar Nutrition 0.000 description 1
- 150000005846 sugar alcohols Chemical class 0.000 description 1
- 235000021092 sugar substitutes Nutrition 0.000 description 1
- 238000000194 supercritical-fluid extraction Methods 0.000 description 1
- 235000021147 sweet food Nutrition 0.000 description 1
- 239000003765 sweetening agent Substances 0.000 description 1
- 230000002194 synthesizing effect Effects 0.000 description 1
- 239000006188 syrup Substances 0.000 description 1
- 235000020357 syrup Nutrition 0.000 description 1
- 239000003760 tallow Substances 0.000 description 1
- QZZGJDVWLFXDLK-UHFFFAOYSA-N tetracosanoic acid Chemical compound CCCCCCCCCCCCCCCCCCCCCCCC(O)=O QZZGJDVWLFXDLK-UHFFFAOYSA-N 0.000 description 1
- ZTUXEFFFLOVXQE-UHFFFAOYSA-N tetradecanoic acid Chemical compound CCCCCCCCCCCCCC(O)=O.CCCCCCCCCCCCCC(O)=O ZTUXEFFFLOVXQE-UHFFFAOYSA-N 0.000 description 1
- RZWIIPASKMUIAC-VQTJNVASSA-N thromboxane Chemical compound CCCCCCCC[C@H]1OCCC[C@@H]1CCCCCCC RZWIIPASKMUIAC-VQTJNVASSA-N 0.000 description 1
- 210000001541 thymus gland Anatomy 0.000 description 1
- 230000005030 transcription termination Effects 0.000 description 1
- 238000005809 transesterification reaction Methods 0.000 description 1
- 230000001131 transforming effect Effects 0.000 description 1
- 230000010474 transient expression Effects 0.000 description 1
- 230000018412 transposition, RNA-mediated Effects 0.000 description 1
- 229940048102 triphosphoric acid Drugs 0.000 description 1
- 230000001228 trophic effect Effects 0.000 description 1
- 229960004799 tryptophan Drugs 0.000 description 1
- 239000002383 tung oil Substances 0.000 description 1
- 238000002525 ultrasonication Methods 0.000 description 1
- 229960005486 vaccine Drugs 0.000 description 1
- 238000001291 vacuum drying Methods 0.000 description 1
- 239000013598 vector Substances 0.000 description 1
- 108700026220 vif Genes Proteins 0.000 description 1
- 230000000007 visual effect Effects 0.000 description 1
- 235000019165 vitamin E Nutrition 0.000 description 1
- 229940046009 vitamin E Drugs 0.000 description 1
- 239000011709 vitamin E Substances 0.000 description 1
- 238000001262 western blot Methods 0.000 description 1
- 239000002023 wood Substances 0.000 description 1
- 230000029663 wound healing Effects 0.000 description 1
- 241000228158 x Triticosecale Species 0.000 description 1
- 235000013618 yogurt Nutrition 0.000 description 1
Images
Classifications
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/64—Fats; Fatty oils; Ester-type waxes; Higher fatty acids, i.e. having at least seven carbon atoms in an unbroken chain bound to a carboxyl group; Oxidised oils or fats
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23D—EDIBLE OILS OR FATS, e.g. MARGARINES, SHORTENINGS, COOKING OILS
- A23D9/00—Other edible oils or fats, e.g. shortenings, cooking oils
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/64—Fats; Fatty oils; Ester-type waxes; Higher fatty acids, i.e. having at least seven carbon atoms in an unbroken chain bound to a carboxyl group; Oxidised oils or fats
- C12P7/6409—Fatty acids
- C12P7/6427—Polyunsaturated fatty acids [PUFA], i.e. having two or more double bonds in their backbone
- C12P7/6434—Docosahexenoic acids [DHA]
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23K—FODDER
- A23K20/00—Accessory food factors for animal feeding-stuffs
- A23K20/10—Organic substances
- A23K20/158—Fatty acids; Fats; Products containing oils or fats
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23L—FOODS, FOODSTUFFS, OR NON-ALCOHOLIC BEVERAGES, NOT COVERED BY SUBCLASSES A21D OR A23B-A23J; THEIR PREPARATION OR TREATMENT, e.g. COOKING, MODIFICATION OF NUTRITIVE QUALITIES, PHYSICAL TREATMENT; PRESERVATION OF FOODS OR FOODSTUFFS, IN GENERAL
- A23L33/00—Modifying nutritive qualities of foods; Dietetic products; Preparation or treatment thereof
- A23L33/10—Modifying nutritive qualities of foods; Dietetic products; Preparation or treatment thereof using additives
- A23L33/115—Fatty acids or derivatives thereof; Fats or oils
- A23L33/12—Fatty acids or derivatives thereof
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
- A61P1/04—Drugs for disorders of the alimentary tract or the digestive system for ulcers, gastritis or reflux esophagitis, e.g. antacids, inhibitors of acid secretion, mucosal protectants
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P1/00—Drugs for disorders of the alimentary tract or the digestive system
- A61P1/14—Prodigestives, e.g. acids, enzymes, appetite stimulants, antidyspeptics, tonics, antiflatulents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P11/00—Drugs for disorders of the respiratory system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P17/00—Drugs for dermatological disorders
- A61P17/02—Drugs for dermatological disorders for treating wounds, ulcers, burns, scars, keloids, or the like
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P19/00—Drugs for skeletal disorders
- A61P19/02—Drugs for skeletal disorders for joint disorders, e.g. arthritis, arthrosis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P19/00—Drugs for skeletal disorders
- A61P19/08—Drugs for skeletal disorders for bone diseases, e.g. rachitism, Paget's disease
- A61P19/10—Drugs for skeletal disorders for bone diseases, e.g. rachitism, Paget's disease for osteoporosis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
- A61P25/20—Hypnotics; Sedatives
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
- A61P25/24—Antidepressants
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
- A61P25/28—Drugs for disorders of the nervous system for treating neurodegenerative disorders of the central nervous system, e.g. nootropic agents, cognition enhancers, drugs for treating Alzheimer's disease or other forms of dementia
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P25/00—Drugs for disorders of the nervous system
- A61P25/30—Drugs for disorders of the nervous system for treating abuse or dependence
- A61P25/32—Alcohol-abuse
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P27/00—Drugs for disorders of the senses
- A61P27/02—Ophthalmic agents
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P29/00—Non-central analgesic, antipyretic or antiinflammatory agents, e.g. antirheumatic agents; Non-steroidal antiinflammatory drugs [NSAID]
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P3/00—Drugs for disorders of the metabolism
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P3/00—Drugs for disorders of the metabolism
- A61P3/06—Antihyperlipidemics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P3/00—Drugs for disorders of the metabolism
- A61P3/08—Drugs for disorders of the metabolism for glucose homeostasis
- A61P3/10—Drugs for disorders of the metabolism for glucose homeostasis for hyperglycaemia, e.g. antidiabetics
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P43/00—Drugs for specific purposes, not provided for in groups A61P1/00-A61P41/00
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P9/00—Drugs for disorders of the cardiovascular system
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P9/00—Drugs for disorders of the cardiovascular system
- A61P9/10—Drugs for disorders of the cardiovascular system for treating ischaemic or atherosclerotic diseases, e.g. antianginal drugs, coronary vasodilators, drugs for myocardial infarction, retinopathy, cerebrovascula insufficiency, renal arteriosclerosis
-
- A—HUMAN NECESSITIES
- A61—MEDICAL OR VETERINARY SCIENCE; HYGIENE
- A61P—SPECIFIC THERAPEUTIC ACTIVITY OF CHEMICAL COMPOUNDS OR MEDICINAL PREPARATIONS
- A61P9/00—Drugs for disorders of the cardiovascular system
- A61P9/12—Antihypertensives
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/14—Fungi; Culture media therefor
- C12N1/16—Yeasts; Culture media therefor
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N1/00—Microorganisms, e.g. protozoa; Compositions thereof; Processes of propagating, maintaining or preserving microorganisms or compositions thereof; Processes of preparing or isolating a composition containing a microorganism; Culture media therefor
- C12N1/14—Fungi; Culture media therefor
- C12N1/16—Yeasts; Culture media therefor
- C12N1/18—Baker's yeast; Brewer's yeast
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/11—DNA or RNA fragments; Modified forms thereof; Non-coding nucleic acids having a biological activity
- C12N15/52—Genes encoding for enzymes or proenzymes
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/74—Vectors or expression systems specially adapted for prokaryotic hosts other than E. coli, e.g. Lactobacillus, Micromonospora
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/74—Vectors or expression systems specially adapted for prokaryotic hosts other than E. coli, e.g. Lactobacillus, Micromonospora
- C12N15/746—Vectors or expression systems specially adapted for prokaryotic hosts other than E. coli, e.g. Lactobacillus, Micromonospora for lactic acid bacteria (Streptococcus; Lactococcus; Lactobacillus; Pediococcus; Enterococcus; Leuconostoc; Propionibacterium; Bifidobacterium; Sporolactobacillus)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N15/00—Mutation or genetic engineering; DNA or RNA concerning genetic engineering, vectors, e.g. plasmids, or their isolation, preparation or purification; Use of hosts therefor
- C12N15/09—Recombinant DNA-technology
- C12N15/63—Introduction of foreign genetic material using vectors; Vectors; Use of hosts therefor; Regulation of expression
- C12N15/79—Vectors or expression systems specially adapted for eukaryotic hosts
- C12N15/80—Vectors or expression systems specially adapted for eukaryotic hosts for fungi
- C12N15/81—Vectors or expression systems specially adapted for eukaryotic hosts for fungi for yeasts
- C12N15/815—Vectors or expression systems specially adapted for eukaryotic hosts for fungi for yeasts for yeasts other than Saccharomyces
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/0004—Oxidoreductases (1.)
- C12N9/0071—Oxidoreductases (1.) acting on paired donors with incorporation of molecular oxygen (1.14)
- C12N9/0083—Miscellaneous (1.14.99)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12N—MICROORGANISMS OR ENZYMES; COMPOSITIONS THEREOF; PROPAGATING, PRESERVING, OR MAINTAINING MICROORGANISMS; MUTATION OR GENETIC ENGINEERING; CULTURE MEDIA
- C12N9/00—Enzymes; Proenzymes; Compositions thereof; Processes for preparing, activating, inhibiting, separating or purifying enzymes
- C12N9/10—Transferases (2.)
- C12N9/1025—Acyltransferases (2.3)
- C12N9/1029—Acyltransferases (2.3) transferring groups other than amino-acyl groups (2.3.1)
-
- C—CHEMISTRY; METALLURGY
- C12—BIOCHEMISTRY; BEER; SPIRITS; WINE; VINEGAR; MICROBIOLOGY; ENZYMOLOGY; MUTATION OR GENETIC ENGINEERING
- C12P—FERMENTATION OR ENZYME-USING PROCESSES TO SYNTHESISE A DESIRED CHEMICAL COMPOUND OR COMPOSITION OR TO SEPARATE OPTICAL ISOMERS FROM A RACEMIC MIXTURE
- C12P7/00—Preparation of oxygen-containing organic compounds
- C12P7/64—Fats; Fatty oils; Ester-type waxes; Higher fatty acids, i.e. having at least seven carbon atoms in an unbroken chain bound to a carboxyl group; Oxidised oils or fats
- C12P7/6436—Fatty acid esters
- C12P7/6445—Glycerides
- C12P7/6472—Glycerides containing polyunsaturated fatty acid [PUFA] residues, i.e. having two or more double bonds in their backbone
-
- A—HUMAN NECESSITIES
- A23—FOODS OR FOODSTUFFS; TREATMENT THEREOF, NOT COVERED BY OTHER CLASSES
- A23V—INDEXING SCHEME RELATING TO FOODS, FOODSTUFFS OR NON-ALCOHOLIC BEVERAGES AND LACTIC OR PROPIONIC ACID BACTERIA USED IN FOODSTUFFS OR FOOD PREPARATION
- A23V2002/00—Food compositions, function of food ingredients or processes for food or foodstuffs
Abstract
描述了能够生产占总油级分的10%以上的花生四烯酸(ARA,一种ω-6多不饱和脂肪酸)的含油酵母解脂耶氏酵母的工程化菌株。这些菌株包含多种表达异源去饱和酶、延伸酶和酰基转移酶的嵌合基因,任选包含多种天然去饱和酶和酰基转移酶敲除,从而能够合成和高度积累ARA。要求保护生产宿主细胞和在所述宿主细胞中生产ARA的方法。
Description
本申请要求2004年11月4日提交的美国临时申请No.60/624812的优先权。
发明领域
本发明涉及生物技术领域。更具体地,本发明涉及含油酵母解脂耶氏酵母(Yarrowia lipolytica)的工程化菌株,它能够以高浓度有效生产花生四烯酸(一种ω-6多不饱和脂肪酸)。
发明背景
花生四烯酸(ARA;顺式-5,8,11,14-二十碳四烯酸;ω-6)是生产类二十烷酸(如前列腺素、血栓烷、前列环素和leukot)的重要前体。此外,ARA被认为是:(1)一种必须的长链多不饱和脂肪酸(PUFA);(2)人脑中的主要ω-6脂肪酸;(3)母乳和很多婴儿配方中的重要成分,这是基于它在早期神经和视觉发育中的作用。尽管成人很容易从食物,如肉类、蛋和乳中获得ARA(并且也可以从饮食亚麻酸(LA)低效合成ARA),但ARA油的商业来源典型地由天然植物来源(如被孢霉(丝状真菌)属、虫霉属、腐霉属和Porphyridium(红藻)的微生物)生产。最值得注意的是,Martek Biosciences Corporation(Columbia,MD)生产一种含ARA的真菌油(U.S.5,658,767),其基本不含EPA,并且来源于高山被孢霉或Pythiuminsidiuosum。这种油的主要市场之一是婴儿配方;例如,含有Martek公司的ARA油的配方目前在世界范围内的60多个国家可以获得。
尽管可以从例如上文描述的天然微生物来源获得ARA,预计用重组方法通过微生物生产ARA相对于从天然微生物来源生产具有一些优点。例如,可以使用具有用于生产油的优选特征的重组微生物,因为通过在宿主中导入新的生物合成途径和/或通过阻抑不需要的途径,可以改变宿主的天然存在的微生物脂肪酸谱,从而导致需要的PUFAs(或其共轭形式)的生产水平增加和不需要的PUFAs的生产减少。其次,重组微生物可以提供特定形式的PUFAs,其可能具有特定用途。最后,可以通过控制培养条件操作微生物油生产,特别是通过提供微生物表达的酶的特定底物来源,或通过加入化合物/基因工程,以阻抑不需要的生物化学途径。因此,例如,可以改变如此产生的ω-3与ω-6脂肪酸的比例,或工程生产特定PUFA(如ARA),而不显著积累其它PUFA下游或上游产物。后一种可能性在本发明的某些实施方案中特别感兴趣,其中需要含有高浓度ARA的微生物油的重组来源,该微生物油额外地缺乏γ-亚麻酸(GLA;γ-亚麻酸;顺式-6,9,12-十八碳三烯酸;ω-6)。
GLA是从LA生物合成具有生物活性的前列腺素的重要中间体。尽管GLA也被认为是具有显著临床、生理和药物价值的必需ω-6PUFA,存在一些应用,其中GLA与ARA起相反作用。因此,商业生产包含ARA并且不含GLA的油将在某些应用中有用。
大多数微生物生产的ARA是通过Δ6去饱和酶/Δ6延伸酶途径(其主要存在于藻类、苔藓、真菌、线虫和人类)合成的,并且其中1.)通过Δ12去饱和酶的作用,油酸转化为LA;2.)通过Δ6去饱和酶的作用,LA转化为GLA;3.)通过C18/20延伸酶的作用,GLA转化为DGLA;和4.)通过Δ5去饱和酶的作用,DGLA转化为ARA(图1)。但是,在一些生物,如类眼虫鞭毛藻物种中,具有用于合成EPA的替代的Δ9延伸酶/Δ8去饱和酶途径,它是形成C20PUFAs的主要途径(Wallis,J.G.,and Browse,J.Arch.Biochem.Biophys.365:307-316(1999);WO 00/34439;和Qi,B.et al.FEBS Letters.510:159-165(2002))。在此途径中,通过Δ9延伸酶,LA转化为EDA,通过Δ8去饱和酶,EDA转化为DGLA,DGLA最终转化为ARA。
尽管目前已经从多种生物鉴定和表征了编码Δ6去饱和酶/Δ6延伸酶和Δ9延伸酶/Δ8去饱和酶途径的基因,并且其中的一些已经与其它PUFA去饱和酶和延伸酶一起异源表达,但这两种途径都还没有导入微生物,如酵母,并且通过复杂代谢工程进行操作,使得能够商业生产商业量的EPA(即大于总脂肪酸的10%)。此外,关于这些工程化的宿主生物的最合适的选择,还存在显著的分歧。
最近,Picataggio et al.(WO 2004/101757和共同未决的美国专利No.60/624812)开发了含油酵母,特别是解脂耶氏酵母(以前分类为解脂假丝酵母)作为用于生产PUFAs如ARA和EPA的微生物的优选类型的用途。含油酵母定义为天然能够合成并积累油的酵母,其中油的积累可以是细胞干重的最多大约80%。尽管这些生物中ω-6和ω-3脂肪酸的生产存在天然缺陷(由于天然生产的PUFAs限于18:2脂肪酸(较不常见是18:3脂肪酸)),Picataggio等人(上文)用相对简单的基因工程方法证明了在解脂耶氏酵母中生产了1.3% ARA和1.9% EPA(占总脂肪酸的比例)。但是,还没有实施更复杂的代谢工程化以便能够在这种特定宿主生物中进行ARA的经济的商业化生产。
申请人通过采用Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径,通过对能够生产总油级分的10-14%的ARA的解脂耶氏酵母菌株的工程化菌株解决了指出的问题(从而生产含有25-29% GLA的10-11% ARA油或不含GLA的10-11% ARA油)。提供了其它代谢工程化和发酵方法,以便进一步增强这些含油酵母中的ARA生产。
发明概述
本发明涉及具有用于生产花生四烯酸的酶途径的耶氏酵母属的重组生产宿主。
因此,本发明提供了用于生产花生四烯酸的重组生产宿主细胞,包括背景耶氏酵母种,其中包含基因集合,所述集合包含ω-3/ω-6脂肪酸生物合成途径的以下基因:
a)至少一种编码Δ6去饱和酶的基因;
b)至少一种编码C18/20延伸酶的基因;和
c)至少一种编码Δ5去饱和酶的基因;
其中所述ω-3/ω-6脂肪酸生物合成途径基因中的至少一种基因是超量表达的。
在本发明的另一种实施方案中,本发明提供了用于生产花生四烯酸的重组生产宿主细胞,包括背景耶氏酵母种,其中包含基因集合,所述集合包含ω-3/ω-6脂肪酸生物合成途径的以下基因:
a)至少一种编码Δ9延伸酶的基因;
b)至少一种编码Δ8去饱和酶的基因;和
c)至少一种编码Δ5去饱和酶的基因;
其中所述ω-3/ω-6脂肪酸生物合成途径基因中的至少一种基因是超量表达的。
在特定实施方案中,本发明的重组生产宿主还包含额外的途径成分,包括但不限于:至少一种编码Δ12去饱和酶的基因;至少一种编码Δ9去饱和酶的基因;至少一种编码C16/18延伸酶的基因;至少一种编码C14/16延伸酶的基因;和至少一种编码酰基转移酶的基因。
在一种特定的实施方案中,本发明提供了用于生产花生四烯酸的重组生产宿主细胞,包括背景耶氏酵母种,其中包含基因集合,所述集合包含ω-3/ω-6脂肪酸生物合成途径的以下基因:
a)至少一种编码Δ6去饱和酶的基因;和
b)至少一种编码C18/20延伸酶的基因;和
c)至少一种编码Δ5去饱和酶的基因;和
d)至少一种编码Δ12去饱和酶的基因;
其中该背景耶氏酵母种不含任何编码苹果酸异丙酯脱氢酶(Leu2-)的天然基因;并且
其中所述ω-3/ω-6脂肪酸生物合成途径基因中的至少一种基因是超量表达的。
在另一种特定的实施方案中,本发明提供了用于生产花生四烯酸的重组生产宿主细胞,包括背景耶氏酵母种,其中包含基因集合,所述集合包含ω-3/ω-6脂肪酸生物合成途径的以下基因:
a)至少一种编码Δ9延伸酶的基因;和
b)至少一种编码Δ8去饱和酶的基因;和
c)至少一种编码Δ5去饱和酶的基因;和
d)至少一种编码Δ12去饱和酶的基因;和
e)至少一种编码C16/18延伸酶的基因;
其中该背景耶氏酵母种不含任何编码酵母氨酸脱氢酶(Lys5-)的天然基因;并且
其中所述ω-3/ω-6脂肪酸生物合成途径基因中的至少一种基因是超量表达的。
在另一种实施方案中,本发明提供了生产包含花生四烯酸的微生物油的方法,包括:
a)培养权利要求1或2的生产宿主,其中生产了包含花生四烯酸的微生物油;和
b)任选回收步骤(a)的微生物油。
在另一种实施方案中,本发明提供了食品,其中包含有效量的通过本发明的方法生产的微生物油。
在另一种实施方案中,本发明提供了选自下组的产品:医学食物、营养补充物、婴儿配方和药物,其中包含有效量的通过本发明的方法生产的微生物油。
在另一种实施方案中,本发明提供了动物饲料,其中包含有效量的通过本发明的方法生产的微生物油。
本发明还提供了提供制备补充了花生四烯酸的产品、食品或饲料的方法,包括将本发明的方法生产的微生物油与产品、食品或饲料混合。
在另一种实施方案中,本发明提供了提供富含花生四烯酸(ARA)的人、动物或水产养殖生物饮食补充物的方法,包括提供通过本发明的方法生产的含花生四烯酸的微生物油,该微生物油是可被人或动物消耗或使用的形式。
生物保藏
以下生物材料保藏在美国典型培养物保藏中心(ATCC),10801University Boulevard,Manassas,VA 20110-2209,具有以下名称、保藏号和保藏日。
生物材料 保藏号 保藏日
质粒pY89-5 ATCC PTA-6048 2004年6月4日
解脂耶氏酵母Y2047 ATCC PTA-___
附图简述和序列说明
图1表示ω-3/ω-6脂肪酸生物合成途径。
图2是含油酵母中脂质积累的生化机理的示意图。
图3是描述多种酰基转移酶在含油酵母中的脂质积累中的作用的示意图。
图4描绘了产生总脂质级分中的多种脂肪酸(包括ARA)的本发明的一些解脂耶氏酵母菌株的开发图。
图5A提供了pY5-30的质粒图。图5B说明了通过组化染色确定的解脂耶氏酵母ATCC #76982菌株中TEF、GPD、GPM、FBA和FBAIN的相对启动子活性。图5C说明了通过组化染色确定的、多种培养基中生长的解脂耶氏酵母中YAT1、TEF、GPAT和FBAIN的相对启动子活性。
图6A是比较解脂耶氏酵母ATCC #76982菌株中GPD、GPM、FBA和FBAIN的启动子活性的图,所述活性是荧光分析确定的。图6B图示概括了实时PCR相对定量的结果,其中将解脂耶氏酵母ATCC#76982菌株(即表达GPD::GUS、GPDIN::GUS、FBA::GUS或FBAIN::GUS嵌合基因)中的GUS mRNA定量到表达pY5-30(即嵌合TEF::GUS基因)的解脂耶氏酵母菌株的mRNA水平。
图7提供了以下质粒的质粒图:(A)pY57.Yl.AHAS.w4971;(B)pKUNF12T6E;(C)pDMW232;和(D)pDMW271。
图8提供了以下质粒的质粒图:(A)pKUNT2;(B)pZUF17;(C)pDMW237;(D)pDMW240;和(E)酵母表达载体pY89-5。
图9显示了纤细眼虫细胞提取物的脂质谱的色谱图。
图10显示了多种纤细眼虫Δ8去饱和酶多肽序列的比对。采用的比对方法相应于“Clustal V比对法”。
图11提供了以下质粒的质粒图:(A)pKUNFmKF2;(B)pDMW277;(C)pZF5T-PPC;(D)pDMW287F;和(E)pDMW297。
图12提供了以下质粒的质粒图:(A)pZP2C16M899;(B)pDMW314;(C)pDM322;和(D)pZKL5598。
图13提供了以下质粒的质粒图:(A)pZP3L37;(B)pY37/F15;(C)pKO2UF2PE;和(D)pZKUT16。
图14提供了以下质粒的质粒图:(A)pKO2UM25E;(B)pZKUGPI5S;(C)pDMW302T16;和(D)pZKUGPE1S。
图15提供了以下质粒的质粒图:(A)pKO2UM26E;(B)pZUF-Mod-1;(C)pMDAGAT1-17;和(D)pMGPAT-17。
图16图示了SEQ ID NOs:97,98,99,100,101,102,103,104,105,106和107之间的关系,其中的每一个序列都与高山被孢霉中的甘油-3-磷酸o-酰基转移酶(GPAT)相关。
图17图示了SEQ ID NOs:53,54,55,56,57,58,59和60之间的关系,其中的每一个序列都与高山被孢霉中的C16/18脂肪酸延伸酶(ELO3)相关。
图18提供了以下质粒的质粒图:(A)pZUF6S;(B)pZUF6S-E3WT;(C)pZKUGPYE1-N;和(D)pZKUGPYE2。
图19提供了以下质粒的质粒图:(A)pZKUGPYE1;(B)pZUF6FYE1;(C)pZP217+Ura;(D)pY20;和(E)pLV13。
通过以下的详细说明和所附的序列说明能够更全面地理解本发明,这些内容构成了本发明的一部分。
以下序列符合37 C.F.R.§1.821-1.825的规定(“Requirements forPatent Applications Containing Nucleotide Sequences and/or AminoAcid Sequence Disclosures-the Sequence Rules”),并且,符合世界知识产权组织(WIPO)标准ST.25(1998)以及EPO和PCT的序列表要求(Rules 5.2 and 49.5(a-bis),and Section 208 and Annex Cof the Administrative Instructions)。用于核苷酸和氨基酸序列数据的符号和格式符合37 C.F.R.§1.822的规定。
SEQ ID NOs:1-112,158-168,209,252,255和357-364是编码表1中所列的启动子、基因或蛋白(或其片段)的ORFs。
表1
基因和蛋白序列号的概括
| 说明 | 核酸SEQ ID NO. | 蛋白SEQ ID NO. |
| 高山被孢霉Δ6去饱和酶 | 1(1374bp) | 2(457AA) |
| 合成的Δ6去饱和酶,来源于高山被孢霉,为在解脂耶氏酵母中表达进行了密码子优化 | 3(1374bp) | 2(457AA) |
| 高山被孢霉Δ6去饱和酶“B” | 4(1521bp) | 5(458AA) |
| 高山被孢霉Δ5去饱和酶 | 6(1341bp) | 7(446AA) |
| 球等鞭金藻(isochrysis galbana)Δ5去饱和酶 | 8(1329bp) | 9(442AA) |
| 合成的Δ5去饱和酶,来源于球等鞭金藻,为在解脂耶氏酵母中表达进行了密码子优化 | 10(1329bp) | 9(442AA) |
| 人Δ5去饱和酶 | 11(1335bp) | 12(444AA) |
| 合成的Δ5去饱和酶,来源于人,为在解脂耶氏酵母中表达进行了密码子优化 | 13(1335bp) | 12(444AA) |
| 异丝水霉Δ17去饱和酶 | 14(1077bp) | 15(358AA) |
| 合成的Δ17去饱和酶基因,来源于异丝水霉,为在解脂耶氏酵母中表达进行了密码子优化 | 16(1077bp) | 15(358AA) |
| 高山被孢霉C18/20延伸酶 | 17(957bp) | 18(318AA) |
| 合成的C18/20 延伸酶基因,来源于高山被孢霉,为在解脂耶氏酵母中表达进行了密码子优化 | 19(957bp) | 18(318AA) |
| 金黄色破囊壶菌C18/20延伸酶 | 20(819bp) | 21(272AA) |
| 合成的C18/20延伸酶基因,来源于金黄色破囊壶菌,为在解脂耶氏酵母中表达进行了密码子优化 | 22(819bp) | 21(272AA) |
| 解脂耶氏酵母Δ12去饱和酶 | 23(1936bp) | 24(419AA) |
| 深黄被孢霉Δ12去饱和酶 | 25(1203bp) | 26(400AA) |
| 串珠镰孢Δ12去饱和酶 | 27(1434bp) | 28(477AA) |
| 构巢曲霉Δ12去饱和酶 | 29(1416bp) | 30(471AA) |
| 黄曲霉Δ12去饱和酶 | -- | 31(466AA) |
| 烟曲霉Δ12去饱和酶 | -- | 32(424AA) |
| Magnaporthe griseaΔ12去饱和酶 | 33(1656bp) | 34(551AA) |
| 粗糙链孢霉Δ12去饱和酶 | 35(1446bp) | 36(481AA) |
| 禾谷镰孢Δ12去饱和酶 | 37(1371bp) | 38(456AA) |
| 高山被孢霉Δ12去饱和酶 | 357(1403bp) | 358(400AA) |
| Saccharomyces kluyveri Δ12去饱和酶 | -- | 359(416AA) |
| 乳克鲁维氏酵母Δ12去饱和酶 | 360(1948bp) | 361(415AA) |
| 白色假丝酵母Δ12去饱和酶 | -- | 362(436AA) |
| 汉逊氏德巴利氏酵母CBS767 Δ12去饱和酶 | -- | 363(416AA) |
| 球等鞭金藻Δ9延伸酶 | 39(792bp) | 40(263AA) |
| 合成的Δ9延伸酶基因,为在解脂耶氏酵母中表达进行了密码子优化 | 41(792bp) | 40(263AA) |
| 纤细眼虫Δ8去饱和酶基团(非功能性;GenBank获取号AAD45877) | 42(1275bp) | 43(419AA) |
| 纤细眼虫Δ8去饱和酶基因(非功能性;Wallis etal.[Archives of Biochem.Biophys.,365:307-316(1999)];WO 00/34439) | -- | 252(422AA) |
| 合成的Δ8去饱和酶基因,为在解脂耶氏酵母中表达进行了密码子优化(D8S-1) | 209(1270bp) | -- |
| 合成的Δ8去饱和酶基因,为在解脂耶氏酵母中表达进行了密码子优化(D8S-3) | 255(1269bp) | -- |
| 纤细眼虫Δ8去饱和酶基因(Eg5) | 44(1271bp) | 45(421AA) |
| 纤细眼虫Δ8去饱和酶基因(Eg12) | 46(1271bp) | 47(421AA) |
| 合成的Δ8去饱和酶基因,为在解脂耶氏酵母中表达进行了密码子优化(D8SF) | 48(1272bp) | 49(422AA) |
| 褐家鼠C16/18延伸酶 | 50(2628bp) | 51(267AA) |
| 合成的C16/18延伸酶基因,来源于褐家鼠,为在解脂耶氏酵母中表达进行了密码子优化 | 52(804bp) | 51(267AA) |
| 高山被孢霉C16/18延伸酶(ELO3) | 53(828bp) | 54(275AA) |
| 高山被孢霉ELO3—部分cDNA序列 | 55(607bp) | -- |
| 高山被孢霉ELO3—3’序列,通过基因组步移获得 | 56(1042bp) | -- |
| 高山被孢霉ELO3—5’序列,通过基因组步移获得 | 57(2223bp) | -- |
| 高山被孢霉ELO3—cDNA重叠群 | 58(3557bp) | -- |
| 高山被孢霉ELO3—内含子 | 59(542bp) | -- |
| 高山被孢霉ELO3—基因组重叠群 | 60(4099bp) | -- |
| 解脂耶氏酵母C16/18延伸酶基因 | 61(915bp) | 62(304AA) |
| 白色假丝酵母的可能的脂肪酸延伸酶(GenBank获取号EAL04510) | -- | 63(353AA) |
| 解脂耶氏酵母C14/16延伸酶基因 | 64(978bp) | 65(325AA) |
| 粗糙链孢霉FEN1基因(GenBank获取号CAD70918) | -- | 66(337AA) |
| 高山被孢霉溶血磷脂酸酰基转移酶(LPAAT1) | 67(945bp) | 68(314AA) |
| 高山被孢霉溶血磷脂酸酰基转移酶(LPAAT2) | 69(927bp) | 70(308AA) |
| 解脂耶氏酵母溶血磷脂酸酰基转移酶(LPAAT1) | 71(1549bp) | 72(282AA) |
| 解脂耶氏酵母溶血磷脂酸酰基转移酶(LPAAT2)—包含基因的基因组片段 | 73(1495bp) | -- |
| 解脂耶氏酵母溶血磷脂酸酰基转移酶(LPAAT2) | 74(672bp) | 75(223AA) |
| 解脂耶氏酵母磷脂:二酰基甘油酰基转移酶(PDAT) | 76(2326bp) | 77(648AA) |
| 解脂耶氏酵母酰基-CoA:固醇-酰基转移酶(ARE2) | 78(1632bp) | 79(543AA) |
| 秀丽新杆线虫酰基-CoA:1-酰基溶血磷脂酰胆碱酰基转移酶(LPCAT) | -- | 80(282AA) |
| 解脂耶氏酵母二酰基甘油酰基转移酶(DGAT1) | 81(1578bp) | 82(526AA) |
| 高山被孢霉二酰基甘油酰基转移酶(DGAT1) | 83(1578bp) | 84(525AA) |
| 粗糙链孢霉二酰基甘油酰基转移酶(DGAT1) | -- | 85(533AA) |
| 玉蜀黍赤霉PH-1二酰基甘油酰基转移酶(DGAT1) | -- | 86(499AA) |
| Magnaporthe grisea二酰基甘油酰基转移酶(DGAT1) | -- | 87(503AA) |
| 构巢曲霉二酰基甘油酰基转移酶(DGAT1) | -- | 88(458AA) |
| 解脂耶氏酵母二酰基甘油酰基转移酶(DGAT2) | 89(2119bp)91(1380bp)93(1068bp) | 90(514AA)92(459AA)94(355AA) |
| 高山被孢霉二酰基甘油酰基转移酶(DGAT2) | 95(996bp) | 96(331AA) |
| 高山被孢霉甘油-3-磷酸酰基转移酶(GPAT) | 97(2151bp) | 98(716AA) |
| 高山被孢霉GPAT—部分cDNA序列 | 99(1212bp) | -- |
| 高山被孢霉GPAT—包含1050bp至+2886bp区的基因组片段 | 100(3935bp) | -- |
| 高山被孢霉GPAT—3’cDNA序列,通过基因组步移获得 | 101(965bp) | -- |
| 高山被孢霉GPAT—5’序列,通过基因组步移获得 | 102(1908bp) | -- |
| 高山被孢霉GPAT—内部序列,通过基因组步移获得 | 103(966bp) | -- |
| 高山被孢霉GPAT—内含子#1 | 104(275bp) | -- |
| 高山被孢霉GPAT—内含子#2 | 105(255bp) | -- |
| 高山被孢霉GPAT—内含子#3 | 106(83bp) | -- |
| 高山被孢霉GPAT—内含子#4 | 107(99bp) | -- |
| 解脂耶氏酵母二酰基甘油胆碱磷酸转移酶(CPT1)—包含基因的基因组片段 | 108(2133bp) | -- |
| 解脂耶氏酵母二酰基甘油胆碱磷酸转移酶(CPT1) | 109(1185bp) | 110(394AA) |
| 啤酒糖酵母肌醇磷酸鞘脂特异性磷脂酶(ISC1) | 111(1434bp) | 112(477AA) |
| 解脂耶氏酵母甘油醛-3-磷酸脱氢酶启动子(GPD) | 158(971bp) | -- |
| 解脂耶氏酵母甘油醛-3-磷酸脱氢酶+内含子启动子(GPDIN) | 159(1174bp) | -- |
| 解脂耶氏酵母磷酸甘油变位酶启动子(GPM) | 160(878bp) | -- |
| 解脂耶氏酵母果糖-二磷酸醛缩酶启动子(FBA) | 161(1001bp) | -- |
| 解脂耶氏酵母果糖-二磷酸醛缩酶+内含子启动子(FBAIN) | 162(973bp) | -- |
| 解脂耶氏酵母果糖-二磷酸醛缩酶+修饰的内含子启动子(FBAINm) | 163(924bp) | -- |
| 解脂耶氏酵母甘油-3-磷酸酰基转移酶启动子(GPAT) | 164(1130bp) | -- |
| 解脂耶氏酵母铵转运蛋白启动子(YAT1) | 165(778bp) | -- |
| 解脂耶氏酵母翻译延伸因子EF1-α启动子(TEF) | 166(436bp) | -- |
| 解脂耶氏酵母嵌合GPM::FBA内含子启动子(GPM::FBAIN) | 167(1020bp) | -- |
| 解脂耶氏酵母嵌合GPM::GPD内含子启动子(GPM::GPDIN) | 168(1052bp) | -- |
| 解脂耶氏酵母输出蛋白启动子(EXP1) | 364(1000bp) | -- |
SEQ ID NOs:113-157是表2中指出的质粒。
表2
质粒序列号的概括
| 质粒 | 相应的附图 | SEQ ID NO |
| pY5-30 | 5A | 113(8,953bp) |
| pKUNF12T6E | 7B | 114(12,649bp) |
| pDMW232 | 7C | 115(10,945bp) |
| pDMW271 | 7D | 116(13,034bp) |
| pKUNT2 | 8A | 117(6,457bp) |
| pZUF17 | 8B | 118(8,165bp) |
| pDMW237 | 8C | 119(7,879bp) |
| pY54PC | -- | 120(8,502bp) |
| pKUNFmkF2 | 11A | 121(7,145bp) |
| pZF5T-PPC | 11C | 122(5,553bp) |
| pDMW297 | 11E | 123(10,448bp) |
| pZP2C16M899 | 12A | 124(15,543bp) |
| pDMW314 | 12B | 125(13,295bp) |
| pDMW322 | 12C | 126(11,435bp) |
| pZKSL5598 | 12D | 127(16,325bp) |
| pZP3L37 | 13A | 128(12,690bp) |
| pY37/F15 | 13B | 129(8,194bp) |
| pKO2UF2PE | 13C | 130(10,838bp) |
| pZKUT16 | 13D | 131(5,833bp) |
| pKO2UM25E | 14A | 132(12,663bp) |
| pZKUGPI5S | 14B | 133(6,912bp) |
| pDMW302T16 | 14C | 134(14,864bp) |
| pZKUGPE1S | 14D | 135(6,540bp) |
| pKO2UM26E | 15A | 136(13,321bp) |
| pZKUM | -- | 137(4,313bp) |
| pMLPAT-17 | -- | 138(8,015bp) |
| pMLPAT-Int | -- | 139(8,411bp) |
| pZUF-MOD-1 | 15B | 140(7,323bp) |
| pMDGAT1-17 | 15C | 141(8,666bp) |
| pMDGAT2-17 | -- | 142(8,084bp) |
| pMGPAT-17 | 15D | 143(9,239bp) |
| pZF5T-PPC-E3 | -- | 144(5,031bp) |
| pZUF6S | 18A | 145(8,462bp) |
| pZUF6S-E3WT | 18B | 146(11,046bp) |
| pZKUGPYE1-N | 18C | 147(6,561bp) |
| pZKUGPYE2 | 18D | 148(6,498bp) |
| pZUF6TYE2 | -- | 149(10,195bp) |
| pZKUGPYE1 | 19A | 150(6,561bp) |
| pZUF6FYE1 | 19B | 151(10,809bp) |
| pYCPT1-17 | -- | 152(8,273bp) |
| pZP217+Ura | 19C | 153(7,822bp) |
| pYCPT1-ZP217 | -- | 154(7,930bp) |
| pTEF::ISC1 | -- | 155(8,179bp) |
| pY20 | 19D | 156(8,196bp) |
| pLV13 | 19E | 157(5,105bp) |
SEQ ID NO:356相应于在耶氏酵母种中优化表达的基因的密码子优化的翻译起始位点。
SEQ ID NOs:169-182分别相应于用于扩增解脂耶氏酵母启动子区的引物YL211,YL212,YL376,YL377,YL203,YL204,GPAT-5-1,GPAT-5-2,ODMW314,YL341,ODMW320,ODMW341,27203-F和27203-R。
SEQ ID NOs:183-186分别是用于实时分析的寡核苷酸YL-URA-16F,YL-URA-78R,GUS-767F和GUS-891R。
SEQ ID NOs:187-202相应于8对寡核苷酸,其一起包括球等鞭金藻Δ9延伸酶的完整的密码子优化的编码区(即分别是IL3-1A,IL3-1B,IL3-2A,IL3-2B,IL3-3A,IL3-3B,IL3-4A,IL3-4B,IL3-5A,IL3-5B,IL3-6A,IL3-6B,IL3-7A,IL3-7B,IL3-8A和IL3-8B)。
SEQ ID NOs:203-206分别相应于用于在密码子优化的Δ9延伸酶基因的合成过程中进行PCR扩增的引物IL3-1F,IL3-4R,IL3-5F和IL3-8R。
SEQ ID NO:207是描述于pT9(1-4)的417bp NcoI/PstI片段,SEQ ID NO:208是描述于pT9(5-8)的377bp PstI/Not1片段。
SEQ ID NOs:210-235相应于13对寡核苷酸,其一起包括纤细眼虫Δ8去饱和酶的完整的密码子优化的编码区(即分别是D8-1A,D8-1B,D8-2A,D8-2B,D8-3A,D8-3B,D8-4A,D8-4B,D8-5A,D8-5B,D8-6A,D8-6B,D8-7A,D8-7B,D8-8A,D8-8B,D8-9A,D8-9B,D8-10A,D8-10B,D8-11A,D8-11B,D8-12A,D8-12B,D8-13A和D8-13B)。
SEQ ID NOs:236-243分别相应于用于在密码子优化的Δ8去饱和酶基因的合成过程中进行PCR扩增的引物D8-1F,D8-3R,D8-4F,D8-6R,D8-7F,D8-9R,D8-10F和D8-13R。
SEQ ID NO:244是描述于pT8(1-3)的309bp Nco/BgIII片段;SEQ ID NO:245是描述于pT8(4-6)的321bp BgIII/XhoI片段;SEQID NO:246是描述于pT8(7-9)的264bp XhoI/SacI片段;SEQ IDNO:247是描述于pT8(10-13)的369bp Sac1/Not1片段。
SEQ ID NOs:248和249分别相应于在pDMW255中合成D8S-2的过程中使用的引物ODMW390和ODMW391。
SEQ ID NOs:250和251是实施例7中描述的嵌合D8S-1::XPR和D8S-2::XPR基因。
SEQ ID NOs:253和254相应于D8S-3的合成过程中使用的引物ODMW392和ODMW393。
SEQ ID NOs:256和257分别相应于用于扩增纤细眼虫Δ8去饱和酶的引物Eg5-1和Eg3-3。
SEQ ID NOs:258-261相应于用于对Δ8去饱和酶克隆进行测序的引物T7,M13-28Rev,Eg3-2和Eg5-2。
SEQ ID NO:262相应于用于扩增D8S-3的引物ODMW404。
SEQ ID NO:263是包含D8S-3的1272bp嵌合基因。
SEQ ID NOs:264和265分别相应于用于在克隆的D8S-3基因中建立新的限制酶位点的引物YL521和YL522。
SEQ ID NOs:266-279分别相应于用于定点诱变反应中,以产生D8SF的引物YL525,YL526,YL527,YL528,YL529,YL530,YL531,YL532,YL533,YL534,YL535,YL536,YL537和YL538。
SEQ ID NO:280是突变AHAS基因,其包含W497L突变。
SEQ ID NOs:281-283分别相应于BD-Clontech Creator cDNA文库试剂盒引物SMART IV寡核苷酸,CDSIII/3’PCR引物和5’-PCR引物。
SEQ ID NO:284相应于用于高山被孢霉cDNA文库测序的M13正向引物。
SEQ ID NOs:285-288和290-291分别相应于用于克隆高山被孢霉LPAAT2 ORF的引物MLPAT-F,MLPAT-R,LPAT-Re-5-1,LPAT-Re-5-2,LPAT-Re-3-1和LPAT-Re-3-2。
SEQ ID NOs:289和292分别相应于解脂耶氏酵母LPAAT1 ORF的5’(1129bp)和3’(938bp)区。
SEQ ID NOs:293和294分别相应于用于建立pZUF-MOD-1“对照”质粒的引物pzuf-mod1和pzuf-mod2。
SEQ ID NOs:295和296分别相应于用于克隆高山被孢霉DGAT1ORF的引物MACAT-F1和MACAT-R。
SEQ ID NOs:297和298分别相应于用于克隆高山被孢霉DGAT2ORF的引物MDGAT-F和MDGAT-R1。
SEQ ID NOs:299和300分别相应于用于简并PCR中,以扩增高山被孢霉GPAT的引物MGPAT-N1和MGPAT-NR5。
SEQ ID NOs:301-303分别相应于用于扩增高山被孢霉GPAT的3’末端的引物MGPAT-5N1,MGPAT-5N2和MGPAT-5N3。
SEQ ID NOs:304和305相应于用于基因组步移(genome-walking)中的、来自ClonTech的Universal GenomeWalkerTM试剂盒的Genome Walker衔接子。
SEQ ID NOs:306-309分别相应于用于基因组步移中的PCR引物::MGPAT-5-1A,Adaptor-1(AP1),MGPAT-3N1和Nested AdaptorPrimer 2(AP2)。
SEQ ID NOs:310和311分别相应于用于扩增高山被孢霉GPAT的引物mgpat-cdna-5和mgpat-cdna-R。
SEQ ID NOs:312和313分别相应于用于基因组步移中,以分离高山被孢霉ELO3的3’末端区的引物MA Elong 3’1和MA elong 3’2。
SEQ ID NOs:314和315分别相应于用于基因组步移中,以分离高山被孢霉ELO3的5’末端区的引物MA Elong 5’1和MA elong 5’2。
SEQ ID NOs:316和317分别相应于用于从高山被孢霉cDNA扩增完整ELO3的引物MA ELONG 5’NcoI 3和MA ELONG 3’NotI 1。
SEQ ID NOs:318和319分别相应于用于扩增解脂耶氏酵母YE2的编码区的引物YL597和YL598。
SEQ ID NOs:320-323分别相应于用于扩增解脂耶氏酵母YE1的编码区的引物YL567,YL568,YL569和YL570。
SEQ ID NOs:324和325分别相应于用于克隆解脂耶氏酵母YE1的过程中的定点诱变的引物YL571和YL572。
SEQ ID NOs:326和327分别相应于用于克隆解脂耶氏酵母CPT1ORF的引物CPT1-5’-NcoI和CPT1-3’-NotI。
SEQ ID NOs:328和329分别相应于用于克隆啤酒糖酵母ISC1ORF的引物Isc1F和Isc1R。
SEQ ID NOs:330和331分别相应于用于克隆啤酒糖酵母PCL1ORF的引物Pcl1F和Pcl1R。
SEQ ID NOs:332-335分别相应于用于解脂耶氏酵母DGAT2基因的靶定破坏的引物P95,P96,P97和P98。
SEQ ID NOs:336-338分别相应于用于筛选破坏的解脂耶氏酵母DGAT2基因的靶定整合的引物P115,P116和P112。
SEQ ID NOs:339-342分别相应于用于解脂耶氏酵母PDAT基因的靶定破坏的引物P39,P41,P40和P42。
SEQ ID NOs:343-346分别相应于用于筛选破坏的解脂耶氏酵母PDAT基因的靶定整合的引物P51,P52,P37和P38。
SEQ ID NOs:347和348分别是用于分离解脂耶氏酵母DGAT1的鉴定为P201和P203的简并引物。
SEQ ID NOs:349-353分别相应于用于建立导向盒的引物P214,P215,P216,P217和P219,所述导向盒用于解脂耶氏酵母中推定的DGAT1基因的靶定破坏。
SEQ ID NOs:354和355分别相应于用于筛选破坏的解脂耶氏酵母DGAT1基因的靶定整合的引物P226和P227。
SEQ ID NOs:365-370分别相应于用于合成突变的解脂耶氏酵母AHAS基因的引物410,411,412,413,414和415,所述突变基因包含W497L突变。
SEQ ID NOs:371和372分别相应于用于扩增包含Aco3’终止子的NotI/PacI片段的引物YL325和YL326。
SEQ ID NO:373相应于存在于真菌Δ15和Δ12去饱和酶中的HisBox1基序。
SEQ ID NO:374相应于表明具有Δ15去饱和酶活性的真菌蛋白的基序,而SEQ ID NO:406相应于表明具有Δ12去饱和酶活性的真菌蛋白的基序。
发明详述
在此全文引入本文引用的所有专利、专利申请和公开文献作为参考。这特别包括了申请人的受让人的以下共同未决的申请:
美国专利申请No.10/840478(2004年5月6日提交),
美国专利申请No.10/840579(2004年5月6日提交),
美国专利申请No.10/840325(2004年5月6日提交)
美国专利申请No.10/869630(2004年6月16日提交),
美国专利申请No.10/882760(2004年7月1日提交),
美国专利申请No.10/985109(2004年11月10日提交),
美国专利申请No.10/987548(2004年11月12日提交)
美国专利申请No.60/624812(2004年11月4日提交),
美国专利申请No.11/024545和No.11/024544(2004年12月29日提交),
美国专利申请No.60/689031(2005年6月9日提交),
美国专利申请No.11/183664(2005年7月18日提交),
美国专利申请No.11/185301(2005年7月20日提交),
美国专利申请No.11/190750(2005年7月27日提交),
美国专利申请No.11/225354(2005年9月13日提交),CL2823和CL3027。
根据本发明,申请人提供了能够生产大于10%花生四烯酸(ARA,20:4,ω-6)的解脂耶氏酵母的生产宿主菌株。该特定多不饱和脂肪酸(PUFA)的积累是通过导入两个不同的功能性ω-3/ω-6脂肪酸生物合成途径中的任意一个而实现的。第一个途径包括将具有Δ6去饱和酶、C18/20延伸酶和Δ5去饱和酶活性的蛋白导入含油酵母以进行高水平重组表达,其中所述ARA油也包括GLA;后一个途径包括具有Δ9延伸酶、Δ8去饱和酶和Δ5去饱和酶活性的蛋白,从而使得能够生产不含任何GLA的ARA油。因此,本公开内容证明了可以对解脂耶氏酵母进行工程化,使得能够商业生产ARA及其衍生物。也要求保护生产方法。
本发明具有多种用途。通过本文所描述的方法制备的PUFAs,或其衍生物可被用作饮食代用品,或补充物,特别是婴儿配方,用于静脉内喂饲的患者或用于预防或治疗营养不良。另外,可以将纯化的PUFAs(或其衍生物)掺入烹饪油、脂肪,或配制的人造奶油中,以便在正常使用时,受体能接受到所需数量的饮食补充。还可将PUFAs掺入婴儿配方,营养补充物或其他食品中,并且可以用作抗炎剂或降胆固醇剂。可任选将所述组合物用于药物学用途(人或兽医)。在这种场合下,PUFAs通常是口服的,不过,可以通过能够成功地吸收它们的任何途径施用,例如,肠胃外(例如皮下,肌内或静脉内)、直肠内、阴道内或局部(例如,作为皮肤油膏或洗液)。
给人或动物补充通过重组方法生产的PUFAs,可以得到水平增加的添加的PUFAs,以及它们的代谢产物。例如,用ARA治疗,不仅能够产生水平增加的ARA,而且还能产生ARA的下游产物,如类二十烷酸。复杂的调节机制,可能需要组合各种PUFAs,或者添加PUFAs的不同的共轭物,以便预防、控制或克服这样的机制,以便在个体内获得理想水平的特定的PUFAs。
在其它实施方案中,通过本文公开的方法制备的PUFAs或其衍生物可以用于合成水产养殖饲料(即干饲料、半湿饲料和湿饲料),因为这些配方通常需要至少1-2%的营养组分是ω-3和/或ω-6 PUFAs。
定义
在本说明书中,使用了多种术语和缩写。提供了以下定义。
“可读框“缩写为ORF。
“聚合酶链反应”缩写为PCR。
“美国典型培养物保藏中心”缩写为ATCC。
“多不饱和脂肪酸”缩写为PUFA(s)。
“二酰基甘油酰基转移酶”缩写为DAG AT或DGAT。
“磷脂:二酰基甘油酰基转移酶”缩写为PDAT。
“甘油-3-磷酸酰基转移酶”缩写为GPAT。
“溶血磷脂酸酰基转移酶”缩写为LPAAT。
“酰基-CoA:1-酰基溶血磷脂酰胆碱酰基转移酶”缩写为“LPCAT”。
“酰基-CoA:固醇-酰基转移酶”缩写为ARE2。
“二酰基甘油”缩写为DAG。
“三酰基甘油”缩写为TAGs。
“辅酶A”缩写为CoA。
“磷脂酰胆碱”缩写为PC。
术语“串珠镰孢(Fusarium moniliforme)”与“Fusariumverticillioides”同义。
术语“食品”是指任何通常适于人类消耗的食物。典型的食品包括但不限于,肉产品、谷物产品、烘烤食品、快餐、乳制品等。
术语“功能性食品”是指包括可能有益健康的产品的食品,包括任何改良的食品或成分,其可提供超过它所包含的常规营养物质的健康益处。功能性食品可以包括诸如谷物、面包和饮料的食品,其可以用维生素、草药和营养品进行强化。功能性食品含有提供超过其营养价值的健康益处的物质,其中所述物质殖天然存在于食品中或是有意添加的。
此处用到的术语“医学食品”是指经过配制,以便在医生的监督下食用或施用的食品,其意欲用于具有独特营养需要的疾病或状况的特殊饮食控制,所述食用或施用是基于例如医学评估确立的公知科学原理[参见Orphan Drug Act(21 U.S.C.360ee(b)(3))的5(b)部分]。仅仅当满足以下条件时,食品是“医学食品”:(i)它是特殊配制并且加工的产品(与以天然状态使用的天然食料不同),用于通过口服或通过胃管进行的肠道喂饲对患者进行部分或全面喂饲;(ii)它意欲用于患者的营养控制,所述患者由于治疗或长期医学需要,摄取、消化、吸收或代谢常规食料或某些营养物质的能力受限或受损,或具有其它特殊的医学决定的营养需要,所述患者的营养控制可以通过对单独的正常饮食的改变而实现;(iii)它提供营养支持,所述营养支持针对独特营养需要的控制进行了特殊改变,所述独特的营养需要是通过医学评估确定的特定疾病或状况导致的;(iv)它意欲在医学监督下使用;和(v)它仅仅意欲用于接受活跃和进行中的医学监督的患者,其中所述患者需要反复的医学监督,包括但不限于对医学食品使用的说明。因此,与饮食补充物或常规食品不同,医学食品意欲用于对疾病或状况进行特殊的饮食控制,对所述疾病或状况建立了独特的营养需要,所述医学食品可能满足与给特定疾病或状况提供独特营养支持相关的科学上有效的要求。通过医学食品在医学监督下使用的要求,医学食品可以与广泛类别的特定饮食用途的食品(如低变应原性食品)和满足健康要求的食品(如营养补充物)区别。
本文用到的术语“药物”是指一种化合物或物质,如果在美国出售,必须受到联邦食品、药品和化妆品法案的505或505部分的控制。
术语“饮食补充物”是指一种产品,它:(i)意欲用于对饮食进行补充,因此不用作常规食品或作为膳食或饮食的唯一项目;(ii)含有一种或多种饮食成分(包括例如微生物、矿物质、草药或其它植物物质、氨基酸、酶和腺体)或其它组成成分;(iii)意欲作为丸剂、胶囊、片剂或液体通过口服摄取;和(iv)标为饮食补充物。
术语“临床状况”表示人类或动物中的状况,它损害健康和人类或动物的状态,并且通过补充PUFAs,特别是w-3和w-6脂肪酸可以纠正。临床状况可以是明确记载的疾病状态形式,如冠心病或由营养调节不良导致的不良健康的一般状况。
“食物类似物”是制造用于模拟其食品对应物(肉类、奶酪、乳等)的食品样产品,意欲具有食品对应物的外观、味道和质地。因此,此处用到的术语“食品”也包括食物类似物。
术语“水产养殖饲料”是指用于补充或替代水产养殖业中的天然饲料的制造的或人工的饮食(配制的饲料)。因此,水产养殖饲料是指用于饲养的有鳍鱼类和甲壳动物(即较低价值的食品鱼物种[如淡水有鳍鱼,如鲤鱼、罗非鱼和鲶鱼]和用于奢侈品或壁龛市场的较高价值的商品物种如虾、鲑鱼、鳟鱼、石首鱼、浅海石斑鱼(seebass)、海鲤(seebream)和石斑鱼])的人工配制的饲料。这些配制饲料包含彼此补充的各种比例的一些成分,形成用于水产养殖物种的营养补充食品。
术语“动物饲料”是指意欲专门用于动物,包括家养动物(宠物、畜牧业动物等)或用于生产食品的动物如饲养鱼类的饲料。
术语“饲料营养物”是指诸如蛋白、脂质、碳水化合物、维生素、矿物质和核酸的营养物质,它们可以来源于酵母生物物质,包括本发明的重组生产宿主。
本文用到的术语“生物物质”特别是指来自重组生产宿主的发酵的、消耗的或使用的酵母细胞材料,所述宿主以商业显著量生产ARA。
术语"脂肪酸"表示具有各种链长的长链脂族酸(链烷酸),从大约C12-C22(不过已知可以采用更长和更短链长的酸)。主要的链长为C16至C22。脂肪酸的结构是通过简单的符号系统"X:Y"表示的,其中,X是在特定脂肪酸中的碳(C)原子的总数,而Y是双键的数量。关于“饱和脂肪酸”与“不饱和脂肪酸”、“单不饱和脂肪酸”与“多不饱和脂肪酸”(或“PUFAs”),以及“ω-6脂肪酸(ω-6或n-6)与“ω-3脂肪酸(ω-3或n-3)的区别的其它细节提供于WO2004/101757。
在下面的表3中示出了用于描述本公开内容中的PUFAs的命名。在标题为"简化符号"的栏目中,利用ω-参考系统表示碳原子的编号,双键的编号,和最接近ω碳原子的双键的位置,从ω碳原子开始计数(它是用于这种目的的第1号)。该表的其余部分归纳了ω-3和ω-6脂肪酸的常用名,在说明书中将要使用的缩写,以及每一种化合物的化学名称。
表3
多不饱和脂肪酸的命名
| 通用名 | 缩写词 | 化学名称 | 简化符号 |
| 豆蔻酸 | -- | 十四烷酸 | 14:0 |
| 棕榈酸 | Palmitate | 十六烷酸 | 16:0 |
| 棕榈油酸 | -- | 9-十六烷酸 | 16:1 |
| 硬脂酸 | -- | 十八烷酸 | 18:0 |
| 油酸 | -- | 顺式-9-十八烷酸 | 18:1 |
| 亚油酸 | LA | 顺式-9,12-十八碳二烯酸 | 18:2 ω-6 |
| γ-亚油酸 | GLA | 顺式-6,9,12-十八碳三烯酸 | 18:3 ω-6 |
| 二十碳二烯酸 | EDA | 顺式-11,14-二十碳二烯酸 | 20:2 ω-6 |
| 二高-γ-亚油酸 | DGLA | 顺式-8,11,14-二十碳三烯酸 | 20:3 ω-6 |
| 花生四烯酸 | ARA | 顺式-5,8,11,14-二十碳四烯酸 | 20:4 ω-6 |
| α-亚麻酸 | ALA | 顺式-9,12,15-十八碳三烯酸 | 18:3 ω-3 |
| 十八碳四烯酸 | STA | 顺式-6,9,12,15-十八碳四烯酸 | 18:4 ω-3 |
| 二十碳三烯酸 | ETrA | 顺式-11,14,17-二十碳三烯酸 | 20:3 ω-3 |
| 二十碳四烯酸 | ETA | 顺式-8,11,14,17-二十碳四烯酸 | 20:4 ω-3 |
| 二十碳-五烯酸 | EPA | 顺式-5,8,11,14,17-二十碳五烯酸 | 20:5 ω-3 |
| 二十二碳-五烯酸 | DPA | 顺式-7,10,13,16,19-二十二碳五烯酸 | 22:5 ω-3 |
| 二十二碳-六烯酸 | DHA | 顺式-4,7,10,13,16,19-二十二碳六烯酸 | 22:6 ω-3 |
术语“高水平ARA生产”是指生产微生物宿主的总脂质的至少约5%的ARA,优选总脂质的至少约10%的ARA,更优选总脂质的至少约15%的ARA,更优选总脂质的至少约20%的ARA,最优选总脂质的至少约25-30%的ARA。ARA的结构形式是不限的;因此,例如,ARA可以作为游离脂肪酸或以酯化形式,如酰基甘油、磷脂、硫苷脂的形式存在于总脂质中。
术语“不含任何GLA”是指当采用具有最低至大约0.1%的可检测水平的装置通过GC分析测量时,微生物宿主的总脂质中缺乏任何可检测的GLA。
术语“必需脂肪酸”表示生物为了生存必须摄入的PUFA,所述生物不能从头合成所述特定必需脂肪酸。例如,哺乳动物不能合成必需脂肪酸亚油酸(18:2,ω-6)。其他必需脂肪酸包括GLA(ω-6),DGLA(ω-6),ARA(ω-6),EPA(ω-3)和DHA(ω-3)。
“微生物油”或“单细胞油”是由微生物在它们的生命过程中天然产生的油。这种油可能包括长链PUFAs。术语“油”表示在25℃下是液体并且通常是多不饱和的脂类。想法,术语“脂肪”表示脂类物质,它在25℃下是固体,并且通常是饱和的。
“脂质体”是指脂质小滴,其通常由特定蛋白和磷脂单层围绕。这些细胞器是大多数微生物转运/储存中性脂质的位点。认为脂质体来源于含有TAG生物合成酶的内质网的微结构域,并且,它们的合成和大小似乎由特定的蛋白成分控制。
“中性脂质”是指通常作为储存脂肪和油存在于脂质体中的那些脂质,如此命名是因为在细胞pH,这些脂质不具有带电基团。通常,它们完全是非极性的,对水没有亲和力。中性脂质通常是指甘油的脂肪酸一、二和/或三酯,分别也称作一酰基甘油、二酰基甘油或TAG(或统称为酰基甘油)。必须发生水解反应,以便从酰基甘油释放脂肪酸。
术语“三酰基甘油”、“油”和“TAGs”是指中性脂质,包含酯化于甘油分子的三个脂肪酰基(所述术语在本发明中可互换使用)。所述油可以包括长链PUFAs、较短的饱和和不饱和脂肪酸,以及较长链的饱和脂肪酸。因此,“油生物合成”通常是指细胞中TAGs的合成。
术语“酰基转移酶”是指负责转转移氨基-酰基以外的基团的酶(EC2.3.1.-)。
术语“DAG AT”是指二酰基甘油酰基转移酶(也称作酰基-CoA-二酰基甘油酰基转移酶或二酰基甘油O-酰基转移酶)(EC 2.3.1.20)。这种酶负责将酰基-CoA和1,2-二酰基甘油转化为TAG和CoA(从而包括在TAG生物合成的终末步骤中)。存在两个家族的DAG AT酶:DGAT1和DGAT2。前一个家族与酰基-CoA:胆固醇酰基转移酶(ACAT)基因家族具有同源性,而后一个家族是不相关的(Lardizabalet al.,J.Biol.Chem.276(42):38862-38869(2001))。
术语“PDAT”是指磷脂:二酰基甘油酰基转移酶(EC 2.3.1.158)。这种酶负责将酰基从磷脂的sn-2位置转移到1,2-二酰基甘油的sn-3位置,从而得到溶血磷脂和TAG(从而包括在TAG生物合成的终末步骤中)。这种酶与DGAT(EC 2.3.1.20)的差异在于通过不依赖于酰基-CoA的机制合成TAG。
术语“ARE2”是指酰基-CoA:固醇-酰基转移酶(EC 2.3.1.26;也称作固醇-酯合酶2),其催化以下反应:酰基-CoA+固醇=CoA+固醇酯。
术语“GPAT”是指由gpat基因编码的甘油-3-磷酸O-酰基转移酶(E.C.2.3.1.15),其将酰基-CoA和sn甘油3-磷酸转化为1-酰基-sn-甘油3-磷酸(磷脂生物合成的第一步)。
术语“LPAAT”是指溶血磷脂酸-酰基转移酶(EC 2.3.1.51)。该酶负责将酰基-CoA基团转移到1-酰基-sn-甘油3-磷酸(即溶血磷脂酸)上,得到CoA和1,2-二酰基-sn-甘油3-磷酸(磷脂酸)。文献中也将LPAAT称作酰基-CoA:1-酰基-sn-甘油-3-磷酸2-O-酰基转移酶、1-酰基-sn-甘油-3-磷酸酰基转移酶和/或1-酰基甘油磷酸酰基转移酶(缩写为AGAT)。
术语“LPCAT”是指酰基-CoA:1-酰基溶血磷脂酰-胆碱酰基转移酶。这种酶负责在CoA和磷脂酰胆碱(PC)之间交换酰基。因此,它也称作参与CoA和其它磷脂,包括溶血磷脂酸(LPA)之间的酰基交换的酶。
“总脂质和油级分中的PUFAs百分比(%)”是指这些级分中PUFAs相对于总脂肪酸的百分比。术语“总脂质级分”或“脂质级分”都指含油生物内的所有脂质的总和(即中性和极性),由此包括位于磷脂酰胆碱(PC)级分、磷脂酰乙醇胺(PE)级分和三酰基甘油(TAG或油)级分中的脂质。但是,该术语“脂质”和“油”在说明书中可以互换使用。
术语“磷脂酰胆碱”或“PC”是指作为细胞膜的主要组成部分的磷脂。PC的化学结构可以通常描述为包含以下成分:胆碱分子、磷酸基团和甘油,其中脂肪酰基链作为R基团连接在甘油分子的sn-1和sn-2位置上。
术语“PUFA生物合成途径酶”表示与PUFA的生物合成相关的以下任何酶(以及编码所述酶的基因),包括:Δ4去饱和酶、Δ5去饱和酶、Δ6去饱和酶、Δ12去饱和酶、Δ15去饱和酶、Δ17去饱和酶、Δ9去饱和酶、Δ8去饱和酶、Δ9延伸酶、C14/16延伸酶、C16/18延伸酶、C18/20延伸酶和/或C20/22延伸酶。
术语“ω-3/ω-6脂肪酸生物合成途径”表示一组基因,当它们在合适的条件下表达时,编码能催化ω-3和/或ω-6脂肪酸生产的酶。通常,参与ω-3/ω-6脂肪酸生物合成途径的基因编码某些或所有以下的酶:Δ12去饱和酶、Δ6去饱和酶、C18/20延伸酶、C20/22延伸酶、Δ9延伸酶、Δ5去饱和酶、Δ17去饱和酶、Δ15去饱和酶、Δ9去饱和酶、Δ8去饱和酶、Δ9延伸酶和Δ4去饱和酶。在图1中示出了典型的途径,它可以经各种中间体将油酸转化成DHA,并且证实了如何由普通来源生产ω-3和ω-6脂肪酸。该途径天然分成两个部分,其中一个部分产生ω-3脂肪酸,另一个部分仅仅产生ω-6脂肪酸。仅仅产生ω-3脂肪酸的部分在此称作ω-3脂肪酸生物合成途径,而仅仅产生ω-6脂肪酸的部分在此称作ω-6脂肪酸生物合成途径。
在ω-3/ω-6脂肪酸生物合成途径的范围中使用的术语“功能性”表示途径中的一些(或所有)基因表达活性酶,导致体内催化或底物转化。应该理解,“ω-3/ω-6脂肪酸生物合成途径”或“功能性ω-3/ω-6脂肪酸生物合成途径”不表示需要前面段落中列出的所有基因,因为很多脂肪酸产物仅仅需要表达该途径的一个亚组的基因。
术语“Δ6去饱和酶/Δ6延伸酶途径”是指最少包括以下基因的ARA脂肪酸生物合成途径:Δ6去饱和酶、C18/20延伸酶和Δ5去饱和酶。在一种相关方式中,术语“Δ9延伸酶/Δ8去饱和酶途径”是指最少包括以下基因的ARA脂肪酸生物合成途径:Δ9延伸酶、Δ8去饱和酶和Δ5去饱和酶。
术语“去饱和酶”表示能够对一种或多种脂肪酸去饱和,即,导入双键,以便产生脂肪酸或感兴趣的前体的多肽。尽管在本说明书中ω-参考系统被用于表示特定的脂肪酸,更方便的是,利用Δ-系统从底物的羧基末端开始计数以表示去饱和酶的活性。本发明特别感兴趣的是:1.)Δ8去饱和酶,它使脂肪酸的位于从该分子的羧基末端开始计数的第8和第9个碳原子之间去饱和,例如,催化EDA转化成DGLA和/或EtrA转化成ETA;2.)Δ6去饱和酶,它能催化LA转化成GLA和/或ALA转化成STA;3.)Δ5去饱和酶,它能催化DGLA转化成ARA和/或ETA转化成EPA;4.)Δ4去饱和酶,它能催化DPA转化成DHA;5.)Δ12去饱和酶,它能催化油酸转化成LA;6.)Δ15去饱和酶,它能催化LA转化成ALA和/或GLA转化成STA;7.)Δ17去饱和酶,它能催化ARA转化成EPA和/或DGLA转化成ETA;和8)Δ9去饱和酶,它能催化棕榈酸转化成棕榈油酸(16:1)和/或硬脂酸转化成油酸(18:1)。
针对本发明Δ15去饱和酶提到的术语“双功能”表示该多肽具有用油酸和亚油酸作为酶底物的能力。“酶底物”表示该多肽在活性位点与底物结合,并且以反应性方式作用于该底物。
术语“延伸酶系统”表示负责延伸脂肪酸碳链,以便产生比延伸酶作用的脂肪酸底物长2个碳原子的脂肪酸的一组四种酶。更具体地,这种延伸过程是与脂肪酸合酶相关发生的,其中,CoA是酰基载体(Lassner et al.,The Plant Cell 8:281-292(1996))。在第一步中(其是底物特异性和限速的),丙二酰-CoA与长链酰基-CoA缩合,产生CO2和β-酮酰基-CoA(其中,酰基部分延长了两个碳原子)。随后的反应包括还原成β-羟酰基-CoA,脱水形成烯酰基-CoA,并且第二次还原,产生延长的酰基-CoA。由延伸酶催化的反应的例子是将GLA转化成DGLA,STA转化成ETA和将EPA转化成DPA。
为了本发明的目的,催化第一个缩合反应(即丙二酰-CoA转化为β-酮酰基-CoA)的酶统称为“延伸酶”。概言之,延伸酶的底物特异性一定程度是宽范围的,但是由于链长度和不饱和化的程度和类型而不同。因此,延伸酶可以具有不同的特异性。例如,C14/16延伸酶利用C14底物(如豆蔻酸),C16/18延伸酶利用C16底物(如棕榈酸),C18/20延伸酶利用C18底物(如GLA、STA),C20/22延伸酶利用C20底物(如EPA)。以相似的方式,Δ9延伸酶能够催化LA和ALA分别转化为EDA和EtrA。重要的是注意,一些延伸酶具有广泛特异性,因此,单个酶可能能够催化一些延伸酶反应(例如,由此既作为C16/18延伸酶,也作为C18/20延伸酶)。在一种优选的实施方案中,最理想的是经验确定脂肪酸延伸酶的特异性,该确定是通过用脂肪酸延伸酶的基因转化合适的宿主,并且确定其对宿主的脂肪酸谱的影响。
术语“高亲和力延伸酶”或“EL1S”或“ELO1”是指底物特异性优先针对GLA的C18/20延伸酶(DGLA是延伸酶反应的产物[即Δ6延伸酶])。一种这样的延伸酶描述于WO 00/12720,在此提供为SEQID NOs:17和18。但是,申请人已经证明了该酶也对18:2(LA)和18:3(ALA)具有一些活性。因此,SEQ ID NO:18显示出Δ9延伸酶活性(以及其Δ6延伸酶活性)。因此,概括出此处提供为SEQ IDNO:18的C18/20延伸酶可以在本发明中描述的Δ6去饱和酶/Δ6延伸酶途径中和Δ9延伸酶/Δ8去饱和酶途径中起作用,作为例如球等鞭金藻Δ9延伸酶(SEQ ID NO:40)的替代物。
术语“EL2S”或“ELO2”是指底物特异性优先针对GLA(DGLA是延伸酶反应的产物)和/或STA(STA是延伸酶反应的底物)的C18/20延伸酶。一种这样的延伸酶描述于U.S.6,677,145,在此提供为SEQ IDNOs:20和21。
术语“ELO3”是指由elo3基因(SEQ ID NO:53)编码的高山被孢霉C16/18脂肪酸延伸酶(在此提供为SEQ ID NO:54)。术语“YE2”是指由此处提供为SEQ ID NO:61的基因编码的解脂耶氏酵母C16/18脂肪酸延伸酶(在此提供为SEQ ID NO:62)。基于此处报道的数据,ELO3和YE2都优先催化棕榈酸(16:0)转化为硬脂酸(18:0)。
术语“YE1”是指由此处提供为SEQ ID NO:64的基因编码的解脂耶氏酵母C14/16脂肪酸延伸酶(此处提供为SEQ ID NO:65)。基于此处报道的数据,YE2优先催化豆蔻酸(14:0)转化为棕榈酸(16:0)。
术语“转化效率”和“底物转化百分比”表示特定的酶(例如,去饱和酶或延伸酶)可以将底物转化成产物的效率。转化效率是按照以下公式计算的:([产物]/[底物+产物])*100,其中‘产物’包括中间产物和衍生自中间产物的所有产物。
术语“含油的”表示倾向于以脂质形式储存它们的能源的生物(Weete,In:Fungal Lipid Biochemistry,2nd ed.,Plenum,1980)。通常,所述微生物的细胞油含量遵循S形曲线,其中,脂质的浓度增加,直到在晚对数期或早期静止生长期的最大值,然后在晚期静止期和死亡期逐渐减少(Yongmanitchai and Ward,Appl.Environ,Microbiol.57:419-25(1991))。
术语“含油酵母”表示被分类为酵母的微生物,它们能够产生油。通常,含油微生物的细胞油或三酰基甘油含量遵循S形曲线,其中,脂质的浓度增加,直到在晚对数期或早期静止生长期的最大值,然后在晚期静止期和死亡期逐渐减少(Yongmanitchai and Ward,Appl.Environ.Microbiol.57:419-25(1991))。含油微生物积累占干细胞重量的大约25%以上的油并非是不常见的。含油酵母的例子包括,但不局限于以下属:耶氏酵母属,假丝酵母属,红酵母属,红冬孢属,隐球酵母属,丝孢酵母属和油脂酵母属。
术语“可发酵的碳源”表示微生物能够代谢以便产生能量的碳源。本发明的典型的碳源包括,但不局限于:单糖,寡糖,多糖,链烷,脂肪酸,脂肪酸酯,甘油一酯,甘油二酯,甘油三酯,二氧化碳,甲醇,甲醛,甲酸盐或酯以及含碳的胺。
在本文中,“分离的核酸片段”是单链或双链RNA或DNA的聚合物,它任选包括合成的,非天然的或改变了的核酸碱基。DNA聚合物形式的分离的核酸片段可能包含一个或多个cDNA、基因组DNA或合成DNA的片段。
氨基酸或核苷酸序列的“主要部分”是包括多肽的一定数目的氨基酸序列或基因的核苷酸序列的部分,所述数目足够通过本领域技术人员对所述序列进行的人工评估,或通过计算机自动化的序列比较,以及使用诸如BLAST的算法的鉴定(Basic Local Alignment SearchTool;Altschul,S.F.,et al.,J.Mol.Biol.215:403-410(1993)而推测性地鉴定该多肽或基因。一般地,为了推测性地鉴定一种多肽或核酸序列与已知的蛋白或基因同源,具有10个或更多个连续氨基酸或30个或更多个核苷酸的序列是必须的。此外,对于核苷酸序列,可以将包含20-30个连续核苷酸的基因特异性寡核苷酸探针用于基因鉴定(如Southern杂交)和分离(如细菌菌落或噬菌体斑的原位杂交)的序列依赖性方法中。此外,可以将12-15个碱基组成的短寡核苷酸用作PCR中的扩增引物,以获得包括引物的特定核酸片段。因此,核苷酸序列的“主要部分”包括足够的序列,以便特异性地鉴定和/或分离包括所述序列的核酸片段。
术语“互补的”被用于表示能够彼此杂交的核苷酸碱基之间的相互关系。例如,对于DNA来说,腺嘌呤互补于胸腺嘧啶,而胞嘧啶互补于鸟嘌呤。
“密码子简并性”表示遗传密码的性质,它允许改变核苷酸序列,而又不影响所编码的多肽的氨基酸序列。技术人员在用核苷酸密码子表示特定氨基酸时充分理解由特定宿主细胞所表现出来的“密码子偏倚”。因此,在合成用于改善在宿主细胞中的表达的基因时,需要设计所述基因,以便它的密码子选择的频率接近宿主细胞的优选密码子选择的频率。
表示DNA序列的“化学合成的”表示组成核苷酸是在体外组装的。DNA的人工化学合成,可以通过使用业已建立的方法完成;或可以利用多种商业化仪器中的一种进行自动化的化学合成。“合成基因”可以由寡核苷酸基本单位组装,它们是利用本领域技术人员所公知的方法化学合成的。将这些基本单位连接在一起并且退火,以便形成基因片段,然后酶促组装,以便构建完整的基因。因此,可以对基因进行修饰,以便根据核苷酸序列的优化优化基因表达,以便体现宿主细胞的密码子偏倚。如果密码子选择偏向宿主优选的密码子,技术人员可以理解成功的基因表达的可能性。优选密码子的确定可以根据对来自宿主细胞的基因的检查,其中,可以获得序列信息。
“基因”表示能表达特定蛋白的核酸片段,其可以表示单独的编码序列,或可以包括位于编码序列前面(5’非编码序列)和后面(3’非编码序列)的调节序列。“天然基因”表示天然与它自身的调节序列一起存在的基因。“嵌合基因”表示不是天然基因的任何基因,包括不是天然一起存在的调节和编码序列。因此,嵌合基因可以包括来自不同来源的调节序列和编码序列,或来自相同的来源,但是以不同于天然存在的方式排列的调节序列和编码序列。“内源基因”表示存在于生物基因组中的天然位置上的天然基因。“外源”基因表示通过基因转移导入所述宿主生物的基因。外源基因可以包括插入非天然生物的天然基因,或嵌合基因。“转基因”是业已通过转化方法导入所述基因组的基因。“密码子优化的基因”是具有经过设计以便模拟宿主细胞的优选的密码子选择频率的密码子选择频率的基因。
“编码序列”表示编码特定氨基酸序列的DNA序列。“合适的调节序列”表示位于编码序列上游(5’非编码序列),序列内,或下游(3’非编码序列)的核苷酸序列,并且,它能影响相关编码序列的转录、RNA加工或稳定性、或翻译。调节序列可以包括启动子、翻译前导序列、内含子、聚腺苷酸化识别序列、RNA加工位点、效应物结合位点和茎-环结构。
“启动子”表示能够控制编码序列或功能性RNA表达的DNA序列。一般,编码序列位于启动子序列的3’。启动子可能完全来自天然基因,或者包括来自天然存在的不同启动子的不同元件,或者甚至包括合成的DNA片段。本领域技术人员可以理解的是,不同的启动子能够指导基因在不同的组织或细胞类型中的表达,或者在发育的不同阶段的表达,或者对不同的环境或生理学条件作出反应的表达。能导致基因在大多数细胞类型中在大多数时间表达的启动子通常被称作“组成型启动子”。还应当理解的是,由于在大多数场合下,调节序列的确切边界未能完全确定,不同长度的DNA片段可能具有相同的启动子活性。
术语“GPAT启动子”或“GPAT启动子区”是指在gpat基因编码的甘油-3-磷酸O-酰基转移酶(E.C.2.3.1.15)的‘ATG’翻译起始密码子前面,并且对于表达是必须的5’上游非翻译区。合适的解脂耶氏酵母GPAT启动子区的实例描述于美国专利申请No.11/225354。
术语“GPD启动子”或“GPD启动子区”是指在gpd基因编码的甘油醛-3-磷酸脱氢酶(E.C.1.2.1.12)的‘ATG’翻译起始密码子前面,并且对于表达是必须的5’上游非翻译区。合适的解脂耶氏酵母GPD启动子区的实例描述于WO 2005/003310。
术语“GPM启动子”或“GPM启动子区”是指在gpm基因编码的磷酸甘油变位酶(EC 5.4.2.1)的‘ATG’翻译起始密码子前面,并且对于表达是必须的5’上游非翻译区。合适的解脂耶氏酵母GPM启动子区的实例描述于WO 2005/003310。
术语“FBA启动子”或“FBA启动子区”是指在fba1基因编码的果糖-二磷酸醛缩酶(E.C.4.1.2.13)的‘ATG’翻译起始密码子前面,并且对于表达是必须的5’上游非翻译区。合适的解脂耶氏酵母FBA启动子区的实例描述于WO 2005/049805。
术语“FBAIN启动子”或“FBAIN启动子区”是指在fba1基因的‘ATG’翻译起始密码子前面并且对于表达是必须的5’上游非翻译区,加上具有fba1基因的内含子的5’编码区的一部分。合适的解脂耶氏酵母FBAIN启动子区的实例描述于WO 2005/049805。
术语“GPDIN启动子”或“GPDIN启动子区”是指在gpd基因的‘ATG’翻译起始密码子前面并且对于表达是必须的5’上游非翻译区,加上具有gpd基因的内含子的5’编码区的一部分。合适的解脂耶氏酵母GPDIN启动子区的实例描述于美国专利申请No.11/183664。
术语“YAT1启动子”或“YAT1启动子区”是指在yat1基因编码的铵转运蛋白酶(TC 2.A.49;GenBank获取号XM_504457)的‘ATG’翻译起始密码子前面,并且对于表达是必须的5’上游非翻译区。合适的解脂耶氏酵母YAT1启动子区的实例描述于美国专利申请No.11/185301。
术语“EXP1启动子”或“EXP1启动子区”是指在解脂耶氏酵母“YALI0C12034g”基因(GenBank获取号XM_501745)编码的蛋白的‘ATG’翻译起始密码子前面,并且对于表达是必须的5’上游非翻译区。基于“YALI0C12034g”与sp|Q12207啤酒糖酵母非经典输出蛋白2(其功能涉及缺乏可切割的信号序列的蛋白的输出的新途径)的显著同源性,该基因在此命名为exp1基因,其编码命名为EXP1的蛋白。合适的解脂耶氏酵母EXP1启动子区的实例描述为SEQ IDNO:364,但不意欲限制其性质。本领域技术人员可以理解,由于EXP1启动子的精确边界未能完全确定,具有增加或减少的长度的的DNA片段可能具有相同的启动子活性。
术语“启动子活性”表示对启动子的转录效力的评估。例如,这可以通过测量从启动子转录的mRNA的量而直接测定(例如通过Northern印迹或引物延伸法)或通过测量从启动子表达的基因产物的量而检测测定。
“内含子”是大多数真核生物的基因序列(编码区、5’非编码区或3’非编码区)中存在的非编码DNA的序列。它们的完整功能还未知,但是,一些增强子位于内含子中(Giacopelli F.et al.,Gene Expr.11:95-104(2003))。这些内含子序列是转录的,但是在mRNA翻译为蛋白之前,从前mRNA转录物中除去。内含子除去的这一过程是通过内含子的任一侧上的序列(外显子)的自我剪接发生的。
术语“增强子”是指能提高邻近的真核启动子的转录水平,从而增加基因转录的顺式调节序列。增强子可以作用于超过数万碱基的DNA上的启动子,并且可以在它们调节的启动子的5’或3’。增强子也可以位于内含子中。
术语“3’非编码序列”和“转录终止子”表示位于编码序列下游的DNA序列。它包括聚腺苷酸化识别序列以及编码能够影响mRNA加工或基因表达的调节信号的其他序列。聚腺苷酸化信号通常以影响聚腺苷酸添加在mRNA前体的3’末端为特征。所述3’区能够影响相关编码序列的转录、RNA加工或稳定性,或翻译。
“RNA转录物”表示由RNA聚合酶催化的DNA序列转录而得到的产物。当RNA转录物是所述DNA序列的完全互补的拷贝时,它被称作初级转录物,或者它可以是来自初级转录物的转录后加工的RNA序列,并且被称作成熟RNA。“信使RNA”或“mRNA”表示没有内含子,并且能够由细胞翻译成蛋白的RNA。“cDNA”表示双链DNA,它是互补于mRNA并且由mRNA衍生的。“有义”RNA表示包括mRNA,并能够由细胞翻译成蛋白的RNA转录物。“反义”RNA表示互补于靶初级转录物或mRNA的全部或部分的RNA,并且它能抑制靶基因的表达(U.S.5,107,065;WO 99/28508)。反义RNA的互补性可以是与特定基因转录物的任何部分,即,在5’非编码序列、3’非编码序列或编码序列处互补的。“功能性RNA”表示反义RNA、核酶RNA、或不能翻译,但仍然能影响细胞加工的其他RNA。
术语“可操作地连接的”表示核酸序列缔合在单个核酸片段上,以便其中一个的功能受到另一个的影响。例如,当启动子能够影响编码序列的表达时,它就是与该编码序列可操作地连接的(即,所述编码序列受所述启动子的转录控制)。编码序列能够沿有义或反义方向可操作地与调节序列连接。
在本文中,术语“表达”表示来自本发明的核酸片段的有义(mRNA)或反义RNA的转录和稳定积累。表达还可以表示mRNA翻译成多肽。
“成熟”蛋白表示翻译后加工的多肽;即,业已除去了存在于初级翻译产物中的所有前肽或原肽的蛋白。“前体”蛋白表示mRNA翻译的初级产物;即,仍然存在前肽和原肽。前肽和原肽可以是(但不局限于)细胞内定位信号。
术语“重组酶”表示进行位点特异性重组,以改变DNA结构的酶,包括转座酶、λ整合/切割酶,以及位点特异性重组酶。
“重组酶位点”或“位点特异性重组酶序列”表示一种DNA序列,它能够被重组酶识别和结合。可以理解,其可以是野生型或突变的重组酶位点,只要保持了功能性,并且重组酶仍然可以识别该位点、结合于DNA序列,并且催化两个相邻重组酶位点之间的重组。
“转化”表示将核酸分子转入宿主生物,导致了遗传学稳定的遗传性。例如,核酸分子可以是能自主复制的质粒;或者它可以整合到宿主生物的基因组中。包括转化的核酸片段的宿主生物被称作“转基因”或“重组”或“转化的”生物。
术语“质粒”,“载体”和“盒”表示染色体外元件,它通常携带有不作为细胞的中心代谢部分的基因,并且通常是环状双链DNA片段形式的。所述元件可以是自主复制的序列、基因组整合序列、噬菌体或核苷酸序列、线性或环状的单或双链DNA或RNA,可以来自任何来源,其中,多个核苷酸序列业已连接或重组成独特的结构,该结构能够将启动子片段和选定的基因产物的DNA序列,以及合适的3’非翻译序列导入细胞。“表达盒”表示包括外源基因,并且除了外源基因之外,还具有能够增强基因在外源宿主中表达的元件的特定载体。
术语“同源重组”表示两个DNA分子之间的DNA片段的交换(在交换过程中)。交换的片段的侧翼是两个DNA分子之间的相同核苷酸序列的位点(即“同源区”)。术语“同源区”是指核酸片段上参与同源重组的、彼此具有同源性的核苷酸序列段。当这些同源区的长度为至少约10bp,优选长度为至少约50bp时,通常发生有效同源重组。典型地,意欲用于重组的片段含有至少两个同源区,其中需要靶定的基因破坏或替代。
术语“序列分析软件”表示可用于分析核苷酸或氨基酸序列的任何计算机算法或软件程序。“序列分析软件”可以是通过商业渠道获得的或者独立开发的。典型的序列分析软件包括,但不局限于:1.)GCG程序组(Wisconsin Package Version 9.0,Genetics ComputerGroup(GCG),Madison,WI);2.)BLASTP,BLASTN,BLASTX(Altschul et al.,J.Mol.Biol.215:403-410(1990));3.)DNASTAR(DNASTAR,Inc.,Madison,WI);和4.)采用了Smith-Waterman算法的FASTA程序(W.R.Pearson,Comput.Methods Genome Res.,[Proc.Int.Symp.](1994),Meeting Date 1992,111-20.Editor(s):Suhai,Sandor.Plenum:New York,NY)。在本申请的范围内应当理解的是,在将序列分析软件用于分析时,分析的结果是基于有关程序的“缺省值”的,除非另有说明。在本文中,“缺省值”表示在初次初始化时最初随所述软件加载的任何组的值或参数。
术语“保守结构域”或“基序”表示在沿着进行比对的进化上相关的蛋白的序列的特定位置上保守的一组氨基酸。尽管其它位置的氨基酸在同源蛋白之间可以不同,但在特定位置高度保守的氨基酸表明这些氨基酸是蛋白的结构、稳定性或活性必须的。由于它们是通过在比对的蛋白同源物家族的序列中的高保守程度鉴定的,它们可以用作标识物,或“信号”,来确定具有新确定的序列的蛋白是否属于以前鉴定的蛋白家族。表明具有Δ15去饱和酶活性的真菌蛋白的基序提供为SEQ ID NO:374,而表明具有Δ12去饱和酶活性的真菌蛋白的基序提供为SEQ ID NO:375。
本发明所使用的标准重组DNA和分子克隆技术是本领域众所周知的,并且描述于以下文献中:Sambrook,J.,Fritsch,E.F.andManiatis,T.,Molecular Cloning:A Laboratory Manual,2nd ed.,Cold Spring Harbor Laboratory:Cold Spring Harbor,NY(1989)(下文称作“Maniatis”);Silhavy,T.J.,Bennan,M.L.and Enquist,L.W.,Experiments with Gene Fusions,Cold Spring HarborLaboratory:Cold Spring Harbor,NY(1984);和Greene PublishingAssoc.and Wiley-Interscience(1987)出版的Ausubel,F.M.et al.,Current Protocols in Molecular Biology。
用于生产ARA的优选微生物宿主:解脂耶氏酵母
在申请人的工作之前(参见Picataggio et al.,WO2004/101757),以前还没有将含油酵母检验为适于用作PUFAs的生产平台的一类微生物。通常鉴定为含油酵母的属包括但不限于:但不局限于:耶氏酵母属、假丝酵母属、红酵母属、红冬孢属、隐球酵母属、丝孢酵母属和油脂酵母属。更具体地讲,典型的油合成酵母包括:类酵母红冬孢、斯达氏油脂酵母、产油油脂酵母、拉可夫氏假丝酵母、铁红假丝酵母、热带假丝酵母、产朊假丝酵母、茁芽丝孢酵母、皮状丝孢酵母、胶粘红酵母、禾木科红酵母和解脂耶氏酵母(以前被划分为解脂假丝酵母)。
认为含油酵母具有一些性质,能够促进它们用作ARA的经济、商业生产的宿主生物。首先,这些生物定义为天然能够合成和积累油的那些,其中油可以占细胞干重的大约25%以上,更优选占细胞干重的大约30%以上,最优选占细胞干重的大约40%以上。其次,已经充分开发了培养具有高油含量的含油酵母的技术(例如,参见EP 0 005277B1;Ratledge,C.,Prog.Ind.Microbiol.16:119-206(1982))。这些生物过去已经在商业上用于多种目的。例如,已经将解脂耶氏酵母的多种菌株用于制造和生产以下物质:异柠檬酸裂解酶(DD259637);酯酶(SU1454852,WO2001083773,DD279267);多羟基链烷酸(WO2001088144);柠檬酸(RU2096461,RU2090611,DD285372,DD285370,DD275480,DD227448,PL160027);赤藓糖醇(EP770683);2-氧戊二酸(DD267999);γ-癸内酯(U.S.6,451,565,FR2734843);γ-十二内酯(EP578388);和丙酮酸(JP09252790)。
在分类为含油酵母的生物中,为了此处的目的,将解脂耶氏酵母选作优选的微生物宿主。这种选择基于以下知识,即含油菌株能够将ω-3脂肪酸级分掺入TAG级分中,该生物易于进行遗传操作,和以前将该物种作为食品级柠檬酸的通常认识到的安全(“GRAS”,根据美国食品和药品监督管理局)来源。在进一步的实施方案中,根据针对具有高脂质含量(测量为干重的百分比)和高体积产率(测量为g/L h-1)的野生型菌株的鉴定的初步研究,最优选的是命名为ATCC#20362、ATCC #8862、ATCC #18944、ATCC #76982和/或LGAM S(7)1的解脂耶氏酵母菌株(Papanikolaou S.,and Aggelis G.,Bioresour.Technol.82(1):43-9(2002))。
如WO 2004/101757中的描述,以前通过导入和表达编码ω-3/ω-6生物合成途径的基因,对解脂耶氏酵母进行基因工程化,以分别生产1.3% ARA和1.9% EPA。更具体地,分别转化两种不同的DNA表达构建体(包含用于合成ARA的Δ6去饱和酶、Δ5去饱和酶和高亲和力PUFA C18/20延伸酶或用于合成EPA的Δ6去饱和酶、Δ5去饱和酶、高亲和力PUFA C18/20延伸酶和密码子优化的Δ17去饱和酶),并且整合到编码乳清苷-5’-磷酸脱羧酶(EC 4.1.1.23)的解脂耶氏酵母染色体URA3基因中。用合适的底物饲养的宿主细胞的GC分析检测到了ARA和EPA的生产。尽管适于证明进行基因工程化以生产ω-6和ω-3脂肪酸的含油宿主的能力,但该工作没有进行复杂的代谢工程化,所述代谢工程化是合成占总油级分的5%以上的ARA,或更优选占总油级分的10%以上的ARA,或甚至更优选占总油级分的15-20%以上的ARA,或最优选占总油级分的25-30%以上的ARA所需要的。
在共同未决的美国专利申请No.60/624812中,进行了解脂耶氏酵母中的复杂的代谢工程化,以便:(1)鉴定允许合成和大量积累EPA的优选去饱和酶和延伸酶;(2)操作酰基转移酶的活性,所述酰基转移酶使得能够将ω脂肪酸转移到存储脂质库中;(3)通过使用强启动子、以多拷贝表达和/或密码子优化,超量表达去饱和酶、延伸酶和酰基转移酶;(4)下调PUFA生物合成中减少EPA的总积累的特定基因的表达;和(5)操作影响EPA生产的途径和总体调节剂。这导致在解脂耶氏酵母的一个特定重组菌株中生产了最高达28%的EPA。
在本申请中,进行了类似的复杂代谢工程化,导致在解脂耶氏酵母的重组菌株中生产了占总油级分的10-14%的ARA。更具体地,对菌株Y2034和Y2047进行基因工程化,以便利用Δ6去饱和酶/Δ6延伸酶途径,并且生产分别包含10% ARA和11% ARA的油;对菌株Y2214进行基因工程化,以便利用Δ9延伸酶/Δ8去饱和酶途径,从而生产包含12% ARA并且不含GLA的油。下文将讨论代谢工程化的方面,并且将讨论发酵方法,以进一步增强该含油酵母中的ARA生产能力。
概述:脂肪酸和甘油三酯的微生物生物合成
一般地,含油微生物中的脂质积累是反应于生长培养基中存在的总体碳氮比而引发的。导致游离棕榈酸(16:0)在含油微生物中从头合成的该过程详细描述于2004/101757。棕榈酸是长链饱和和不饱和脂肪酸衍生物的前体,其通过延伸酶和去饱和酶的作用形成。例如,通过Δ9去饱和酶的作用,棕榈酸转化为其不饱和衍生物[棕榈油酸(16:1)]。类似地,棕榈酸由C16/18脂肪酸延伸酶延伸形成硬脂酸(18:0),其可以通过Δ9去饱和酶转化为其不饱和衍生物,从而产生油酸(18:1)。
通过一系列反应形成TAGs(脂肪酸的主要存储单位),所述一系列反应包括:1.)一个分子的酰基-CoA通过酰基转移酶酯化为甘油-3-磷酸,以产生溶血磷脂酸;2.)第二个分子的酰基-CoA通过酰基转移酶酯化,产生1,2-二酰基甘油磷酸(通常鉴定为磷脂酸);3.)由磷脂酸磷酸酶除去一个磷酸,产生1,2-二酰基甘油(DAG);和4.)通过酰基转移酶的作用加入第三个脂肪酸,以形成TAG(图2)。
可以将广泛的脂肪酸掺入TAGs,包括饱和和不饱和脂肪酸,以及短链和长链脂肪酸。可以通过酰基转移酶掺入TAGs的脂肪酸的一些非限定性实例包括:癸酸(10:0)、月桂酸(12:0)、豆蔻酸(14:0)、棕榈酸(16:0)、棕桐油酸(16:1)、硬脂酸(18:0)、油酸(18:1)、11-十八碳酸烯酸(18:1)、LA、桐酸(18:3)、ALA、GLA、花生酸(20:0)、EDA、EtrA、DGLA、ETA、ARA、EPA、山俞酸(22:0)、DPA、DHA、二十四酸(24:0)、神经酸(24:1)、蜡酸(26:0)和褐煤酸(28:0)脂肪酸。在本发明的一种优选实施方案中,最理想的是将EPA掺入TAG。
ARA,即一种ω-3脂肪酸的生物合成
将油酸转化为ARA的代谢过程包括通过加入碳原子而进行的碳链延伸和通过加入双键使分子去饱和。这需要内质网膜中存在一系列的去饱和酶和延伸酶。但是,如图1所示和下文的描述,ARA生产存在两种替代途径。
具体地,所有途径都需要通过Δ12去饱和酶的作用最初将油酸转化为LA(18:2),即ω-6脂肪酸的第一个。然后,利用ARA生物合成的“ω-6 Δ6去饱和酶/Δ6延伸酶途径”,PUFAs通过如下过程形成:(1)通过Δ6去饱和酶的作用,LA转化为GLA;(2)通过C18/20延伸酶的作用,GLA转化为DGLA;和(3)通过Δ5去饱和酶的作用,DGLA转化为ARA。
或者,通过“ω-6 Δ9延伸酶/Δ8去饱和酶途径”,LA通过Δ9延伸酶的作用转化为EDA。然后,Δ8去饱和酶将EDA转化为DGLA。随后,如上文所述,DGLA随后通过Δ5去饱和酶的作用去饱和,产生ARA。
为了清楚,在下表中概括了这些途径的每一个,以及它们的区别特征:
表4
ARA生物合成的替代生物合成途径
| 名称 | 合成ARA最少需要的基因* | 途径 |
| Δ6去饱和酶/Δ6延伸酶途径 | Δ6D,C18/20 ELO,Δ5D | --- |
| Δ9延伸酶/Δ8去饱和酶途径 | Δ9 ELO,Δ8D,Δ5D | 生产不含GLA的油 |
| 组合Δ6去饱和酶/Δ6延伸酶途径和Δ9延伸酶/Δ8去饱和酶途径 | Δ6D,C18/20 ELO,Δ9ELO,Δ8D,Δ5D |
*缩写:“D”=去饱和酶;“ELO”=延伸酶.
如果需要,可以用ARA作为底物,生产一些其它的PUFAs。例如,通过Δ17去饱和酶的作用,ARA可以进一步去饱和为EPA,随后通过C20/22延伸酶和Δ4去饱和酶的作用,转化为DHA。
选择用于合成ARA的微生物基因
需要导入解脂耶氏酵母以生产ARA的特定功能性将依赖于宿主细胞(及其天然PUFA谱和/或去饱和酶/延伸酶谱)、底物的可获得性和需要的终产物。至于天然宿主细胞,已知解脂耶氏酵母可以天然产生18:2脂肪酸,因此具有天然Δ12去饱和酶(SEQ ID NOs:23和24;参见WO 2004/104167)。至于需要的终产物,上文已经在如此产生的油的最终脂肪酸谱(即最终的高ARA油组合物中的%GLA)方面描述了与Δ9延伸酶/Δ8去饱和酶途径表达不同的Δ6去饱和酶/Δ6延伸酶途径表达的结果。
因此,在一些实施方案中,需要通过Δ6去饱和酶/Δ6延伸酶途径生产ARA。因此,为了进行ARA生物合成,最少必须将以下基因导入宿主生物并表达:Δ6去饱和酶、C18/20延伸酶和Δ5去饱和酶。在进一步优选的实施方案中,宿主菌株还包含下组的至少一种:Δ9去饱和酶、Δ12去饱和酶、C14/16延伸酶和C16/18延伸酶。
在一种替代的实施方案中,需要生产ARA,而不共同合成GLA(因此需要表达Δ9延伸酶/Δ8去饱和酶途径)。因此,为了进行ARA生物合成,该策略最少必须将以下基因导入宿主生物并表达:Δ9延伸酶、Δ8去饱和酶和Δ5去饱和酶。在进一步优选的实施方案中,宿主菌株还包含下组的至少一种:Δ9去饱和酶、Δ12去饱和酶、C14/16延伸酶和C16/18延伸酶。
本领域技术人员将能够鉴定编码ARA生物合成所需的每一种酶的多种候选基因。有用的去饱和酶和延伸酶序列可以来源于任何来源,如分离自天然来源(如细菌、藻类、真菌、植物、动物等),通过半合成途径产生或从头合成。尽管导入宿主中的去饱和酶和延伸酶基因的特定来源对于本发明不是关键的,但用于选择具有去饱和酶或延伸酶活性的特定多肽的考虑包括:1)多肽的底物特异性;2)多肽或其成分是否是限速酶;3.)去饱和酶或延伸酶对于需要的PUFA的合成是否是必须的;和/或4.)多肽需要的辅因子。表达的多肽优选具有与其在宿主细胞中的位置的生化环境相容的参数。例如,多肽可能必须与宿主细胞中的其它酶竞争底物。因此,在确定特定多肽对于改变特定宿主细胞中的PUFA生产的合适性中,可能需要考虑多肽的KM分析和比活性。用于特定宿主细胞中的多肽是可以在目的宿主细胞中存在的生化条件下起作用的多肽,但也可以是具有能够改变需要的PUFA的去饱和酶或延伸酶活性的任何多肽。
在其它实施方案中,考虑每种特定去饱和酶和/或延伸酶的转化效率也是有用的。更具体地,由于每种酶很少以100%的效率将底物转化为产物,在宿主细胞中生产的未纯化油的最终脂质谱将典型是多种PUFAs的混合物,其由需要的ARA和多种上游中间体PUFAs组成(例如,与100% ARA油不同)。因此,当对ARA的生物合成进行优化时,考虑每种酶的转化效率也是重要的,必须根据产物的最终需要的脂质谱进行考虑。
根据上述每一种考虑,可以根据公众可获得的文献(如GenBank)、专利文献和具有生产PUFAs的能力的微生物的实验分析,鉴定具有合适去饱和酶和延伸酶活性的候选基因。例如,以下GenBank获取号表示公众可获得的用于ARA生物合成的基因的实例:AY131238,Y055118,AY055117,AF296076,AF007561,L11421,NM_031344,AF465283,AF465281,AF110510,AF465282,AF419296,AB052086,AJ250735,AF126799,AF126798(Δ6去饱和酶);AF199596,AF226273,AF320509,AB072976,AF489588,AJ510244,AF419297,AF07879,AF067654,AB022097(Δ5去饱和酶);AAG36933,AF110509,AB020033,AAL13300,AF417244,AF161219,AY332747,AAG36933,AF110509,AB020033,AAL13300,AF417244,AF161219,X86736,AF240777,AB007640,AB075526,AP002063(Δ12去饱和酶);NP_441622,BAA18302,BAA02924,AAL36934(Δ15去饱和酶);AF338466,AF438199,E11368,E11367,D83185,U90417,AF085500,AY504633,NM_069854,AF230693(Δ9去饱和酶);AF390174(Δ9延伸酶);AF139720(Δ8去饱和酶);和NP_012339,NP_009963,NP_013476,NP_599209,BAB69888,AF244356,AAF70417,AAF71789,AF390174,AF428243,NP_955826,AF206662,AF268031,AY591335,AY591336,AY591337,AY591338,AY605098,AY605100,AY630573(C14/16,C16/18和C18/20延伸酶)。类似地,专利文献提供了很多参与PUFA生产的基因的额外DNA序列(和/或关于上述一些基因和它们的分离方法的细节)[例如,WO02/077213(Δ9延伸酶);WO 00/34439和WO 04/057001(Δ8去饱和酶);U.S.5,968,809(Δ6去饱和酶);U.S.5,972,664和U.S.6,075,183(Δ5去饱和酶);WO 94/11516,U.S.5,443,974,WO 03/099216和WO 05/047485(Δ12去饱和酶);WO 93/11245(Δ15去饱和酶);WO 91/13972和U.S.5,057,419(Δ9去饱和酶);U.S.2003/0196217 A1(Δ17去饱和酶);以及WO 00/12720,U.S.6,403,349,U.S.6,677,145,U.S.2002/0139974A1,U.S.2004/0111763(C14/16,C16/18和C18/20延伸酶)]。在此全文引入这些专利和申请作为参考。
上文的实例不是限制性的,来源于不同来源的许多其它基因也适于导入解脂耶氏酵母,所述其它基因编码(1)Δ6去饱和酶、C18/20延伸酶和Δ5去饱和酶(和任选的其它编码Δ9去饱和酶、Δ12去饱和酶、C14/16延伸酶和/或C16/18延伸酶的基因);或(2)Δ9延伸酶、Δ8去饱和酶和Δ5去饱和酶(和任选的其它编码Δ9去饱和酶、Δ12去饱和酶、C14/16延伸酶和/或C16/18延伸酶的基因)。
用于ARA合成的优选基因
尽管可以广泛选自能够适于在解脂耶氏酵母中表达的去饱和酶和延伸酶,但是,在本发明的优选实施方案中,去饱和酶和延伸酶选自以下这些(或其衍生物):
表5
用于在解脂耶氏酵母中进行ARA生物合成的优选去饱和酶和延伸酶
| ORF | 生物 | 参考文献 | SEQ IDNOs |
| Δ6去饱和酶 | 高山被孢霉 | GenBank获取号AF465281;U.S.5,968,809 | 1,2 |
| Δ6去饱和酶 | 高山被孢霉 | GenBank获取号AB070555 | 4,5 |
| C18/20延伸酶(“ELO1”) | 高山被孢霉 | GenBank获取号AX464731;WO 00/12720 | 17,18 |
| C18/20延伸酶(“ELO2”) | 金黄色破囊壶菌 | U.S.6,677,145 | 20,21 |
| Δ9延伸酶 | 球等鞭金藻 | GenBank获取号AF390174 | 39,40 |
| Δ8去饱和酶 | 纤细眼虫 | 共同未决的美国专利申请11/166993 | 44,45 |
| Δ5去饱和酶 | 高山被孢霉 | GenBank获取号AF067654;U.S.6,075,183 | 6,7 |
| Δ5去饱和酶 | 球等鞭金藻 | WO 02/081668 A2 | 8,9 |
| Δ5去饱和酶 | 人 | GenBank获取号NP_037534 | 11,12 |
| C16/18延伸酶(“YE2”) | 解脂耶氏酵母 | -- | 61,62 |
| C16/18延伸酶(“ELO3”) | 高山被孢霉 | - | 53,54 |
| C16/18延伸酶(rELO2) | 褐家鼠 | GenBank获取号AB071986 | 50,51 |
| C14/16延伸酶(“YE1”) | 解脂耶氏酵母 | -- | 64,65 |
| Δ12去饱和酶 | 解脂耶氏酵母 | WO 2004/104167 | 23,24 |
| Δ12去饱和酶 | 深黄被孢霉 | GenBank获取号AF417245 | 25,26 |
| Δ12去饱和酶(Fm d12) | 串珠镰孢 | WO 2005/047485 | 27,28 |
| Δ12去饱和酶(An d12) | 构巢曲霉 | 构巢曲霉基因组项目中的重叠群1.15(支架1);AAG36933;WO 2005/047485 | 29,30 |
| Δ12去饱和酶 | 黄曲霉 | GenBank获取号AY280867(VERSION AY280867.1;gi:30721844);WO 2005/047485 | 31 |
| Δ12去饱和酶(Afd12p) | 烟曲霉 | 烟曲霉基因组项目中的反向AFA.133c 344248:345586(AfA5C5.001c);WO2005/047485 | 32 |
| Δ12去饱和酶(Mg d12) | Magnaporthegrisea | M.grisea基因组项目中的重叠群2.375中的基因座MG01985.1;WO 2005/047485 | 33,34 |
| Δ12去饱和酶(Nc d12) | 粗糙链孢霉 | GenBank获取号AABX01000374;WO2005/047485 | 35,36 |
| Δ12去饱和酶(Fg d12) | 禾谷镰孢 | 禾谷镰孢基因组项目中的重叠群1.233;WO 2005/047485 | 37,38 |
| Δ12去饱和酶(Mad12) | 高山被孢霉 | GenBank获取号AB020033 | 357,358 |
| Δ12去饱和酶(Skd12) | Saccharomyceskluyveri | GenBank获取号BAD08375 | 359 |
| Δ12去饱和酶(Kld12p) | 乳克鲁维氏酵母 | 来自“Yeast projectGenolevures”的乳克鲁维氏酵母数据库的反义Klla0B:35614..36861(m)的gnl|GLV|KLLA0B00473gORF(Center forBioinformatics,LaBRI,Talence Cedex,France) | 360,361 |
| Δ12去饱和酶(Cad12p) | 白色假丝酵母 | GenBank获取号EAK94955 | 362 |
| Δ12去饱和酶(Dhd12p) | 汉逊氏德巴利氏酵母CBS767 | GenBank获取号CAG90237 | 363 |
*注:烟曲霉基因组项目由Sanger Institute、University of Manchester的合作者和The Institute of Genome Research(TIGR)资助;构巢曲霉基因组项目由Center for Genome Research(CGR),Cambridge,MA资助;M.grisea基因组项目由CGR和International Rice Blast Genome Consortium资助;禾谷镰孢基因组项目由CGR和theInternational Gibberella zeae Genomics Consortium(IGGR)资助。
申请人进行了关于多种延伸酶的大量分析,以确定或证实在解脂耶氏酵母中表达时每种酶的底物特异性和/或底物选择性。例如,尽管两种解脂耶氏酵母延伸酶的编码序列都是公众可获得的,并且每种蛋白被注释为推定的长链脂肪酰基延伸酶或与其它脂肪酸延伸酶具有显著的同源性,但还没有确定这些酶的底物特异性。基于此处进行的分析,YE1被肯定地确定为优先利用C14脂肪酸作为底物制备C16脂肪酸的脂肪酸延伸酶(即C14/16 延伸酶),YE2确定为优先利用C16脂肪酸作为底物制备C18脂肪酸的脂肪酸延伸酶(即C16/18 延伸酶)。相关地,当鉴定出新的高山被孢霉ELO3基因时,该序列表征为与其它脂肪酸延伸酶同源。但是,需要进行脂质谱分析,以证实ELO3作为C16/18延伸酶的特异性
对于Δ12去饱和酶,申请人得到了出乎意料的发现,即串珠镰孢Δ12去饱和酶(由SEQ ID NO:27编码)以比天然解脂耶氏酵母Δ12去饱和酶更高的效率起作用,在解脂耶氏酵母中生产18:2(参见WO2005/047485)。具体地,确定解脂耶氏酵母中TEF启动子控制下的串珠镰孢Δ12去饱和酶的表达,以产生比以前通过表达TEF启动子控制下的编码解脂耶氏酵母Δ12去饱和酶的嵌合基因可获得的水平(59%的LA产物积累)更高水平的18:2(68%的LA产物积累)。这相应于底物转化百分比的差异(计算为([18:2+18:3]/[18:1+18:2+18:3])*100),分别是85%和74%)。在这些结果的基础上,相对于其它已知Δ12去饱和酶,作为对解脂耶氏酵母的高ARA生产菌株进行工程化的一种手段的本发明的真菌串珠镰孢Δ12去饱和酶的表达是优选的(但是,本领域技术人员可以预期,串珠镰孢Δ12去饱和酶的活性可以在解脂耶氏酵母中增强,然后进行例如密码子优化)。
尽管目前将串珠镰孢Δ12酶鉴定为优选的Δ12去饱和酶,最近已经鉴定了五种新的Δ12去饱和酶,它们可能能够在解脂耶氏酵母中以改进的效率起作用。具体地,Saccharomyces kluyveri Δ12去饱和酶(GenBank获取号BAD08375)描述于Watanabe et al.(Biosci.Biotech.Biocheml.68(3):721-727(2004)),而来自高山被孢霉的Δ12去饱和酶(GenBank获取号AB182163)描述于Sakuradani et al.(Eur.J.Biochem.261(3):812-820(1999))。由于随后用这两种序列鉴定S.Kluyveri和高山被孢霉Δ15去饱和酶(分别是GenBank获取号BAD11952和AB182163),这两对蛋白提供了与串珠镰孢、构巢曲霉、Magnaporthe grisea、粗糙链孢霉和禾谷镰孢的Δ12和Δ15去饱和酶相似的密切相关的真菌Δ12和Δ15去饱和酶的额外实例(参见WO 2005/047480和WO 2005/047485)。这一发现提供了额外的支持,证明申请人以前的假设,即,“成对的”真菌Δ12去饱和酶样序列很可能包含一种具有Δ15去饱和酶活性的蛋白和一种具有Δ12去饱和酶活性的蛋白。因此,本文中在乳克鲁维氏酵母、白色假丝酵母、汉逊氏德巴利氏酵母CBS767和烟曲霉中鉴定了相似的“成对的”Δ12去饱和酶样蛋白;并且,如预测的,比对的每对蛋白中的一个成员与以前鉴定的S.kluyveri Δ12去饱和酶(即乳克鲁维氏酵母gnl|GLV|KLLA0B00473g ORF,白色假丝酵母GenBank获取号EAK94955和汉逊氏德巴利氏酵母CBS767 GenBank获取号CAG90237)关系更密切,而另一个成员与S.kluyveri Δ15去饱和酶(乳克鲁维氏酵母GenBank获取号XM_451551,汉逊氏德巴利氏酵母CBS767 GenBank获取号CAG88182,白色假丝酵母GenBank获取号EAL03493)关系更密切。因此,基于这一分析,申请人鉴定了在此表示为SEQ ID Nos:358、359、361、362和363的去饱和酶为推定的真菌Δ12去饱和酶,它们在解脂耶氏酵母中的超量表达可以用于增加ω-6脂肪酸的生产。
在额外的实施方案中,申请人鉴定了容易区分具有Δ15去饱和酶活性与Δ12去饱和酶活性的真菌序列的手段。具体地,当分析包含Δ12去饱和酶(即Mad12,Skd12,Nc d12,Fm d12,Mg d12,An d12,Fg d12,Dhd12p,Kld12p,Cad12p,Afd12p,Mad15,Skd15,Nc d15,Fm d15,Mg d15,An d15,Fg d15,Dhd15p,Kld15p,Cad15p和Afd15p(参见上文的表))以及Δ15去饱和酶(即来自串珠镰孢、构巢曲霉、Magnaporthe grisea、粗糙链孢霉、禾谷镰孢、高山被孢霉、乳克鲁维氏酵母、白色假丝酵母、Saccharomyces kluyveri、汉逊氏德巴利氏酵母CBS767和烟曲霉)的氨基酸序列比对时,很明显的是所有真菌Δ15或Δ12去饱和酶分别在相应于串珠镰孢Δ15去饱和酶(WO2005/047479中的SEQ ID NO:2)的102位并且与高度保守的His Box1(“HECGH”;SEQ ID NO:373)仅仅间隔三个氨基酸残基的位置含有Ile或Val氨基酸残基(表6)。
表6
在真菌Δ12和Δ15去饱和酶的保守His Box 1附近的氨基酸比对
申请人概括出此位置的Ile和Val分别是真菌去饱和酶中Δ15和Δ12去饱和酶特异性的决定簇。更具体地,申请人认为在相应残基具有Ile(即,或基序IXXHECGH[SEQ ID NO:374])的任何真菌Δ12去饱和酶样蛋白将是Δ15去饱和酶,在相应残基具有Val(即,或基序VXXHECGH[SEQ ID NO:375])的任何真菌Δ12去饱和酶样蛋白将是Δ12去饱和酶。因此,这一单个的亮氨酸/缬氨酸将是未来鉴定和注释真菌去饱和酶时考虑的重要残基。此外,申请人认为在编码真菌Δ12去饱和酶样蛋白(如WO 2005/047479中描述为SEQ ID NO:2的串珠镰孢去饱和酶)的基因中,在此位置导致Ile改变为Val的突变将改变酶特异性,例如对Δ12去饱和的特异性;并且,相反,在此位置的导致Val改变为Ile的突变将改变酶特异性,例如对Δ15去饱和的特异性。
当然,在本发明的替代实施方案中,也可以用与SEQ ID NOs:2,5,7,9,12,18,21,24,26,28,30-32,34,36,38,40,45,51,54,62,65,358,359和361-363编码的去饱和酶和延伸酶基本相同的其它DNAs在解脂耶氏酵母中生产ARA。“基本相同”表示以优选性增加的顺序与选定的多肽或编码氨基酸序列的核酸序列表现出至少80%、90%或95%同源性的氨基酸序列或核酸序列。对于多肽,比较序列的长度通常是至少16个氨基酸,优选至少20个氨基酸,或最优选35个氨基酸。对于核酸,比较序列的长度通常是至少50个核苷酸,优选至少60个核苷酸,更优选至少75个核苷酸,最优选110个核苷酸。
同源性典型地是用序列分析软件测量的,其中术语“序列分析软件”表示可用于分析核苷酸或氨基酸序列的任何计算机算法或软件程序。“序列分析软件”可以是通过商业渠道获得的或者独立开发的。典型的序列分析软件包括,但不局限于:1.)GCG程序组(WisconsinPackage Version 9.0,Genetics Computer Group(GCG),Madison,WI);2.)BLASTP,BLASTN,BLASTX(Altschul et al.,J.Mol.Biol.215:403-410(1990));3.)DNASTAR(DNASTAR,Inc.,Madison,WI);和4.)采用了Smith-Waterman算法的FASTA程序(W.R.Pearson,Comput.Methods Genome Res.,[Proc.Int.Symp.](1994),Meeting Date 1992,111-20.Editor(s):Suhai,Sandor.Plenum:New York,NY)。在本申请的范围内应当理解的是,在将序列分析软件用于分析时,分析的结果是基于有关程序的“缺省值”的,除非另有说明。在本文中,“缺省值”表示在初次初始化时最初随所述软件加载的任何组的值或参数。一般地,所述计算机软件通过给多个取代、缺失和其它修饰赋予同源性程度而匹配相似的序列。
在更优选的实施方案中,利用了编码与SEQ ID NOs:2、9、12、18、21、40、45和51描述的序列基本相同的去饱和酶和延伸酶的密码子优化的基因。具体地,如本领域技术人员公知的,可以通过增加编码的mRNAs的翻译效率,增加异源基因的表达,所述翻译效率的增加是通过用选择的宿主微生物中进行最优基因表达的密码子替代天然基因中的密码子而实现的。因此,修饰要在外源宿主中表达的编码特定多肽的密码子的一部分通常是有用的,这样,修饰的多肽利用替代宿主优选的密码子,并且,宿主优选的密码子的利用可以显著增强编码多肽的外源基因的表达。
一般地,通过检验蛋白(优选以最大量表达的那些)中的密码子选择和确定哪些以最高的频率使用,可以在特定感兴趣的宿主物种中确定宿主优选的密码子。然后,可以用宿主物种中优选的密码子全部或部分地合成感兴趣的多肽(如去饱和酶、延伸酶、酰基转移酶)的编码序列。也可以合成DNA的全部(或部分),以除去转录的mRNA中存在的任何二级结构去稳定序列或区域。也可以合成DNA的全部(或部分),以将碱基组成改变成在需要的宿主细胞中更优选的组成。
此外,已经发现翻译起始密码子‘ATG’附近的核苷酸序列影响酵母细胞中的表达。如果需要的多肽在酵母中的表达不良,可以修饰外源基因的核苷酸序列,以引入有效的酵母翻译起始序列,获得最优的基因表达。为了在酵母中表达,可以通过对效率不佳地表达的基因的定点诱变而实现,所述诱变是通过将其与内源酵母基因,优选高表达的基因符合读框地融合而实现的。或者,如此处对于解脂耶氏酵母所证明的,可以确定宿主中的共有翻译起始序列,并且将该序列工程化为异源序列,以便它们在感兴趣的宿主中最优表达。
在本发明中,表5的一些去饱和酶和延伸酶基因为在解脂耶氏酵母中表达进行了密码子优化,该优化是基于上文描述的宿主优先性。这可能是通过首先确定解脂耶氏酵母密码子选择谱(参见WO04/101757)并且鉴定优选的密码子。然后,为了进一步优化解脂耶氏酵母中的基因表达,确定‘ATG’起始密码子附近的共有序列(即‘MAMMATGNHS’(SEQ ID NO:356),其中使用的核酸简并密码如下:M=A/C;S=C/G;H=A/C/T;和N=A/C/G/T)。下文的表7比较了在解脂耶氏酵母中表达时天然和密码子优化的基因的活性,并且提供了关于每种密码子优化的基因的细节。% Sub.Conv.是“底物转化百分比”的缩写,Codon-Opt.是“密码子优化的”的缩写。
表7
对于在解脂耶氏酵母中进行ARA生物合成最优选的密码子优化的去饱和酶和延伸酶
| 天然基因 | 天然基因%Sub.Conv. | Codon-Opt.基因中的修饰的总碱基数 | Codon-Opt.基因%Sub.Conv. | 参考文献 | Codon-Opt.SEQID NO |
| 高山被孢霉Δ6去饱和酶(GenBank获取号AF465281) | 30% | 1374bp的152bp(相应于144个密码子) | 42% | WO04/101753 | 3 |
| 高山被孢霉高亲和力C18/20延伸酶(GenBank获取号AX464731) | 30% | 957bp的94bp(相应于85个密码子) | 47% | WO04/101753 | 19 |
| 金黄色破囊壶菌C18/20延伸酶(“ELO2”) | 33% | 817bp的114bp(相应于108个密码子) | 46% | --- | 22 |
| 异丝水酶Δ17去饱和酶(US 2003/0196217A1) | 23% | 1077bp的127bp(相应于117个密码子) | 45% | 共同未决的美国专利申请No.10/840478 | 16 |
| 球等鞭金藻Δ9延伸酶 | --- | 789bp的126bp(相应于123个密码子) | 30% | --- | 41 |
| 纤细眼虫Δ8去饱和酶 | --- | 1263bp的207bp(相应于192个密码子) | 75% | 共同未决的美国专利申请No.11/166993 | 48 |
| 球等鞭金藻Δ5去饱和酶 | 7% | 1323bp的203bp(相应于193个密码子) | 32% | --- | 10 |
| 人Δ5去饱和酶(GenBank获取号NP_037534) | --- | 1335bp的227bp(相应于207个密码子) | 30% | --- | 13 |
| 褐家鼠C16/18延伸酶(GenBank获取号AB071986) | --- | 792bp的127bp(相应于125个密码子) | 43% | --- | 52 |
在本发明的额外的替代方案中,尽管其它一些DNAs不是与SEQID NOs:3、10、13、16、19、22、41、48和52所示的优选去饱和酶和延伸酶基本相同,也可以用于此处的目的。例如,可以用于根据本发明的教导导入解脂耶氏酵母中的编码Δ6去饱和酶多肽的DNA序列可以获自具有生产GLA或STA的能力的微生物。所述微生物包括例如属于以下属的微生物:被孢霉属、耳霉属、腐霉属、疫霉属、青霉属、紫球藻属、Coidosporium、毛霉属、镰孢属、曲霉属、红酵母属和虫霉属。在紫球藻属中,特别感兴趣的是P.Cruentum。在被孢霉属中,特别感兴趣的是长形被孢霉、微小被孢霉、喜湿被孢霉、拉曼被孢霉angulispora变种和高山被孢霉。在毛霉属中,特别感兴趣的是卷枝毛霉和爪哇毛霉。
或者,例如,一种相关的去饱和酶不是与高山被孢霉Δ6去饱和酶基本相同,但可以在距离分子的羧基末端的第6个碳的位置对脂肪酸分子去饱和,该去饱和酶也可以用于本发明中作为Δ6去饱和酶,认为该去饱和酶仍然可以有效将LA转化为GLA和/或将ALA转化为STA。因此,通过与此处公开的去饱和酶和延伸酶起基本相同的作用的能力,可以鉴定(或制备)相关的去饱和酶和延伸酶。
如上文所提示的,在另一实施方案中,本领域技术人员可以制备具有适于此处目的的Δ12去饱和酶和Δ6去饱和酶活性的融合蛋白。通过将Δ12去饱和酶和Δ6去饱和酶与连接的接头融合在一起,这是可能的。Δ12去饱和酶或Δ6去饱和酶可以位于融合蛋白的N末端部分。本领域技术人员很容易了解设计和合成合适的接头分子的方法,例如,接头可以是一段丙氨酸或赖氨酸,并且不影响融合酶的活性。
最后,本领域公知,文献中充分记载了合成序列和将序列连接在一起的方法。因此,可以用体外诱变和选择、定点诱变、化学诱变、“基因改组”方法或其它方法,获得天然存在的去饱和酶和/或延伸酶基因的突变。这使得能够在体内生产分别具有去饱和酶或延伸酶活性的多肽,其具有在宿主细胞中起作用的理想的物理和动力学参数(如更长的半衰期或生产需要的PUFA的更高速度)。
总之,尽管提出了编码适于在解脂耶氏酵母中生产ARA的PUFA生物合成途径酶的优选去饱和酶和延伸酶基因的序列,这些基因不意欲限制本发明。可以从多种来源(如具有合适去饱和酶或延伸酶活性的野生型、密码子优化的、合成的和/或突变的酶)分离适于此处目的的许多其它编码PUFA生物合成途径酶的基因。这些替代去饱和酶的特征可以在于以下能力:1.)在从分子的羧基末端开始计数的第8和9个碳原子之间对脂肪酸去饱和,并且催化EDA转化为DGLA(Δ8去饱和酶);2.)催化LA转化为GLA(Δ6去饱和酶);3.)催化DGLA转化为ARA(Δ5去饱和酶);4.)催化油酸转化为LA(Δ12去饱和酶);和/或5.)催化棕榈酸转化为棕榈油酸和/或硬脂酸转化为油酸(Δ9去饱和酶)。以相似的方式,用于此处目的的合适延伸酶不限于来自特定来源的那些。相反,具有适于此处目的的用途的酶的特征在于它们将脂肪酸碳链相对于延伸酶作用的底物延长2个碳,从而生产单或多不饱和脂肪酸的能力。更特别地,这些酶的特征在于以下能力:1.)将LA延伸为EDA(Δ9延伸酶);2.)延伸C18底物,以生产C20产物(C18/20延伸酶);3.)延伸C14底物,以生产C16产物(C14/16延伸酶);和/或4.)延伸C16底物,以生产C18产物(C16/18延伸酶)。重要的是注意一些延伸酶能够催化一些延伸酶反应,作为广泛底物特异性的结果。
酰基转移酶及其在TAG生物合成的终末步骤中的作用
酰基转移酶密切参与TAGs的生物合成。关于酵母中的TAG生物合成,包括参与的基因的细节和导致TAG合成的代谢中间体的两篇全面的短综述是:D.Sorger and G.Daum,Appl.Microbiol.Biotechnol.61:289-299(2003);和H.Müllner and G.Daum,ActaBiochimica Polonica,51(2):323-347(2004)。尽管这些综述的作者明确概括了真核动物酰基转移酶基因家族的不同种类(下文),但他们也承认TAG合成的调节方面和脂质颗粒中中性脂质的形成仍然还不清楚。
已经鉴定了四种真核动物酰基转移酶基因家族,它们参与酰基-CoA依赖性或非依赖性酯化反应,导致中性脂质合成:
(1)酰基-CoA:胆固醇酰基转移酶(ACAT)家族,EC 2.3.1.26(通常称作固醇酰基转移酶)。这个基因家族包括负责将酰基-CoA和固醇转化为CoA和固醇酯的酶。这个家族也包括参与TAG生物合成的终末步骤的DGAT1。
(2)卵磷脂:胆固醇酰基转移酶(LCAT)家族,EC 2.3.1.43。这个基因家族负责将磷脂酰胆碱和固醇转化为固醇酯和1-酰基甘油磷酸胆碱。这个家族也包括参与将酰基从磷脂的sn-2位置转移到1,2-二酰基甘油的sn-3位置,导致TAG生物合成的磷脂:二酰基甘油酰基转移酶(PDAT)。
(3)二酰基甘油酰基转移酶(DAG AT)家族,EC 2.3.1.20。这个基因家族(包括DGAT2)参与TAG生物合成的终末步骤。
(4)甘油-3-磷酸酰基转移酶和酰基-CoA溶血磷脂酸酰基转移酶(GPAT/LPAAT)家族。GPAT(E.C.2.3.1.15)蛋白负责TAG生物合成的第一步,而LPAAT(E.C.2.3.1.51)参与TAG生物合成的第二步。这个家族也包括催化磷脂和CoA之间的酰基交换的溶血磷脂酰胆碱酰基转移酶(LPCAT)。
这4个酰基转移酶基因家族一起代表用于中性脂质形成的重叠生物合成系统,并且似乎是差异调节、可变定位和不同底物特异性的结果(H.Müllner and G.Daum,supra)。在此将基于它们对解脂耶氏酵母中的代谢工程化的重要性对这4个基因家族进行讨论,以使得能够合成高于10-30%的ARA。
多种酰基转移酶的功能性
图4中概括了解脂耶氏酵母中很多所述酰基转移酶之间的相互作用。最初注意TAG生物合成的直接机理,该过程的第一步是一个分子的酰基-CoA经GPAT酯化为sn-甘油-3-磷酸,以生产溶血磷脂酸(LPA)(CoA作为副产物)。然后,通过第二个分子的酰基-CoA的酯化,溶血磷脂酸转化为磷脂酸(PA)(CoA作为副产物),该反应是LPAAT催化的。然后,磷脂酸磷脂酶负责从磷脂酸除去磷酸基团,产生1,2-二酰基甘油(DAG)。随后,通过DAG AT(如DGAT1、DGAT2或PDAT)将第三个脂肪酸加入DAG的sn-3位置,形成TAG。
以前,认为DGAT1是唯一专门参与TAG合成的酶,催化负责将酰基-CoA和DAG转为为TAG和CoA的反应,其中酰基-CoA基团转移到DAG,形成TAG。已知DGAT1与ACATs同源;但是,最近的研究鉴定了新的DAG AT酶家族,它们与ACAT基因家族无关。因此,现在的命名区分了与ACAT基因家族相关的DAG AT酶(DGAT1家族)和不相关的DAG AT酶(DGAT2家族)(Lardizabalet al.,J.Biol.Chem.276(42):38862-38869(2001))。在真核生物的所有主要门(真菌、植物、动物和基础真核生物)中鉴定了DGAT2家族的成员。
最近,Dahlqvist等人(Proc.Nat.Acad.Sci.(USA)97:6487-6492(2000))和Oelkers等人(J.Biol.Chem.275:15609-15612(2000))发现了在不存在酰基-CoA的条件下,通过酰基-CoA非依赖性机理,也可以发生TAG合成。具体地,PDAT除去磷脂酰胆碱底物sn-2位置的酰基,转移到DAG,以生产TAG。该酶与LCAT家族结构上相关;尽管PDAT的功能不像DGAT2那样充分表征,已经推测PDAT在从一些油籽植物中的磷脂除去“不常见”脂肪酸中起主要作用(Banas,A.et al.,Biochem.Soc.Trans.28(6):703-705(2000))。
对于啤酒糖酵母中的TAG合成,描述了三种途径(Sandager,L.et al.,J.Biol.Chem.277(8):6478-6482(2002))。首先,主要通过DGAT2活性(由DGA1基因编码),从DAG和酰基-CoAs合成TAGs。但是,更近年来,也鉴定了PDAT(由LRO1基因编码)。最后,已知两种酰基-CoA:固醇-酰基转移酶(由ARE1和ARE2基因编码)利用酰基-CoAs和固醇生产固醇酯(和低量TAGs;参见Sandager et al.,Biochem.Soc.Trans.28(6):700-702(2000))。PDAT和DGAT2一起负责啤酒糖酵母中的大约95%的油生物合成。
基于一些公众可得到的编码DGAT1s、DGAT2s、PDATs和ARE2s的序列(参见下文),申请人在解脂耶氏酵母中分离和表征了编码DGAT1(SEQ ID NO:81)、DGAT2(SEQ ID NOs:89、91和93[其中SEQ ID NO:89含有至少两个额外的嵌套ORFs,如SEQ ID NOs:91和93中提供的;SEQ ID NO:93编码的ORF与其它已知DGAT酶具有高度的相似程度,SEQ ID NO:93中的破坏消除了天然基因的DGAT功能,从而证明了SEQ ID NO:94的多肽具有DGAT功能])、PDAT(SEQ ID NO:76)和ARE2(SEQ ID NO:78)的基因。但是,与在啤酒糖酵母中开发的模型(其中PDAT和DGAT2负责大约95%的油生物合成)相反,发现解脂耶氏酵母的PDAT、DGAT2和DGAT1负责最高达大约95%的油生物合成(而ARE2可能额外是油生物合成的次要决定因素)。
功能对于解脂耶氏酵母的TAG级分中的ARA聚集重要的最后一种酰基转移酶是LPCAT。如图4所示,该酶(EC 2.3.1.23)被假定为负责sn-磷脂酰胆碱的sn-2位置上的双向酰基交换,以增强ω-6和ω-3PUFA生物合成。这种假定基于以下研究:(1)Stymne S.and A.K.Stobart(Biochem J.223(2):305-14(1984)),他们假定LPCAT影响酰基-CoA集合和磷脂酰胆碱(PC)集合之间的交换;(2)Domergue,F.et al.(J.Bio.Chem 278:35115(2003)),它们提示PC的sn-2位置上GLA的积累和不能在酵母中有效合成ARA,是参与酰基-CoA集合中发生的PUFA生物合成的延伸步骤的结果,而Δ5和Δ6去饱和步骤主要发生在PC的sn-2位置;(3)Abbadi,A.et al.(The Plant Cell,16:2734-2748(2004)),他们基于对转基因油籽植物中PUFA积累的约束的分析,提示LPCAT在Δ6去饱和酶/Δ6延伸酶途径的成功重建中起关键作用;和(4)WO 2004/076617 A2(Renz,A.et al.),他们提供了编码来自秀丽新杆线虫的LPCAT(T06E8.1)的基因,该LPCAT显著改进了啤酒糖酵母中遗传学导入的Δ6去饱和酶/Δ6延伸酶途径中的延伸效率。本发明人概括出LPCAT使得能够在磷脂和酰基-CoA集合之间有效和连续交换新合成的脂肪酸,因为去饱和酶催化脂质偶联的脂肪酸(sn-2酰基PC)中双键的导入,而延伸酶专门催化CoA酯化的脂肪酸(酰基-CoAs)的延伸。
用于ARA合成的异源酰基转移酶基因的选择
由于解脂耶氏酵母中天然生产的PUFAs限于18:2脂肪酸(较不常见的是18:3脂肪酸),很可能宿主生物中编码GPAT、LPAAT(即LPAAT1或LPAAT2)、DGAT1、DGAT2、PDAT和LPCAT的天然基因难以有效合成包含18:3和更长(如ARA)的脂肪酸的TAGs。因此,在某些情况下,异源(或“外源”)酰基转移酶比天然酶优选。
在多种生物中鉴定了许多酰基转移酶基因,并且公开于公共和专利文献中。例如,以下GenBank获取号表示公众可得到的、用于脂质生物合成中的酰基转移酶基因:CQ891256,AY441057,AY360170,AY318749,AY093169,AJ422054,AJ311354,AF251795,Y00771,M77003(GPATs);Q93841,Q22267,Q99943,O15120,Q9NRZ7,Q9NRZ5,Q9NUQ2,O35083,Q9D1E8,Q924S1,Q59188,Q42670,P26647,P44848,Q9ZJN8,O25903Q42868,Q42870,P26974,P33333,Q9XFW4,CQ891252,CQ891250,CQ891260,CQ891258,CQ891248,CQ891245,CQ891241,CQ891238,CQ891254,CQ891235(LPAATs);AY445635,BC003717,NM_010046,NM_053437,NM_174693,AY116586,AY327327,AY327326,AF298815和AF164434(DGAT1s);以及NC_001147[基因座NP_014888],NM_012079,NM_127503,AF051849,AJ238008,NM_026384,NM_010046,AB057816,AY093657,AB062762,AF221132,AF391089,AF391090,AF129003,AF251794 and AF164434(DGAT2s);P40345,O94680,NP_596330,NP_190069和AB006704[gi:2351069](PDATs)。类似地,专利文献提供了很多参与TAG生产的基因的额外DNA序列(和/或关于上文的一些基因及其分离方法的细节)[例如U.S.5,210,189,WO 2003/025165(GPATs);EP1144649 A2,EP1131438,U.S.5,968,791,U.S.6,093,568,WO 2000/049156和WO 2004/087902(LPAATs);U.S.6,100,077,U.S.6,552,250,U.S.6,344,548,US2004/0088759A1和US 20040078836A1(DGAT1s);US 2003/124126,WO 2001/034814,US 2003/115632,US 2003/0028923和US2004/0107459(DGAT2s);WO 2000/060095(PDATs);以及WO2004/076617 A2(LPCATs)。
上文的实例不是限制性的,来源于不同来源的许多其它编码DGAT1、DGAT2、PDAT、GPAT、LPCAT和LPAAT的基因也适于导入解脂耶氏酵母中。例如,申请人从高山被孢霉(SEQ ID NOs:83和84)、粗糙链孢霉(SEQ ID NO:85)、玉蜀黍赤霉PH-1(SEQID NO:86)、Magnaporthe grisea(SEQ ID NO:87)以及构巢曲霉(SEQ ID NO:88)鉴定了新的DGAT1;并且从高山被孢霉鉴定了新的DGAT2(SEQ ID NOs:95和96)、GPAT(SEQ ID NOs:97和98)、LPAAT1(SEQ ID NOs:67和68)以及LPAAT2(SEQ IDNOs:69和70)。
用于ARA合成的优选酰基转移酶基因
尽管可以适于在解脂耶氏酵母中表达的酰基转移酶有广泛的选择,但是,在本发明的优选实施方案中,DGAT1、DGAT2、PDAT、GPAT、LPAAT和LPCAT选自生产大量较长链ω-6(如ARA)和/或ω-3(如EPA、DHA)PUFAs的生物。因此,特别优选的是以下酶(或其衍生物):
表8
用于在解脂耶氏酵母的高ARA生产菌株中表达的优选异源酰基转移酶
| ORF | 生物 | 参考文献 | SEQ ID NOs |
| DGAT1 | 高山被孢霉 | 共同未决的美国专利申请11/024544 | 83,84 |
| DGAT2 | 高山被孢霉 | 共同未决的美国专利申请11/024545 | 95,96 |
| GPAT | 高山被孢霉 | - | 97,98 |
| LPAAT1 | 高山被孢霉 | -- | 67,68 |
| LPAAT2 | 高山被孢霉 | 共同未决的美国专利申请60/689031 | 69,70 |
| LPCAT | 秀丽新杆线虫 | 克隆T06E8.1;WO 2004/076617 A2 | 80 |
尽管不意欲限制本发明,选择高山被孢霉作为异源酰基转移酶的优选来源,因为该天然生物能够合成浓度高于总脂肪酸(TFAs)的50%的ARA。以相似的方式,秀丽新杆线虫能够生产最高达其TFAs的20-30%的EPA。
当然,在本发明的替代实施方案中,也可以将与SEQ ID NOs:67,68,69,70,80,83,84,95,96,97和98编码的酰基转移酶基本相同的其它DNAs用于在解脂耶氏酵母中进行异源表达,以促进TAG级分中ARA的生产和积累。在更优选的实施方案中,利用了编码与SEQ ID NOs:67-70,80,83,84和95-98基本相同的酰基转移酶的密码子优化的基因。
用于表达外源基因的通用表达系统、盒、载体和转化
含有指导外源蛋白的高水平表达的调节序列,如导致高水平生产ARA的调节序列的微生物表达系统和表达载体是本领域技术人员公知的。上述任何载体都可以用于构建编码优选的去饱和酶、延伸酶和酰基转移酶的嵌合基因。然后可以用标准转化方法将这些嵌合基因导入解脂耶氏酵母中,以提供编码的酶的高水平表达。
用于转化宿主细胞的载体或DNA盒是本领域公知的。构建体中存在的序列的特定选择依赖于需要的表达产物、宿主细胞的性质和提出的分离转化的细胞与未转化细胞的方法。但是,典型地,载体或盒含有指导相关基因转录和翻译的序列、选择标记和使得能够进行自主复制和染色体整合的序列。合适的载体包含控制转录起始的基因的5’的区域(如启动子)和控制转录终止的DNA片段3’的区域(如终止子)。当两个控制区都来自转化的宿主细胞的基因时是最优选的,但是,应该理解,所述控制区不一定必须来自选作生产宿主的特定物种的天然基因。
当从分开的复制载体表达两种或更多种基因时,理想的是每种载体具有不同的选择方法,并且应该缺乏与其它构建体的同源性,以维持稳定的表达并且防止构建体之间元件的重新分配。调节区的明智选择、选择方法和导入的构建体的增殖方法可以实验确定,由此以必须的水平表达所有导入的基因,并且提供需要的产物的合成。
可以通过任何标准技术将包含感兴趣的基因的构建体导入宿主细胞。这些技术包括转化(如醋酸锂转化[Methods in Enzymology,194:186-187(1991)])、原生质体融合、bolistic impact、电穿孔、微注射或任何其它将感兴趣的基因导入宿主细胞的方法。适用于解脂耶氏酵母的更特别的教导包括美国专利No.4,880,741和No.5,071,764以及Chen,D.C.et al.(Appl Microbiol Biotechnol.48(2):232-235(1997))。
为了方便起见,业已通过任何方法操作以摄入DNA序列(例如,表达盒)的宿主细胞在本文中被称作“转化的”或“重组的”。转化的宿主具有至少一个拷贝的表达构建体,并且可以具有两个或两个以上拷贝,这取决于所述基因是整合在所述基因组上、扩增的、或者存在于具有多个拷贝的染色体外元件上。可以通过多种选择技术鉴定转化的宿主细胞,如WO 2004/101757和WO 2005/003310中的描述。
此处使用的优选选择方法是对卡那霉素、潮霉素和氨基糖苷G418的抗性以及在缺少尿嘧啶、亮氨酸、赖氨酸、色氨酸或组氨酸的培养基上生长的能力。在替代的实施方案中,为了选择酵母Ura-转化体,使用5’-氟乳清酸(5-氟尿嘧啶-6-羧酸一水合物;“5-FOA”)。该化合物对具有起作用的编码乳清苷5’-一磷酸脱羧酶(OMP脱羧酶)的URA3基因的酵母细胞具有毒性;因此,基于该毒性,5-FOA对于Ura-突变酵母株的选择和鉴定特别有用(Bartel,P.L.and Fields,S.,Yeast 2-Hybrid System,Oxford University:New York,v.7,pp109-147,1997)。
此处用到的一种替代的优选选择方法依赖于解脂耶氏酵母的显性、非抗生素标记,该标记基于磺酰脲抗性。该技术通常也适用于其它可能是单倍、二倍、非整倍或杂合的工业酵母株。预计它克服了对于开发工业酵母株的遗传转化系统的两个主要限制,这两个限制是:(1)几乎不存在天然营养缺陷株,并且由于这些株的倍性阻碍了自发或诱导的营养缺陷突变体的分离;和(2)抗生素抗性标记的利用可能限制了菌株的商业用途,这是因为对携带抗生素抗性基因的遗传修饰的生物的释放的限制。尽管Puig等人(J.Agric.Food Chem.46:1689-1693(1998))开发了基于靶菌株的基因工程的、克服这些限制的方法以使其对于尿苷是营养缺陷的,并且随后利用了URA3标记以诱导感兴趣的形状,但该策略仍然被认为对于常规工作来说过于费力。
此处公开的用于转化解脂耶氏酵母的新的磺酰脲抗性选择标记不仅仅依赖于外源基因,也依赖于突变的天然基因。因此,它不需要营养缺陷,也不导致营养缺陷,并且允许转化野生型菌株。更具体地,标记基因(SEQ ID NO:280)是一种天然的乙酰羟酸合酶(AHAS或乙酰乳酸合酶;E.C.4.1.3.18),其具有赋予磺酰脲除草剂抗性的单个氨基酸改变(W497L)。AHAS是支链氨基酸的生物合成途径中的第一种常见酶,它是磺酰脲和咪唑烷酮除草剂的靶。在啤酒糖酵母中报道了W497L突变(Falco,S.C.,et al.,Dev.Ind.Microbiol.30:187-194(1989);Duggleby,R.G.,et.al.Eur.J.Biochem.270:2895(2003))。初步的测试确定了耶氏酵母细胞对于除草剂不是天然具有抗性的,这是因为1.)对除草剂的摄取不良或不摄取;2.)存在天然的除草剂抗性形式的AHAS;和/或3.)利用了除草剂灭活机制。因此,这使得能够合成和利用突变的AHAS基因(SEQ ID NO:280)作为选择转化体的手段。
一种再循环选择标记的额外方法依赖于位点特异性重组酶系统。简言之,该位点特异性重组系统由两个元件组成:(1)具有特征性DNA序列[如LoxP]的重组位点;和(2)当两个或多个重组位点在相同DNA分子[如Cre]上以特定间隔按相同方向定位时特异性结合于DNA序列并且催化DNA序列之间的重组(即切割)的重组酶。该方法具有作为选择工具的用途,因为它可以“再循环”一对优选的选择标记,用于它们在多次连续转化中的用途。
更具体地,建立了整合构建体,其包含需要插入宿主基因组的靶基因(如去饱和酶、延伸酶、酰基转移酶)以及侧翼于重组位点的第一选择标记(如Ura3、潮霉素磷酸转移酶[HPT])。在转化体的转化和选择后,通过导入携带第二选择标记(如磺酰脲抗性[AHAS])和适于识别导入基因组中的位点特异性重组位点的重组酶的复制质粒,从染色体切下第一选择标记。选择携带第二标记的转化体和证明第一选择标记从宿主基因组切下后,在无选择的条件下从宿主收获复制的质粒。这产生了既不存在第一标记也不存在第二标记的转化体,因此,经过消除的菌株可以用于另一轮转化。本领域技术人员可以意识到,该方法不限于用于本发明的特定选择标记或位点特异性重组系统。
外源基因在解脂耶氏酵母中的超量表达
正如技术人员所了解的,仅仅将基因(如去饱和酶)插入克隆载体,不能确保它能够在需要的水平上成功地表达。可能需要操纵多种不同的遗传元件,这些遗传元件控制转录,翻译,蛋白稳定性,含氧极限,以及从宿主细胞的分泌。更具体地讲,可以通过改变以下方面控制基因表达:相关转录启动子和终止子序列的性质;克隆基因的拷贝数量;基因是质粒携带的或者是整合到宿主细胞的基因组上;合成的外源蛋白的最终细胞定位;宿主生物的翻译效率;克隆的基因蛋白在宿主细胞中的内在稳定性;和克隆的基因内的密码子选择,这样它的频率接近宿主细胞的优选密码子使用频率。下文将讨论这些超量表达方法中的一些,并且用于本发明中作为在解脂耶氏酵母中超量表达例如去饱和酶、延伸酶和酰基转移酶的手段。
通过使用较强的启动子(如调节型或组成型)导致表达增加、通过从mRNA或编码的蛋白除去/缺失去稳定序列、或通过给mRNA增加稳定序列(U.S.4,910,141),可以在转录水平增加需要的基因的表达。
可用于在需要的宿主细胞中驱动去饱和酶、延伸酶和酰基转移酶基因表达的起始控制区或启动子是多种多样的,并且为本领域技术人员所熟知。实际上,能够指导这些基因在解脂耶氏酵母中表达的任何启动子都适合本发明。在宿主细胞中的表达能够以瞬时或稳定的方式进行。瞬时表达可以通过诱导可操作地连接在感兴趣的基因上的可调节启动子的活性而实现;或者稳定表达可以通过使用可操作地与感兴趣的基因连接的组成型启动子而实现。例如,当宿主细胞是酵母时,提供能够在酵母细胞中起作用的转录和翻译区,特别是来自宿主物种的那些。例如,转录起始调节区可以从以下渠道获得:1.)糖酵解途径中的基因,如醇脱氢酶、甘油醛-3-磷酸脱氢酶、磷酸甘油变位酶、果糖二磷酸醛缩酶、磷酸葡萄糖异构酶、磷酸甘油酸激酶、甘油-3-磷酸O-酰基转移酶等;或2.)可调节的基因,如酸性磷酸酶、乳糖酶、金属硫蛋白、葡糖淀粉酶、翻译延伸因子EF1-α(TEF)蛋白(U.S.6,265,185)、核糖体蛋白S7(U.S.6,265,185)等。可以使用多种调节序列中的任意一种,这取决于是需要组成型转录还是诱导型转录、启动子在表达感兴趣的ORF方面的效率、构建的方便性等。上面提供的实例不意欲限制本发明。
本领域技术人员了解,可以用多种方法比较多种启动子的活性。这种类型的比较可以用于促进确定用于将来的应用中的每种启动子的强度,其中一组启动子对于构建用于生产ω-6和ω-3脂肪酸的嵌合基因是必要的。因此,可能有用的是基于报道基因表达(即编码β-葡萄醛酸酶(GUS)的大肠杆菌基因)而对启动子活性进行间接定量的方法。在替代的实施方案中,有时用更定量性的方法对启动子活性进行定量可能是有用的。一种合适的方法是使用实时PCR(对于实时PCR应用的一般综述,参见Ginzinger,D.J.,Experimental Hematology,30:503-512(2002))。实时PCR基于荧光报道分子的检测和定量。信号的增加与反应中PCR产物的量成正比。通过记录每个循环的荧光发射量,可以在指数期监测PCR反应,其中PCR产物量的第一次显著增加与靶模板的起始量相关。存在两种用于定量检测扩增子的通用方法:(1)使用荧光探针;或(2)使用DNA结合剂(如SYBR-green I、溴化乙锭)。对于相对基因表达比较,有必要使用内源对照作为内部参照(如染色体编码的16S rRNA基因),从而对加入每个实时PCR反应中的总DNA量的差异进行标准化。用于实时PCR的具体方法在本领域有充分记载。参见例如the Real Time PCR SpecialIssue(Methods,25(4):383-481(2001))。
在实时PCR反应后,用记录的荧光强度对模板的量进行定量,这是通过采用:1.)绝对标准物方法(其中使用已知量的标准物,如体外翻译的RNA(cRNA));2.)相对标准物方法(其中在每一轮的测定设计中都包括已知量的靶核酸);或3.)用于对基因表达进行相对定量的比较性CT法(ΔΔCT)(其中将靶序列的相对量与选择的任何参照值进行比较,结果相对于参照值给出)。比较性CT法需要确定靶和标准化物的CT值之间的差异(ΔCT),其中:ΔCT=CT(靶)-CT(标准化物)。对要定量的每个样品计算该值,并且必须选择一种样品作为进行每次比较的参照。比较性ΔΔCT计算包括发现每个样品的ΔCT和基线的ΔCT之间的差异,然后根据公式2-ΔΔCT将这些值转化为绝对值。
尽管可以适于在解脂耶氏酵母中表达的启动子有广泛选择,但是,在本发明的优选实施方案中,启动子选自表9所示的那些(或其衍生物)。
表9
优选用于在解脂耶氏酵母中超量表达的天然启动子
| 启动子名称 | 位置* | 天然基因 | 活性“等级” | 参考文献 | SEQID NO |
| TEF | --- | 翻译延伸因子EF1-α | 1 | U.S.6,265,185(Muller etal.);GenBank获取号AF054508 | 166 |
| GPD | -968bp至+3bp | 甘油醛-3-磷酸-脱氢酶 | 2 | WO 2005/003310 | 158 |
| GPM | -875bp至+3bp | 磷酸甘油变位酶 | 1 | WO 2005/003310 | 160 |
| FBA | -1001bp至-1bp | 果糖-二磷酸醛缩酶 | 4 | WO 2005/049805 | 161 |
| FBAIN | -804bp至+169bp(包括102bp内含子[+64至+165]) | 果糖-二磷酸醛缩酶 | 7 | WO 2005/049805 | 162 |
| FBAINm | -804bp至+169bp具有修饰*** | 果糖-二磷酸醛缩酶 | 5 | WO 2005/049805 | 163 |
| GPDIN | -973bp至+201bp(包括146bp内含子[+49至+194]) | 甘油醛-3-磷酸-脱氢酶 | 3 | 共同未决的美国专利申请No.11/183664 | 159 |
| GPAT | -1130至+3bp | 甘油-3-磷酸O-酰基转移酶 | 5 | 共同未决的美国专利申请No.11/225354 | 164 |
| YAT1 | -778至-1bp | 铵转运蛋白酶 | 6 | 共同未决的美国专利申请No.11/185301 | 165 |
| EXP1 | -1000至-1bp | 输出蛋白 | 6 | ---- | 364 |
*位置是相对于天然基因,其中‘ATG’翻译起始密码子的‘A’位置表示为+1。
***FBAINm启动子是修饰形式的FBAIN启动子,其中FBAINm在ATG翻译起始密码子和FBAIN启动子的内含子之间具有52bp缺失(从而仅仅包括N-末端的22个氨基酸)并且具有内含子后的新的翻译共有序列基序。此外,尽管当与要表达的基因的编码区融合时FBAIN启动子产生融合蛋白,但FBAINm启动子不产生这样的融合蛋白。
GPM的活性大约与TEF相同,而GPD、FBA、FBAIN、FBAINm、GPDIN、GPAT、YAT1和EXP1的活性都大于TEF(活性是在标题为“活性等级”的列中以相对方式定量的,其中“1”相应于具有最低活性的启动子,而“7”相应于具有最高活性的启动子)。这种定量是基于比较性研究,其中将每种启动子用于建立嵌合基因,该嵌合基因具有编码作为报道分子的β-葡萄醛酸酶(GUS)的大肠杆菌基因(Jefferson,R.A.Nature.14;342:837-838(1989))和耶氏酵母Xpr基因的3’区的约100bp。通过组织化学和/或荧光测量分析(Jefferson,R.A.Plant Mol.Biol.Reporter 5:387-405(1987))和/或采用实时PCR,测量每个表达的构建体中的GUS活性。
YAT1启动子是独特的,因为申请人将其表征为在耶氏酵母中鉴定的第一种在含油条件(即氮限制)下可诱导的启动子。具体地,尽管YAT1启动子在含氮(如最多约0.5%硫酸铵)的培养基中有活性,但当宿主细胞在氮限制条件下(如在含有非常低水平的铵或缺乏铵的培养基中)生长时,启动子的活性增加。因此,优选的培养基将是含有少于约0.1%硫酸铵或其它合适的铵盐的培养基。在一种更优选的实施方案中,当宿主细胞在具有高碳氮比(即C:N)的培养基,如含有大约8-12%葡萄糖和大约0.1%或更少硫酸铵的高葡萄糖培养基(HGM)中生长时,YAT1启动子被诱导。这些条件也足够诱导那些含油酵母(如解脂耶氏酵母)中的含油性。基于细胞提取物的GUS活性,当细胞从基本培养基转移到HGM中并生长24小时后,YAT1启动子的活性增加约73倍;在HGM中生长120小时后,活性某种程度降低,当仍然比含氮的基本培养基中的活性高25倍(实施例1)。
当然,在本发明的替代实施方案中,其它衍生自上文表9中描述的启动子区的其它启动子也可以用于在解脂耶氏酵母中异源表达,以促进TAG级分中ARA的生产和积累。具体地,上文描述的任何启动子的长度的改变可以导致具有相同活性的突变启动子,因为还没有完全确定这些调节序列的精确边界。在替代实施方案中,位于FBAIN和GPDIN启动子的内含子内的增强子可以用于建立嵌合启动子,其相对于天然耶氏酵母启动子具有增加的活性(例如,当驱动GUS报道基因与耶氏酵母Xpr基因的3’区的约100bp一起表达时,嵌合GPM::FBAIN和GPM::GPDIN启动子(SEQ ID NOs:182和183)相对于单独的GPM启动子具有增加的活性)。
终止区可源于基因的3’区(起始区是从该区获得的)或者从不同的基因获得。大量终止区是已知的,并且能在多种宿主(当在与产生它们的相同和不同的属和种中使用时)中发挥令人满意的作用。终止区通常在更大程度上是根据方便性选择的,而不是因为任何特殊特性。优选的是,终止区源于酵母基因,特别是糖酵母属、裂殖酵母属、假丝酵母属、耶氏酵母属或克鲁维酵母属。同样已知编码γ-干扰素和α-2干扰素的哺乳动物基因的3’-区在酵母中起作用。终止控制区还可以来自优选宿主天然具有的各种基因。终止位点可以任选是必需的;不过,如果具有,则是最优选的。尽管不意欲限制,本发明公开中使用的终止区包括:解脂耶氏酵母胞外蛋白酶的3’区的约100bp(XPR;GenBank获取号M17741);酰基-coA氧化酶(Aco3:GenBank获取号AJ001301和No.CAA04661;Pox3:GenBank获取号XP_503244)终止子;Pex20(GenBank获取号AF054613)终止子;Pex16(GenBank获取号U75433)终止子;Lip1(GenBank获取号Z50020)终止子;Lip2(GenBank获取号AJ012632)终止子;和3-氧酰基-coA硫解酶(OCT;GenBank获取号X69988)终止子。
可以将上文描述的额外拷贝(即一个拷贝以上)的去饱和酶、延伸酶和/或酰基转移酶基因导入解脂耶氏酵母,从而增加ARA生产和积累。具体地,可以在单个表达构建体中克隆额外拷贝的基因;和/或可以通过增加质粒拷贝数或通过将克隆的基因多次整合到基因组中将额外拷贝的克隆的基因导入宿主细胞(参见下文)。例如,在一种实施方案中,对解脂耶氏酵母的菌株(即菌株Y2214)进行工程化,以通过将嵌合基因导入和整合到耶氏酵母基因组中而生产大于14%的ARA,所述嵌合基因包含:5个拷贝的Δ9延伸酶、3个拷贝的Δ8去饱和酶、4个拷贝的Δ5去饱和酶、1个拷贝的Δ12去饱和酶和1个拷贝的C16/18延伸酶。类似地,在一种替代实施方案中,对解脂耶氏酵母的Y2047株进行工程化,以通过将嵌合基因导入和整合到耶氏酵母基因组中而生产大于11%的ARA,所述嵌合基因包含:1个拷贝的Δ6去饱和酶、2个拷贝的C18/20延伸酶、3个拷贝的Δ5去饱和酶和1个拷贝的Δ12去饱和酶。
一般地,一旦获得了适合在含油酵母中表达的DNA(如包含启动子、ORF和终止子的嵌合基因),将它放入能够在宿主细胞中自主复制的质粒载体上,或者将它直接整合到宿主细胞的基因组中。表达盒的整合可以在宿主基因组中随机地进行,或者可以通过使用含有与宿主基因组具有足够同源性以靶定宿主基因座内的重组的区域的构建体靶定。尽管本发明不完全基于此,当所述构建体被靶定于内源基因座时,转录和翻译调节区的全部或某些可以通过内源基因座提供。
在本发明中,在解脂耶氏酵母中表达基因的优选方法是通过将线性DNA整合到宿主的基因组中;并且,当需要基因的高水平表达时,整合到基因组中的多个位置是特别有用的。为此,需要鉴定基因组中以多个拷贝存在的序列。
Schmid-Berger等(J.Bact.176(9):2477-2482(1994))发现了解脂耶氏酵母中的第一个反转录转座子样元件Ylt1。该反转录转座子的特征在于存在长的末端重复序列(LTRs;每一个的长度是大约700bp),其称作ζ区。Ylt1和单独的ζ元件分别以至少35个拷贝/基因组和50-60个拷贝/基因组,以分散的方式存在于基因组中;确定这两个元件都作为同源重组位点起作用。此外,Juretzek等(Yeast18:97-113(2001))的研究证明了通过将质粒靶定到酵母基因组的重复区中(用两端都具有LTR ζ区的线性DNA),与用低拷贝质粒转化体获得的表达相比,可以显著增加基因表达。因此,定位于ζ的整合作为确保将多个质粒DNA整合到解脂耶氏酵母中的手段是理想的,从而允许高水平基因表达。然而,不幸的是,并不是解脂耶氏酵母的所有菌株都具有ζ区(如鉴定为ATCC #20362的菌株)。当菌株缺乏所述区域时,也可以将包含表达盒的质粒DNA整合到替代的基因座中,以达到表达盒的需要的拷贝数。例如,优选的替代基因座包括:Ura3基因座(GenBank获取号AJ306421)、Leu2基因座(GenBank获取号AF260230)、Lys5基因(GenBank获取号M34929)、Aco2基因座(GenBank获取号AJ001300)、Pox3基因座(Pox3:GenBank获取号XP_503244;或Aco3:GenBank获取号AJ001301)、Δ12去饱和酶基因基因座(SEQ ID NO:23)、Lip1基因座(GenBank获取号Z50020)和/或Lip2基因座(GenBank获取号AJ012632)。
有利的是,Ura3基因可以与5-FOA选择组合,重复使用(参见上文)。更具体地,可以首先敲除天然Ura3基因,生产具有Ura表型的菌株,其中选择是基于5-FOA抗性发生的。然后,可以将一簇多个嵌合基因和新的Ura3基因整合到耶氏酵母基因组的不同基因座中,从而产生具有Ura+表型的新菌株。当敲除导入的Ura3基因时,随后的整合将产生新的Ura3-菌株(再次用5-FOA选择进行鉴定)。这样,可以将Ura3基因(与5-FOA选择组合)用作多轮转化中的选择标记,从而容易地使基因修饰以容易的方式整合到耶氏酵母基因组中。
对于一些应用,将本发明的蛋白导向于不同的细胞腔室(如酰基-CoA集合与磷脂酰胆碱集合)将是有用的。为了此处描述的目的,ARA可以以游离脂肪酸或酯化形式如酰基甘油、磷脂、硫脂或糖脂形式存在。预计可以将上文描述的编码允许ARA生物合成的多肽的嵌合基因进一步工程化,以包括合适的细胞内靶定序列。
Juretzek等(Yeast,18:97-113(2001))注意到解脂耶氏酵母中整合的质粒拷贝数的稳定性依赖于各个转化体,即受体株和使用的靶定平台。因此,技术人员将理解,必须筛选多个转化体,以获得展示需要的表达水平和模式的菌株。所述筛选可以通过DNA印迹的Southern分析(Southern,J.Mol.Biol.98:503(1975))、mRNA表达的Northern分析(Kroczek,J.Chromatogr.Biomed.Appl.,618(1-2):133-145(1993))、蛋白表达的Western分析、表型分析或PUFA产物的GC分析实现。
总之,可以将上文描述的每一种方法用于增加解脂耶氏酵母中特定基因产物(如去饱和酶、延伸酶、酰基转移酶)的表达;并且,生物技术领域的技术人员能够容易地选择允许ARA的高水平生产的合适方法组合。
用于增加ARA生产的途径工程化
尽管上文描述的方法可以用于上调各个异源基因的表达,但增加解脂耶氏酵母中的ARA生产的挑战更复杂,并且可能需要多个代谢途径的协调操作。首先将解决PUFA生物合成途径的操作,然后是TAG生物合成途径和TAG降解途径的理想操作。
如上文描述的,生产占总油级分的5%以上的ARA,或更优选占总油级分的10%以上的ARA,或甚至更优选占总油级分的15-20%以上的ARA,或最优选占总油级分的25-30%以上的ARA的解脂耶氏酵母株的构建至少需要以下用于表达Δ6去饱和酶/Δ6延伸酶途径的基因:Δ6去饱和酶、C18/20延伸酶和Δ5去饱和酶;或至少需要以下用于表达Δ9延伸酶/Δ8去饱和酶途径的基因:Δ9延伸酶、Δ8去饱和酶和Δ5去饱和酶。但是,在任意一种实施方案中,可能需要在宿主菌株中额外包括Δ9去饱和酶、Δ12去饱和酶、C14/16延伸酶和/或C16/18延伸酶。
在一些情况下,用串珠镰孢Δ12去饱和酶代替天然解脂耶氏酵母Δ12去饱和酶可能证明是有利的,因为串珠镰孢Δ12去饱和酶增加底物转化百分比(WO 2005/047485)。更具体地,尽管这两种Δ12去饱和酶都催化油酸转化为LA,这两种酶的总体特异性不同(从而影响酶的底物转化百分比)。申请人确定了串珠镰孢Δ12去饱和酶比解脂耶氏酵母Δ12去饱和酶具有更高的将LA加载到磷脂酰胆碱底物的sn-2位置上的能力(从而促进随后通过Δ6去饱和酶进行的反应)。基于此,串珠镰孢Δ12去饱和酶的超量表达和解脂耶氏酵母Δ12去饱和酶敲除的组合可能导致产物增加,用于随后转化为ARA。
在一些实施方案中,调节宿主基因组的天然DAG ATs活性可能是有用的,从而使得能够操纵解脂耶氏酵母宿主中脂质和油中PUFAs的百分比。具体地,由于预计油合成竞争含油性的多不饱和化,可能减少或灭活生物的一种或多种酰基转移酶(如PDAT和/或DGAT1和/或DGAT2)的活性,从而减少油生物合成的总体速度,而同时增加掺入脂质和油级分中的PUFAs百分比(相对于总脂肪酸)。这是由于允许多不饱和化更有效性进行;或者,也就是说,通过下调特定DAG ATs的活性,油生物合成和多不饱和化之间的底物竞争减少,有利于含油性的多不饱和化。
本领域技术人员将具有阐释下调的最优水平所必须的技术和获得所述抑制需要的方法。例如,在一些优选实施方案中,可能需要操纵单个DAG AT的活性(如建立DGAT1敲除,而不改变PDAT和DGAT2的活性)。在替代实施方案中,含油生物包含总共“n”种天然DAGATs,修饰总共“n-1”种酰基转移酶的活性,导致油生物合成的速度降低,而剩余的酰基转移酶保留其野生型活性。在一些情况下,可能需要操纵一些优选的含油生物中的所有天然DAG ATs的活性,从而在多不饱和化速度方面实现油生物合成的最优速度。
以相似的方式,申请人假定异源酰基转移酶的表达与相应的天然解脂耶氏酵母酰基转移酶的敲除的组合能够显著增加宿主细胞中生产的总ARA。具体地,如上文所提示的,对C20和更高级的脂肪酸具有特异性的异源GPAT、LPAAT、DGAT1、DGAT2、PDAT和LPCAT酰基转移酶比天然酶优选,因为解脂耶氏酵母中天然生产的PUFAs限于18:2脂肪酸,并且天然酶可能不能有效催化与更长链脂肪酸的反应。基于该结论,申请人鉴定了编码高山被孢霉中的GPAT、LPAAT、DGAT1和DGAT2的基因,并且在生产EPA的工程化耶氏酵母宿主中表达了这些基因,导致PUFA生物合成的增加(本文的实施例14-17)。随后,减少或敲除了解脂耶氏酵母中一些天然酰基转移酶(如DGAT1和DGAT2)的活性,作为减少天然和异源酰基转移酶之间的底物竞争的手段。在生产ARA的工程化耶氏酵母宿主中可以预期相似的结果。
也必须考虑途径的操纵和影响ARA生产的总体调节剂。例如,通过增加长链饱和和不饱和脂肪酸如棕榈酸(16:0)和硬脂酸(18:0)的前体的可获得性而增加碳流入PUFA生物合成途径是有用的。前者的合成依赖于C14/16延伸酶的活性,而后者的合成依赖于C16/18延伸酶的活性。因此,天然解脂耶氏酵母C14/16延伸酶(SEQ ID NOs:64和65)的超量表达显著增加了16:0和16:1脂肪酸的生产(相对于对照菌株增加22%);类似地,天然解脂耶氏酵母C16/18延伸酶(SEQ IDNOs:61和62)的超量表达增加了18:0、18:1、18:2和18:3脂肪酸的生产(相对于对照菌株增加18%)并减少了C16脂肪酸的积累(相对于对照菌株减少22%)。当然,如此处证明和Inagaki,K.等(Biosci.Biotech.Biochem.66(3):613-621(2002))的研究证明的,在本发明的某些实施方案中,共表达异源C16/18延伸酶(如来自褐家鼠[GenBank获取号AB071986;此处是SEQ ID NOs:50和51]和/或来自高山被孢霉[SEQ ID NO:53和54)可能是有用的。因此,尽管必须最小操纵解脂耶氏酵母宿主菌株表达:Δ6去饱和酶、C18/20延伸酶和Δ5去饱和酶;或Δ9延伸酶、Δ8去饱和酶和Δ5去饱和酶用于ARA的生物合成,但在进一步优选的实施方案中,宿主菌株额外包括以下至少一种:Δ9去饱和酶、Δ12去饱和酶、C14/16延伸酶和/或C16/18延伸酶。
在另一种优选实施方案中,在本发明的解脂耶氏酵母中修饰影响脂肪酸降解和TAG降解的途径,使在细胞中的酰基-CoA集合或TAG级分中积累的ARA的降解最少。这些途径分别由酰基-CoA氧化酶和脂肪酶基因代表。更具体地,酰基-CoA氧化酶(EC 1.3.3.6)催化过氧化物酶体β-氧化反应,其中每个循环的降解都产生酰基-CoA分子和比脂肪酸底物短两个碳原子的脂肪酸。解脂耶氏酵母中存在5种酰基-CoA氧化酶同工酶,由POX1、POX2、POX3、POX4和POX5基因(也称作Aco1、Aco2、Aco3、Aco4和Aco5基因)编码,所述基因分别相应于GenBank获取号AJ001299-AJ001303(也参见相应的GenBank获取号XP_504703、XP_505264、XP_503244、XP_504475和XP_502199)。每种同工酶具有不同的底物特异性;例如,POX3基因编码对短链脂肪酸具有活性的酰基-CoA氧化酶,而POX2基因编码对长链脂肪酸具有活性的酰基-CoA氧化酶(Wang H.J.,et al.J.Bacteriol.,181:5140-5148(1999))。认为任何上述基因的活性都可以被减少或消除,从而以对此处的目的有利的方式修饰本发明的宿主细胞中的过氧化物酶体β-氧化。最后,为了避免混淆,申请人将上文描述的酰基-CoA氧化酶称作POX基因,但根据一些公开文献,该术语可以与Aco基因命名互换使用。
类似地,已经在解脂耶氏酵母中检测到了一些脂肪酶(EC3.1.1.3),包括细胞内、膜结合的和细胞外酶(Choupina,A.,et al.Curr.Genet.35:297(1999);Pignede,G.,et al.J.Bacteriol.182:2802-2810(2000))。例如,Lip1(GenBank获取号Z50020)和Lip3(GenBank获取号AJ249751)是细胞内或膜结合的,而Lip2(GenBank获取号AJ012632)编码细胞外脂肪酶。这些脂肪酶中的每一种都是破坏的靶,因为这些酶催化反应,所述反应中TAG和水直接降解为DAG和脂肪酸阴离子。
在其它替代实施方案中,可以在解脂耶氏酵母的优选宿主菌株中操纵一些脂肪酶的活性。磷脂酶在膜脂质的生物合成和降解中起关键作用。更具体地,术语“磷脂酶”是指具有水解甘油磷脂中的一个或多个酯键的能力的一组异源酶。尽管所有的磷脂酶都靶定磷脂作为底物,每一种酶都具有切割特定酯键的能力。因此,磷脂酶命名区分了各个磷脂酶,并且表明了磷脂分子中靶定的特定键。例如,磷脂酶A1(PLA1)水解甘油部分sn-1位置上的脂肪酰基酯键,而磷脂酶A2(PLA2)除去该分子sn-2位置上的脂肪酸。PLA1(EC 3.1.1.32)和PLA2(EC 3.1.1.4)的作用分别导致游离脂肪酸和2-酰基溶血磷脂或1-酰基溶血磷脂的积累。磷脂酶C(PLC)(EC 3.1.4.3)水解磷脂主链中的磷酸二酯键,并且依赖于涉及的特定磷脂种类,即,磷脂酰胆碱、磷脂酰乙醇胺等(如PLC1负责以下反应:1-磷脂酰-1D-肌醇4,5-二磷酸+H2O=1D-肌醇1,4,5-三磷酸+DAG;ISC1编码肌醇鞘磷脂特异性磷脂酶C[Sawai,H.,et al.J.Biol.Chem.275,39793-39798(2000)])。磷脂酶D(PLD)(EC 3.1.4.4)切割第二个磷酸二酯键,产生磷脂酸和胆碱或乙醇胺,再次依赖于涉及的磷脂类型。磷脂酶B(PLB)具有除去sn-1和sn-2脂肪酸的能力,并且在具有水解酶(其中该酶从磷脂[PLB活性]和溶血磷脂[溶血磷脂酶活性]切割脂肪酸,以释放脂肪酸)和溶血磷脂酶-转酰基酶(其中该酶能够通过将游离脂肪酸转移到溶血磷脂上而产生磷脂)活性方面是独特的。超量表达一种或多种所述磷脂酶可能是有用的,以增加在转化的耶氏酵母宿主细胞的总油级分中积累的ARA浓度。假定观察到该影响,因为磷脂酶将酰基从PC释放到CoA集合中,以延伸或掺入到甘油三酯中。
在另一种替代实施方案中,也可以在解脂耶氏酵母的优选宿主菌株中操纵CDP-胆碱途径中负责磷脂酰胆碱(PC)生物合成的酶,作为增加总ARA生物合成的手段。通过编码二酰基甘油胆碱磷酸转移酶(EC 2.7.8.2)的解脂耶氏酵母CPT1基因的超量表达证明了该技术的用途,从而导致工程化的解脂耶氏酵母菌株的EPA生物合成增加。本领域技术人员熟悉PC生物合成途径,并且了解其它合适的候选酶。
尽管操纵诸如上文描述的生物化学途径的方法是本领域技术人员公知的,下文将简述用于减少或消除天然基因活性的一些技术的综述。这些技术可以用于下调上文描述的天然解脂耶氏酵母Δ12去饱和酶、GPAT、LPAAT、DGAT1、DGAT2、PDAT、LPCAT、酰基-CoA氧化酶2(Aco2或Pox2)、酰基-CoA氧化酶3(Aco3或Pox3)和/或脂肪酶基因的表达。
尽管本领域技术人员很容易确定用于减少或消除天然基因活性的最合适的技术,但一般地,可以通过例如以下方法减少或消除特定基因的内源活性:1.)通过插入、取代和/或缺失靶基因的全部或部分而破坏基因;2.)提供用于将反义序列转录为基因的转录产物的盒;3.)使用天然具有很少或不具有[或经过突变而具有很少或不具有]特定基因的活性的宿主细胞;4.)超量表达诱变的遗传亚单位(hereosubunit)(即,在包含两个或多个遗传亚单位的酶中),从而由于“显性失活作用”减少酶活性;和5.)采用iRNA技术。在某些情况下,也可以通过利用特定抑制剂(如去饱和酶抑制剂,如U.S.4,778,630中描述的那些)实现不需要的基因途径的抑制。
为了进行基因破坏,将外源DNA片段(通常是选择标记基因,但任选是在表达时赋予需要的表型的嵌合基因或嵌合基因簇)插入要破坏的结构基因,以便破坏它的编码序列,并因此功能性灭活所述基因。将所述破坏盒转化入宿主细胞,导致了非通过同源重组用无功能的破坏的基因替代功能性天然基因(例如,参见:Hamilton et al.J.Bacteriol.171:4617-4622(1989);Balbas et al.Gene 136:211-213(1993);Gueldener et al.Nucleic Acids Res.24:2519-2524(1996);和Smith et al.Methods Mol.Cell.Biol.5:270-277(1996))。
当靶基因的序列已知时,反义技术是下调基因的另一种方法。为了实现这一目的,克隆来自所需基因的核酸片段,并且可操作地与启动子连接,以便RNA的反义链能够转录。然后将该构建体导入宿主细胞,并且生产RNA的反义链。反义RNA能通过阻止编码感兴趣的蛋白的mRNA的积累,抑制基因表达。本领域技术人员可以理解的是,特殊考虑与使用反义技术相关,以便减少特定基因的表达。例如,反义基因的适当水平的表达可能需要使用不同的嵌合基因,其中采用本领域技术人员所公知的不同的调节元件。
尽管当序列已知时,靶定的基因破坏和反义技术提供了下调基因的有效方法,业已开发了不是基于序列的其他特异性更低的方法(例如通过UV辐射/化学试剂进行诱变,或使用可转座元件/转座子;参见WO 04/101757)。
在替代的实施方案中,可以通过操纵控制蛋白表达的调节序列减少特定基因的内源活性。如本领域公知的,与编码序列相关的调节序列包括转录和翻译“控制”核苷酸序列,其位于编码序列上游(5’非编码序列)、序列内、或下游(3’非编码序列),并且,影响相关编码序列的转录、RNA加工或稳定性、或翻译。因此,特定基因的调节序列的操纵可以表示基因启动子、翻译前导序列、内含子、增强子、起始控制区、聚腺苷酸化识别序列、RNA加工位点、效应物结合位点和茎-环结构的操纵。因此,例如,可以去除或破坏DAG AT的启动子,以下调DAG AT的表达,从而实现脂质和油生物合成的速度降低。或者,可以用启动子活性比天然启动子低的异源启动子取代驱动DAG AT表达的天然启动子。用于操纵调节序列的方法是本领域技术人员公知的。
总之,采用此处提供的教导,转化的含油微生物宿主将生产占微生物宿主的总脂质的至少约5%的ARA,优选占总脂质的至少约10%的ARA,更优选占总脂质的至少约15%的ARA,更优选占总脂质的至少约20%的ARA,最优选占总脂质的至少约25-30%的ARA。
用于生产ARA的发酵过程
转化的微生物宿主细胞是在优化嵌合基因(如编码去饱和酶、延伸酶、酰基转移酶等)的表达,并且产生最大和最经济的ARA产率的条件下生长的。一般,可以优化的培养基条件包括碳源的类型和用量、氮源的类型和用量、碳氮比、氧水平、生长温度、pH、生物量生产期的长度、油积累期的长度,以及细胞收获时间。解脂耶氏酵母在复杂培养基(例如,酵母提取物-蛋白胨-右旋糖培养液(YPD))或缺少生长所必需的成分,以便强制选择需要的表达盒的限定基本培养基)(例如,Yeast Nitrogen Base(DIFCO Laboratories,Detroit,MI))中生长。
本发明的发酵培养基必须包括合适的碳源。合适的碳源包括,但不局限于:单糖(例如,葡萄糖、果糖)、二糖(例如,乳糖、蔗糖)、寡糖、多糖(例如,淀粉、纤维素或它们的混合物)、糖醇(例如,甘油)或可更新饲料(例如,干酪乳清渗透物、玉米浆、甜菜糖浆、大麦芽)的混合物。另外,碳源可以包括链烷、脂肪酸、脂肪酸酯、甘油一酯、甘油二酯、甘油三酯、磷脂,和脂肪酸的各种商业来源,包括植物油(例如,大豆油)和动物脂肪。另外,碳源可以包括一碳源(例如,二氧化碳、甲醇、甲醛、甲酸和含碳的胺),业已证实了其转化成关键生物化学中间体的代谢转化。因此,预计,用于本发明的碳源包括可以包括多种含碳的来源。尽管上述所有碳源及其混合物预计都适合本发明,优选的碳源是糖和/或脂肪酸。最优选的是葡萄糖和/或含有10-22个碳的脂肪酸。
氮可以由无机(例如,(NH4)2SO4)或有机来源(例如,尿素或谷氨酸)提供。除了合适的碳和氮源之外,发酵培养基必须还包括合适的矿物质、盐、辅因子、缓冲液、维生素,和本领域技术人员已知的适合含油酵母生长,并且促进ARA生产所需要的酶途径的其他成分。特别关注若干金属离子(例如,Mn+2、Co+2、Zn+2、Mg+2),它们能促进脂质和PUFAs的合成(Nakahara,T.et al.,Ind.Appl.SingleCell Oils,D.J.Kyle and R.Colin,eds.pp 61-97(1992))。
本发明的优选的生长培养基是常见的商业制备的培养基,如YeastNitrogen Base(DIFCO Laboratories,Detroit,MI)。其他确定的或合成的生长培养基也可以使用,并且适合特定解脂耶氏酵母生长的培养基是微生物学或发酵科学领域的技术人员所公知的。用于发酵的合适的pH范围通常为大约pH 4.0-pH 8.0,其中pH 5.5-pH 7.0是起始生长条件的优选范围。发酵可以在需氧或厌氧条件下进行,其中,微需氧条件是优选的。
通常,高水平PUFAs在含油酵母细胞中的积累,需要两个阶段的过程,因为在脂肪的生长和合成/储存之间的代谢状态必须是“平衡的”。因此,最优选的是,需要两个阶段的发酵过程在解脂耶氏酵母中生产ARA。该方法描述于WO 2004/101757,在生长过程中具有多个合适的发酵过程设计(即分批、补料分批和连续)和考虑。
ARA的纯化和加工
PUFAs,包括ARA,可以作为游离脂肪酸形式出现在宿主微生物中,或者以诸如酰基甘油、磷脂、硫脂或糖脂的脂化形式出现在宿主微生物中,并且可以通过本领域所熟知的多种方法从宿主细胞中提取。酵母脂质的提取技术、品质分析和可接受性标准的一篇综述是:Z.Jacobs(Critical Reviews in Biotechnology 12(5/6):463-491(1992))。有关下游加工的简单综述还可以参见A.Singh和O.Ward(Adv.Appl.Microbiol.45:271-312(1997))。
一般地,纯化ARA和其它PUFAs的方法可以包括用有机溶剂萃取、超声波处理、超临界流体萃取(例如,使用二氧化碳)、皂化和物理方法,如压力,或它们的组合。其它细节可参考WO 2004/101757的教导。
可以氢化含有已经精制和/或纯化的ARA的油,从而得到具有多种熔解特性和质地的脂肪。很多经过加工的脂肪,包括糊、甜食脂肪、硬质脂肪、人造黄油、烘烤起酥等,需要室温下的各种程度的固化,并且仅仅可以通过改变原料油的物理特性而制备。这最常见是通过催化氢化实现的。
氢化是一种化学反应,其中在诸如镍的催化剂辅助下将氢加入不饱和脂肪酸双键。氢化具有两个主要作用。首先,由于不饱和脂肪酸含量的减少,油的氧化稳定性增加。其次,油的物理性质改变,这是因为脂肪酸修饰增加了熔点,在室温下得到半液体或固体脂肪。
存在许多影响氢化反应的变量,其随后改变终产物的组成。包括压力、温度、催化剂类型和浓度、搅拌和反应器设计的操作条件是可以控制的较重要的参数中的一些。选择性氢化条件可以相对于较不饱和的脂肪酸而言优先用于氢化更饱和的脂肪酸。通常用非常轻的或刷洗氢化来增加液体油的稳定性。进一步的氢化将液体油转化为物理固体的脂肪。氢化的程度依赖于针对特定终产物设计的所需性能和熔解特征。用于制备烘烤产品的液体起酥、用于商业煎炸和烘烤操作的固体脂肪和起酥,以及用于制造人造黄油的碱原料是通过氢化得到的多种可能的油和脂肪产品中的一些。氢化和氢化产物的更详细描述可以参见Patterson,H.B.W.,Hydrogenation of Fats and Oils:Theory andPractice.The American Oil Chemists’Society,1994。
用于食料中的生产ARA的解脂耶氏酵母菌株
目前的市场需要大量掺入ω-3和/或ω-6脂肪酸(特别是ARA、EPA和DHA)的食品和饲料产品。考虑本发明的包含ARA的酵母微生物油将在食品和饲料产品中起作用,赋予本发明的制剂的健康益处。
由此处描述的酵母宿主生产的含ω-3和/或ω-6脂肪酸的微生物油将适用于多种食品和饲料产品中,包括但不限于食物类似物、肉类产品、谷物产品、烘烤食品、快餐和乳制品。此外,本发明的微生物油可以用于制剂中,在包括医学营养品、饮食补充物、婴儿配方和药品的医学食品中赋予健康益处。食品加工和食品制剂领域的技术人员将理解微生物油的量和组成如何添加到食物或食品中。所述量在此称作“有效”量,并且依赖于食品或饲料产品、该产品意欲补充的饮食或该医学食品或医学营养品意欲矫正或治疗的医学状况。
可以用本领域技术人员公知的方法制备食物类似物。可以提到的是肉类似物、苷酪类似物、乳类似物等。从大豆制备的肉类似物含大豆蛋白或豆腐和其它混合在一起的成分,以模拟各种肉类。这些肉替代品以冷冻、罐装或干燥食品出售。通常,它们可以以它们所替代的食品相同的方式使用。肉类似物的实例包括但不限于:火腿类似物、香肠类似物、咸肉类似物等。
根据功能和组成特征,可以将食物类似物分类为模拟物或替代物。例如,模拟干酪仅仅需要模拟其设计用于替代的干酪。然而,只有当一种产品在营养上等同于它所要替代的干酪和满足该干酪的最小组成需要,才能将其称作替代干酪。因此,替代干酪通常比模拟干酪具有更高的蛋白水平,并且可以用维生素和矿物质进行强化。
乳类似物和非乳制食品包括但不限于:模拟乳和非乳制甜点(如从大豆和/或大豆蛋白产品制备的那些)。
肉产品包括广泛的产品。在美国,“肉”包括从牛、狗和羊生产的“红肉”。除了红肉外,还有禽类,包括鸡、火鸡、鹅、珍珠鸡、鸭,以及鱼类和甲壳类动物。存在各种类型的调味和加工的肉类产品:新鲜的、熟化和煎炸的、以及熟化和蒸煮的。香肠和热狗是加工的肉产品的实例。因此,此处用到的“肉产品”包括但不限于加工的肉产品。
谷物食品是来源于谷物加工的食品。谷物包括来自产生可食用谷物(籽粒)的草本科植物的任何植物。最常见的谷物是大麦、玉米、小米、燕麦、奎藜、稻、黑麦、高粱、黑小麦、小麦和野生稻。谷物食品的实例包括但不限于:全谷、全谷粗粉、粗谷粉、面粉、麸、胚芽、早餐谷物、寄出食品、意大利面食等。
烘烤食品包括上文描述的任何谷物食品,并且以能够烘烤,即通过热干燥或硬化的方式烘烤或加工过。烘烤食品的实例包括但不限于:面包、蛋糕、炸面圈、面包条、意大利面食、面包屑、烘烤的快餐、小饼干、小薄脆饼干、小甜饼和小脆饼干。如上文提到的,本发明的油可以用作一种成分。
快餐食品包括上文或下文描述的任何食品。
煎炸食品包括经过煎炸的上文或下文描述的任何食品。
饮料可以是液体或干粉形式。
例如,可以提到的是非碳酸饮料;果汁、新鲜的、冷冻的、罐装的或浓缩饮料;调味的或普通的乳饮料等。成人和婴儿营养配方是本领域公知的并且可以商购(如来自Ross Products Division,AbbottLaboratories的和)。婴儿配方是给婴儿或幼儿喂食的液体或重配粉末。它们作为母乳的替代品。婴儿配方在婴儿饮食中起作特殊作用,因为它们通常是婴儿营养物质的唯一来源;并且尽管母乳喂养是婴儿的最佳营养,但婴儿配方足够接近,因此婴儿不仅存活,还茁壮成长。婴儿配方正在变得越来越接近于母乳。
乳制品是来源于乳的产品。乳类似物或非乳制品来源于除乳以外的来源,例如,上文讨论的大豆乳。这些产品包括但不限于:全乳、脱脂乳、发酵乳产品如酸奶酪或酸奶、乳酪、黄油、炼乳、脱水乳、调咖啡用白油、咖啡伴侣、冰激凌、干酪等。
可以掺入本发明的含ARA的油的其它食品包括,例如:口香糖、糖果和糖霜、明胶和布丁、硬糖和软糖、果酱和果冻、白砂糖、糖替代品、甜调料、糕点装饰物和糖浆,以及干粉混合物。
健康食品和药品
健康食品是赋予健康益处的任何食品,包括功能性食品、医学食品、医学营养品和饮食补充物。此外,本发明的微生物油可以用于标准药物组合物中。包含ARA的本发明的工程化的解脂耶氏酵母菌株或由其制备的微生物油可以容易地掺入上述任何食品中,从而生产例如功能性或医学食品。例如,包含ARA的更浓缩的制剂包括胶囊、粉末、片剂、软凝胶、凝胶帽、液体浓缩物和乳状液,它们可以用作人或人以外的其它动物的营养补充物。
用于饮食补充物中
更浓缩的含ARA的制剂包括胶囊、粉末、片剂、软凝胶、凝胶帽、液体浓缩物和乳状液,它们可以用作人或人以外的其它动物的营养补充物。特别地,本发明的ARA油特别适于掺入饮食补充物,如婴儿配方或婴儿食物。
婴儿配方是给婴儿或幼儿喂食的液体或重配粉末。此处将“婴儿配方”定义为肠道营养品,其可以在哺乳期婴儿中代替母乳,并且通常包含与在水溶液中与所需百分比的碳水化合物和蛋白混合的所需百分比的脂肪(例如参见U.S.4,670,285)。基于世界范围的组成研究以及专家组规定的水平,平均母乳典型地含约0.20%-0.40%的总脂肪酸(假定大约50%的热卡来自脂肪);并且,DHA与ARA的比例通常是大约1:1-1:2(参见例如Enfamil LIPILTM[Mead Johnson & Company]和Similac AdvanceTM[Ross Products Division,Abbott Laboratories]的配方)。婴儿配方在婴儿饮食中起作特殊作用,因为它们通常是婴儿营养物质的唯一来源;并且尽管母乳喂养是婴儿的最佳营养,但婴儿配方足够接近,因此婴儿不仅存活,还茁壮成长。
用于动物饲料中
动物饲料在此一般定义为意欲用作除人之外的动物的饲料或混合于饲料中的产品。并且,如上文提到的,本发明的包含ARA的油可以用作各种动物饲料中的成分。
更具体地,尽管不进行限制,预计本发明的油可以用于宠物食品中、反刍动物和禽类动物食品中以及水产养殖食品中。宠物食品是意欲喂饲宠物[如狗、猫、鸟类、爬行动物、啮齿类动物]的产品;这些产品包括上文所述的谷物和健康食品,以及肉和肉副产品、大豆蛋白产品、草和干草产品(如紫花苜蓿、梯牧草、燕麦或雀麦草、蔬菜)。反刍动物和禽类动物食品是意欲喂饲给例如火鸡、鸡、牛和猪的食品。至于上文所述的宠物食品,这些食品包括谷物和上文列出的健康食品、大豆蛋白产品、肉和肉副产品、以及草和干草产品。水产养殖食品(或“水产养殖饲料”)是意欲用于水产养殖的产品,所述水产养殖涉及淡水或海水中水生生物和/或动物的繁殖、培养和饲养。
认为本发明的生产高浓度ARA、EPA和/或DHA的工程化的解脂耶氏酵母菌株特别可以用于掺入大多数动物饲料配方中。除了提供必要的ω-3和/或ω-6 PUFAs,酵母本身是一种有用的蛋白和其它营养物质(如维生素、矿物质、核酸、复杂碳水化合物等)的来源,其能够有利于总体动物健康和营养,并且增加配方的美味性。因此,认为加入包含本发明的重组生产宿主的酵母生物量,将是动物饲料配方中的极佳的额外饲料营养物质来源。更具体地,解脂耶氏酵母(ATCC#20362)具有以下大约的化学组成(相对于干细胞总量的百分比):35%蛋白、40%脂质、10%碳水化合物、5%核酸、5%灰分和5%水。此外,在碳水化合物级分中,β-葡聚糖占大约45.6mg/g,甘露聚糖占大约11.4mg/g,几丁质占大约52.6mg/g(而海藻糖是最少的成分[大约0.7mg/g])。
大量的文献检验了β-葡聚糖、甘露聚糖和几丁质的免疫调节作用。最充分地研究了细菌和真菌细胞壁的主要成分β-葡聚糖刺激非特异性免疫(即“免疫刺激作用”)从而改进水产养殖物种、宠物和畜牧动物以及人的健康的方式,类似地,也发现几丁质和甘露聚糖是有用的免疫刺激剂。具体地,使用β-葡聚糖可以获得免疫应答的总体增强,因为这些β-1,3-D-聚葡萄糖分子以非特异性方式刺激白细胞(如巨噬细胞、中性粒细胞和单核细胞)的产生,从而增加抗多种致病抗原或环境刺激物的敏感性和防御能力。更具体地,许多研究证明了β-葡聚糖能够:赋予增强的抗病毒、细菌、真菌和寄生虫感染的保护作用;当与抗生素和疫苗联合使用时,施加佐剂作用;增强伤口愈合;抵抗自由基导致的破坏;增强肿瘤消退;调节细菌内毒素的毒性;和增强粘膜免疫(综述于Raa,J.et al.,Norwegian Beta GlucanResearch,Clinical Applications of Natural Medicine.Immune:Depressions Dysfunction & Deficiency(1990))。记载了酵母β-葡聚糖、甘露聚糖和几丁质在传统动物饲养和水产养殖中的作用的现有文献的例子包括:L.A.White et al.(J.Anim.Sci.80:2619-2628(2002)),supplementation in weanling pigs;K.S.Swanson et al.(J.Nutr.132:980-989(2002)),supplementation in dogs;J. et al.(Vet.Immunol.Immonopath.85:41-50(2002)),whole Saccharomycescerevisiae administered to gilthead seabream;A.Rodríguez et al.(FishShell.Immuno.16:241-249(2004)),whole Mucor circinelloidesadministered to gilthead seabream;M.Bagni et al.(Fish Shell.Immuno.18:311-325(2005)),supplementation of sea bass with a yeastextract containing β-glucans;J.Raa(In:Cruz-Suárez,L.E.,Ricque-Marie,D.,Tapia-Salazar,M.,Olvera-Novoa,M.A.yCivera-Cerecedo,R.,(Eds.).Avances en Nutrición Acuícola V.Memorias del V Simposium Internacional de Nutrición Acuícola.19-22Noviembre,2000.Mérida,Yucatán,Mexico),a review of the use ofimmune-stimulants in fish and shellfish feeds。
基于解脂耶氏酵母的独特蛋白:脂质:碳水化合物组成,以及独特的复杂碳水化合物谱(包括甘露聚糖:β-葡聚糖:几丁质的大约1:4:4.6的比例),认为本发明的基因工程化的酵母细胞(或其部分)是动物饲料配方的有用添加剂(例如作为完整的[冻干的]酵母细胞、作为纯化的细胞壁、作为纯化的酵母碳水化合物或在多种其它分级分离形式内)。
在水产养殖业方面,对各种鱼类物种的营养需要的了解增加和饲料制造的技术进步,使得制造的或人工饲料(配方饲料)的开发和使用能够补充或代替水产养殖业的天然饲料。但是,一般地,包含在鱼类的水产养殖饲料中的各种营养物质的一般比例包括(以干饲料的百分比表示):32-45%蛋白、4-28%脂肪(其中至少1-2%是ω-3和/或ω-6 PUFAs)、10-30%碳水化合物、1.0-2.5%矿物质和1.0-2.5%维生素。可以任选将大量其它成分加入配方中。这些其它成分包括:(1)类胡萝卜素,特别是用于鲑鱼类和观赏性的“水族馆”鱼类,以分别增强新鲜感和皮肤颜色;(2)结合剂,给饲料团提供稳定性,并且减少营养物质泄露到水中(如牛心、淀粉、纤维素、果胶、明胶、阿拉伯树胶、洋槐豆、琼脂、鹿角菜胶和其它藻酸盐);(3)防腐剂,如抗微生物剂和抗氧化剂,以延长鱼饲料的保质期,并且减少脂肪的腐臭(如维生素E、丁基化的羟基苯甲醚、丁基化的羟基甲苯、乙氧基喹、和丙酸、苯甲酸或山梨酸的钠盐和钾盐);(4)化学引诱物和调味剂,以增强饲料的美味性及其摄食;和(5)其它饲料。所述其它饲料可以包括诸如纤维和灰分的材料(分别用作填料以及钙和磷的来源)和植物物质和/或鱼或鱿鱼食物(如活的、冷冻的或干燥的藻类、海虾、轮虫或其它浮游动物),以增强饲料的营养价值并增加鱼类对它的接受性。Nutrient Requirements of Fish(National ResearchCouncil,National Academy:Washington D.C.,1993)提供了鱼类的必须营养物质和各种成分的营养含量的详细描述。
制造水产养殖配方需要考虑大量因素,因为完整的饲料必须是营养平衡的、美味的、在水中稳定的,并且具有合适的大小和质地。关于水产养殖饲料的营养组成,可以参考Handbook on Ingredients forAquaculture Feeds(Hertrampf,J.W.and F.Piedad-Pascual.KluwerAcademic:Dordrecht,The Netherlands,2000)和Standard Methodsfor the Nutrition and Feeding of Farmed Fish and Shrimp(Tacon,A.G.J.Argent Laboratories:Redmond,1990)。一般地,饲料配制为干的(即最终水含量是6-10%)、半湿的(即水含量是35-40%)或湿的(即水含量是50-70%)。干饲料包括以下这些:干成分的简单疏松的混合物(即“浆”或“粗粉”);压实的小团、碎屑或颗粒;以及薄片。根据鱼类的饲养需要,可以制备沉入水中或漂浮的小团。半湿和湿饲料是从单个或混合的成分(如无价值的鱼类或煮熟的豆类)制备,并且可以成形为饼状或球状。
认为本发明的生产高浓度ARA的工程化的解脂耶氏酵母菌株特别可以用于包括在大多数水产养殖饲料中。除了提供必要的ω-6PUFAs,酵母自身是可以增加配方的美味性的有用蛋白来源。在替代的实施方案中,由本发明的解脂耶氏酵母菌株生产的油可以在从细胞团提取和纯化后,直接加入水产养殖饲料配方中。
优选实施方案的描述
本发明证明了占含油酵母解脂耶氏酵母的总脂质比例的最多10-14%的ARA的合成。如图4所示,通过将多个基因整合到野生型ATCC#20362解脂耶氏酵母中,建立了许多解脂耶氏酵母菌株,其中每种转化菌株都能够生产不同量的PUFAs(包括ARA)。菌株Y2034和Y2047(表达Δ6去饱和酶/Δ6延伸酶途径)和菌株Y2214(表达Δ9延伸酶/Δ8去饱和酶途径)的完整脂质谱示于下表10。脂肪酸鉴定为16:0、16:1、18:0、18:1(油酸)、18:2(LA)、GLA、DGLA和ARA;并且每一种所述脂肪酸的组成表示为总脂肪酸的百分比。“脂质% dcw”表示通过细胞干重测量的细胞中脂质的百分比。
表10
解脂耶氏酵母菌株Y2034、Y2047和Y2214的脂质谱
下文描述了菌株Y2047中包含的基因修饰的更详细概括(其中完整的细节提供于实施例中):
(1)1个拷贝的串珠镰孢Δ12去饱和酶的表达,在FBA::F.Δ12::LIP2嵌合基因中;
(2)来源于高山被孢霉Δ6去饱和酶的1个拷贝的合成Δ6去饱和酶基因(为在解脂耶氏酵母中表达进行了密码子优化)的表达,在FBAIN::Δ6S::LIP1嵌合基因中;
(3)来源于人Δ5去饱和酶的1个拷贝的合成Δ5去饱和酶基因(为在解脂耶氏酵母中表达进行了密码子优化)的表达,在TEF::H.D5S::PEX16嵌合基因中;
(4)来源于高山被孢霉高亲和力C18/20延伸酶的1个拷贝的合成的高亲和力C18/20延伸酶基因(为在解脂耶氏酵母中表达进行了密码子优化)的表达,在FBAIN::EL1S::PEX20嵌合基因中;
(5)来源于金黄色破囊壶菌C18/20延伸酶的1个拷贝的合成C18/20延伸酶基因(为在解脂耶氏酵母中表达进行了密码子优化)的表达,在TEF::EL2S::XPR嵌合基因中;
(6)编码编码苹果酸异丙酯脱氢酶的天然解脂耶氏酵母Leu2基因的破坏。
相似地,下文描述了菌株Y2214内包含的基因修饰的更详细概括(其中完整的细节提供于实施例中):
(1)来源于球等鞭金藻Δ9延伸酶基因的5个拷贝的合成Δ9延伸酶基因(为在解脂耶氏酵母中表达进行了密码子优化)的表达,在GPAT::IgD9e::PEX20、TEF::IgD9e::LIP1和FBAINm::IgD9e::OCT嵌合基因中;
(2)来源于纤细眼虫Δ8去饱和酶基因的3个拷贝的合成Δ8去饱和酶基因(为在解脂耶氏酵母中表达进行了密码子优化)的表达,在FBAIN::D8SF::PEX16和GPD::D8SF::PEX16嵌合基因中;
(3)2个拷贝的高山被孢霉Δ5去饱和酶的表达,在FBAIN::MAΔ5::PEX20和GPAT::MAΔ5::PEX20嵌合基因中;
(4)来源于球等鞭金藻Δ5去饱和酶的2个拷贝的合成Δ5去饱和酶基因(为在解脂耶氏酵母中表达进行了密码子优化)的表达,在YAT1::I.D5S::LIP1和GPM/FBAIN::I.D5S::OCT嵌合基因中;
(5)1个拷贝的串珠镰孢Δ12去饱和酶的表达,在FBAIN::F.D12S::PEX20嵌合基因中;
(6)来源于褐家鼠rELO基因的1个拷贝的合成C16/18延伸酶基因(为在解脂耶氏酵母中表达进行了密码子优化)的表达,在GPM/FBAintron::rELO2S::OCT嵌合基因中;和
(7)编码酵母氨酸脱氢酶的天然解脂耶氏酵母Lys5基因的破坏。
尽管申请人分别证明了在这些特定的解脂耶氏酵母重组菌株中生产了11%和14% ARA,但根据此处公开的本发明,宿主细胞中EPA的浓度可以通过额外的基因修饰显著增加。此外,基于此处描述的教导和结果可以预见到本领域技术人员采用Δ6去饱和酶/Δ6延伸酶途径和/或Δ9延伸酶/Δ8去饱和酶途径,采用含油酵母作为合成多种ω-3和/或ω-6PUFAs的平台建立的可行性和商业用途。
实施例
在以下实施例中进一步限定本发明。应当理解的是,在这些实施方案中,尽管指明了是本发明的优选实施方案,但是,是以说明形式提供的。通过上文的讨论和这些实施例,本领域技术人员可以确定本发明的实质性特征,并且,在不超出本发明的构思和范围的前提下,可以对本发明进行各种改变和改进,以便适应各种用途和条件。
一般方法
用于实施例中的标准重组DNA和分子克隆技术为本领域所熟知的,并且披露于以下文献中1.)Sambrook,J.,Fritsch,E.F.andManiatis,T.Molecular Cloning:A Laboratory Manual;Cold SpringHarbor Laboratory:Cold Spring Harbor,NY(1989)(Maniatis);2.)T.J.Silhavy,M.L.Bennan,and L.W.Enq uist,Experiments withGene Fusions;Cold Spring Harbor Laboratory:Cold Spring Harbor,NY(1984);和3.)Ausubel,F.M.et al.,Current Protocols in MolecularBiology,由Greene Publishing Assoc.and Wiley-Interscience(1987)出版。
适合微生物培养物维持和生长的材料和方法为本领域所公知。适合在以下实施例中使用的技术可以参见以下文献:Manual of Methodsfor General Bacteriology(Phillipp Gerhardt,R.G.E.Murray,RalphN.Costilow,Eugene W.Nester,Willis A.Wood,Noel R.Krieg and G.Briggs Phillips,Eds),American Society for Microbiology:Washington,D.C.(1994));或参见Thomas D.Brock inBiotechnology:A Textbook of Industrial Microbiology,2nd ed.,Sinauer Associates:Sunderland,MA(1989)。用于生长和维持微生物细胞的所有试剂,限制酶和材料都是从Aldrich Chemicals(Milwaukee,WI),DIFCO Laboratories(Detroit,MI),GIBCO/BRL(Gaithersburg,MD),或Sigma Chemical Company(St.Louis,MO)获得的,除非另有说明。
大肠杆菌(XL1-Blue)感受态细胞是从Stratagene公司(SanDiego,CA)购买的。大肠杆菌菌株通常是在37℃下在Luria Bertani(LB)平板上生长的。
按照标准方法(Sambrook等,同上)进行一般分子克隆。寡核苷酸是由Sigma-Genosys(Spring,TX)合成的。在50μl总体积中进行各个PCR扩增反应,其中包括:PCR缓冲液(含10mM KCl、10mM(NH4)2SO4、20mM Tris-HCl(pH 8.75)、2mM MgSO4、0.1% Triton X-100)、100μg/mL BSA(终浓度)、每种三磷酸脱氧核糖核苷酸各200μM、每种引物各10pmole和1μl Pfu DNA聚合酶(Stratagene,San Diego,CA),除非另有说明。用Stratagene’sQuickChangeTM定点诱变试剂盒,按照生产商的说明进行定点诱变。当PCR或定点诱变参与亚克隆时,对所述构建体进行测序,以便证实没有将误差导入所述序列。将PCR产物克隆到Promega’s pGEM-T-easy载体(Madison,WI)中。
用载体和插入片段特异性引物的组合,采用染料终止子技术(U.S.5,366,860;EP 272,007)在ABI自动测序仪上产生DNA序列。在测序仪(Gene Codes Corporation,Ann Arbor,MI)上进行序列编辑。所有序列代表每个方向覆盖至少两次。用DNASTAR软件(DNA Star,Inc.)完成基因序列的比较。或者,基因序列的操作使用可以从GeneticsComputer Group Inc.(Wisconsin Package Version 9.0,GeneticsComputer Group(GCG),Madison,WI)获得的程序组进行。使用了所述GCG程序“Pileup”,空位形成缺省值为12,空位延伸缺省值为4。使用了GCG“Gap”或“Bestfit”程序,缺省的空位形成罚分为50,缺省的空位延伸罚分为3。除非另有说明,在所有其他场合下,都使用GCG程序的缺省参数。
进行BLAST(Basic Local Alignment Search Tool;Altschul,S.F.,et al.,J.Mol.Biol.215:403-410(1993)和Nucleic Acids Res.25:3389-3402(1997))检索,以鉴定与包含在BLAST“nr”数据库中的序列(包括所有非冗余的GenBank CDS翻译、来源于三维结构Brookhaven Protein数据库的序列、SWISS-PROT蛋白序列数据库、EMBL和DDBJ数据库)具有相似性的分离的序列。按照所有可读框翻译序列,并且采用NCBI提供的BLASTX算法(Gish,W.andStates,D.J.Nature Genetics 3:266-272(1993))比较与“nr”数据库中包含的所有公众可获得的蛋白序列的相似性。
根据同一性百分比、相似性百分比和预期值报告了BLAST比较的结果,该结果概括了与询问序列具有最多相似性的序列。“同一性百分比”定义为在两种蛋白之间相同的氨基酸的百分比。“相似性百分比”定义为在两种蛋白之间相同或保守的氨基酸的百分比。“预期值”估计了匹配的统计学显著性,该显著性用特定评分指出匹配数目,该评分是在对该大小的数据库进行检索而预期的,这种预期完全是随机的。
缩写的含义如下:“sec”表示秒,“min”表示分钟,“h”表示小时,“d”表示天,“μL”表示微升,“mL”表示毫升,“L”表示升,“μM”表示微摩尔浓度,“mM”表示毫摩尔浓度,“M”表示摩尔浓度,“mmol”毫摩尔,“μmole”表示微摩尔,“g”表示克,“μg”表示微克,“ng”表示纳克,“U”表示单位,“bp”表示碱基对,而“kB”表示千碱基。
解脂耶氏酵母的转化和培养
从美国典型培养物保藏中心(Rockville,MD)购买解脂耶氏酵母菌株ATCC #20362、#76982和#90812。解脂耶氏酵母菌株在28℃下在YPD琼脂(1%酵母提取物,2%细菌用蛋白胨,2%葡萄糖,2%琼脂)上生长。或者,“SD”培养基包含:含有硫酸铵而不含氨基酸的0.67%的酵母氮基和2%葡萄糖。
根据Chen,D.C.et al.(Appl.Microbiol Biotechnol.48(2):232-235(1997))的方法进行解脂耶氏酵母的转化,除非另外指明。简言之,将耶氏酵母划线到YPD平板上,在30℃下生长大约18小时。从平板上刮下几个大满环的细胞,重悬于1ml转化缓冲液中,该缓冲液含有:2.25mL 50% PEG,平均分子量3350;0.125mL 2M醋酸锂,pH 6.0;0.125mL 2M DTT;和50μg剪切的鲑鱼精子DNA。然后,将大约500ng线性化的质粒DNA在100μl重悬的细胞中温育,以15分钟间隔涡旋下在39℃维持1小时。将细胞铺板到选择培养基板上,在30℃下维持2-3天。
为了选择转化体,一般使用SD培养基或基本培养基(“MM”);MM的组成如下:不含硫酸铵或氨基酸的0.17%酵母氮基(DIFCOLaboratories,Detroit,MI)、2%葡萄糖、0.1%脯氨酸,pH 6.1。合适的情况下加入腺嘌呤、亮氨酸、赖氨酸和/或尿嘧啶补充物,达到0.01%的终浓度(从而得到“MMA”、“MMLe”、“MMLy”和“MMU”选择培养基,每种都是用20g/L琼脂制备的)。
或者,在5-氟乳清酸(“FOA”;也称作5-氟尿嘧啶-6羧酸一水合物)选择培养基上选择转化体,该培养基包含:不含硫酸铵或氨基酸的0.17%酵母氮基(DIFCO Laboratories)、2%葡萄糖、0.1%脯氨酸、75mg/L尿嘧啶、75mg/L尿苷、900mg/L FOA(Zymo ResearchCorp.,Orange,CA)和20g/L琼脂。
最后,对于设计用于促进含油性的条件的“两阶段生长条件”,按照如下制备高葡萄糖培养基(“HGM”):14g/L KH2PO4、4g/LK2HPO4、2g/L MgSO4·7H2O、80g/L葡萄糖(pH 6.5)。根据以下方案,在“两阶段生长条件”下培养菌株:首先,30℃下250rpm/min振荡条件下将三份细胞在液体MM中生长48小时。通过离心收集细胞,提取上清液。将沉淀的细胞重悬于HGM中,30℃下250rpm/min振荡条件下生长72小时或96小时。通过离心再次收集细胞,提取上清液。
解脂耶氏酵母的脂肪酸分析
为了进行脂肪酸分析,通过离心收集细胞,并且按照披露于以下文献中的方法提取脂质:Bligh,E.G.& Dyer,W.J.(Can.J.Biochem.Physiol.37:911-917(1959))。脂肪酸甲酯是通过脂质提取物与甲醇钠的脂交换反应制备的(Roughan,G.,and Nishida I.Arch BiochemBiophys.276(1):38-46(1990)),并且随后用装有30-m X 0.25mm(内径)HP-INNOWAX(Hewlett-Packard)柱的Hewlett-Packard 6890GC进行分析。烘箱温度以3.5℃/分钟的速度从170℃(25分钟保持时间)提高到185℃。
为了进行直接的碱酯交换,收获耶氏酵母培养物(3mL),在蒸馏水中洗涤一次,在Speed-Vac中真空干燥5-10分钟。将甲醇钠(100μl,1%)加入样品,然后涡旋并摇动样品20分钟。加入3滴1M NaCl和400μl己烷后,涡旋和离心样品。取出上层,按照上文的描述通过GC进行分析。
实施例1
鉴定用于在解脂耶氏酵母中高表达的启动子
进行了比较研究,研究TEF、GPD、GPDIN、GPM、GPAT、FBA、FBAIN和YAT1启动子的启动子活性,该研究是通过合成包含每种启动子和作为报道基因的编码葡糖醛酸酶(GUS)的大肠杆菌基因的构建体而进行的(Jefferson,R.A.Nature.14(342):837-838(1989))。然后,通过组织化学和荧光测定(Jefferson,R.A.Plant Mol.Biol.Reporter 5:387-405(1987))和/或通过使用实时PCR进行mRNA定量而测量GUS活性。
构建包含嵌合启动子::GUS::XPR基因的质粒
质粒pY5-30(图5A;SEQ ID NO:113)包含:耶氏酵母自主复制序列(ARS18);ColE1质粒复制起点;用于在大肠杆菌中进行选择的氨苄青霉素抗性基因(AmpR);用于在耶氏酵母中进行选择的耶氏酵母LEU2基因;和嵌合TEF::GUS::XPR基因。基于该质粒,建立了一系列质粒,其中用多种其它天然解脂耶氏酵母启动子代替TEF启动子。
通过PCR扩增推定的启动子区,扩增中采用下表11所示引物和基因组解脂耶氏酵母DNA作为模板,或含有克隆到pGEM-T-easyvector(Promega,Madison,WI)载体中的合适DNA区的基因组DNA的片段。
表11
包含嵌合启动子::GUS::XPR基因的质粒的构建
| 启动子 | 引物 | 位置 | RE位点 | 质粒名称 |
| GPD | YL211,YL212(SEQ IDNOs:169和170) | gpd基因的‘ATG’翻译起始位点-968bp(SEQ ID NO:158) | SalI和NcoI | pYZGDG |
| GPDIN | YL376,YL377(SEQ ID NOs:171和172) | gpd基因周围的-973bp至+201bp(从而包括146bp内含子,其中该内含子位于+49bp至+194bp的位置)(SEQ ID NO:159) | PstI/NcoI(用于启动子)和PstI/SalI(用于载体) | pDMW222 |
| GPM | YL203,YL204(SEQ ID NOs:173和174) | gpm基因的‘ATG’翻译起始位点-875bp(SEQ ID NO:160) | NcoI和SalI | pYZGMG |
| GPAT | GPAT-5-1,GPAT-5-2(SEQ ID NOs:175和176) | gpat基因的‘ATG’翻译起始位点-1130bp(SEQ ID NO:164) | SalI和NcoI | pYGPAT-GUS |
| FBA | ODMW314,YL341(SEQ ID NOs:177和178) | Fba基因周围的-1001bp至-1bp(SEQ ID NO:161) | NcoI和SalI | pDMW212 |
| FBAIN | ODMW320,ODMW341(SEQ ID NOs:179和180) | Fba基因周围的-804bp至+169bp(从而包括102bp内含子其中内含子位于+62bp至+165bp的位置)(SEQ ID NO:162) | NcoI和SalI | pDMW214 |
| YAT1 | 27203-F,27203-R(SEQ ID NOs:181和182) | yat1基因周围的-778bp至-1bp(SEQ ID NO:165) | HindIII和SalI;也是NcoI和HindIII | pYAT-GUS |
注:‘ATG’翻译起始密码子的‘A’核苷酸称作+1
按照一般方法中的描述,在50μl总体积中进行GPD、GPDIN、GPM、FBA和FBAIN的各个PCR扩增反应。热循环仪条件设置为35个循环的95℃下1分钟、56℃下30秒和72℃下1分钟,最后,72℃下进行最终延伸10分钟。
用预先混合的2X PCR溶液(TaKaRa Bio Inc.,Otsu,Shiga,520-2193,Japan)的1:1稀释液,在50μl总体积中进行GPAT启动子的PCR扩增。最终的组合物含有25mM TAPS(pH 9.3)、50mMKCl、2mM MgCl2、1mM 2-巯基乙醇、每种三磷酸脱氧核糖核苷酸各200μM、每种引物各10pmole、50ng模板和1.25U TaKaRa ExTaqTM DNA聚合酶(Takara Mirus Bio,Madison,WI)。热循环仪条件设置为30个循环的94℃下2.5分钟、55℃下30秒和72℃下2.5分钟,最后,72℃下进行最终延伸6分钟。
在与上文对GPAT描述的相似组合物中进行YAT1启动子的PCR扩增。首先将反应混合物加热到94℃持续150秒。扩增进行30个循环的94℃下30秒、55℃下30秒和72℃下1分钟,最后,72℃下进行最终延伸7分钟。
用Qiagen PCR纯化试剂盒纯化每种PCR产物,然后用限制酶消化(根据上表,用标准条件),在1%(w/v)琼脂糖中进行凝胶电泳后纯化消化的产物。然后将消化的PCR产物(除来自YAT1的那些)连接到相似消化的pY5-30载体中。然后将来自每个反应的连接的DNA用于分别转化大肠杆菌Top10、大肠杆菌DH10B或大肠杆菌DH5α。在含有氨苄青霉素(100μg/mL)的LB琼脂上选择转化体。
YAT1在克隆到pY5-30中之前,需要额外的操作。具体地,用HindIII和SalI消化YAT1 PCR产物后,得到了约600bp的片段;用NcoI和HindIII消化后,得到了约200bp的片段。分离和纯化这两种产物。然后,用SalI和NcoI消化质粒pYGPAT-GUS,分离和纯化约9.5kB的片段。将三种DNA片段连接在一起,得到pYAT-GUS。
分析来自每个转化反应的质粒,证明了预期的质粒的存在。这些质粒命名如下:pYZGDG(包含GPD::GUS::XPR嵌合基因),pDMW222(包含GPDIN::GUS::XPR嵌合基因),pYZGMG(包含GPM::GUS::XPR嵌合基因),pYGPAT-GUS(包含GPAT::GUS::XPR嵌合基因),pDMW212(包含FBA::GUS::XPR嵌合基因),pDMW214(包含FBAIN::GUS::XPR嵌合基因)和pYAT-GUS(包含YAT1::GUS::XPR嵌合基因)。
按照一般方法中的描述,将上述质粒,以及额外的质粒pY5-30(包含TEF::GUS::XPR嵌合基因)分别转化到解脂耶氏酵母中。解脂耶氏酵母宿主是解脂耶氏酵母ATCC #76982或解脂耶氏酵母ATCC#20362,菌株Y2034(下文[实施例4],能够通过去饱和酶/Δ6延伸酶途径生产10% ARA)。将所有转化的细胞铺板到缺乏亮氨酸的基本培养基中,在30℃下维持2-3天。
通过GUS表达的组织化学分析对耶氏酵母启动子进行的比较分析
使含有质粒pY5-30、pYZGDG、pYZGMG、pDMW212和pDMW214的解脂耶氏酵母ATCC #76982菌株30℃下在3mL MM中从单菌落生长到OD600为约1.0。然后,通过离心收集100μl细胞,重悬于100μl组织化学染色缓冲液中,30℃下温育。将5mg 5-溴-4-氯-3-吲哚基葡糖苷酸(X-Gluc)溶解于50μl二甲基甲酰胺中,然后加入5mL 50mM NaPO4,pH 7.0,以制备染色缓冲液。组织化学染色的结果(图5B)表明,构建体pY5-30中的TEF启动子、构建体pYZGDG中的GDP启动子、构建体pYZGMG中的GPM启动子、构建体pDMW212中的FBA启动子、构建体pDMW214中的FBAIN启动子都是有活性的。FBA和FBAIN启动子似乎都比其它启动子强得多,FBAIN启动子具有最强的启动子活性。
在一个独立的实验中,使含有质粒pY5-30、pYGPAT-GUS、pYAT-GUS和pDMW214的解脂耶氏酵母Y2034菌株30℃下在5mLSD培养基中从单菌落生长到OD600为约8.0。然后,通过离心收集1mL细胞。离心剩余的培养物,用HGM洗涤两次,分别重悬于5mL HGM中,在30℃下进一步生长。24和120小时后,离心约0.25mL的每种培养物,收集细胞。将细胞样品分别重悬于100μl组织化学染色缓冲液(参见上文)中。在每个样品中加入消解酶20T(5μl,1mg/mL;ICN Biomedicals,Costa Mesa,CA),30℃下温育混合物。
组织化学染色的结果显示,当在SD培养基中生长24小时,构建体pYGPAT-GUS中的GPAT启动子是有活性的,构建体pYAT-GUS中的YAT1启动子也是有活性的(图5C,“在SD培养中24hr”)。比较而言,GPAT启动子似乎比TEF启动子强得多,并且相对于FBAIN启动子的活性减少。同样,当细胞在SD培养基中生长24小时,YAT1启动子似乎比TEF启动子强得多,但比FBAIN启动子和GPAT启动子明显更弱。然而,更有趣的是,发现在HGM中生长24小时的细胞中,YAT1启动子比GPAT启动子强,并且与FBAIN启动子相似(图5C,“在HG培养基中24小时”)。在HGM中120小时后依然如此(图5C,“在HG培养基中120小时”)。因此,YAT1启动子在HGM中似乎被诱导,HGM是由于氮限制而促进含油生长条件的培养基。
通过GUS表达的荧光测定对解脂耶氏酵母启动子进行的比较分析
通过荧光测定相应的底物β-葡糖苷酸对4-甲基伞形酮(4-MU)的生产(Jefferson,R.A.Plant Mol.Biol.Reporter 5:387-405(1987)),也测定了GUS活性。
使含有质粒pY5-30、pYZGDG、pYZGMG、pDMW212和pDMW214的解脂耶氏酵母ATCC #76982菌株30℃下在3mL MM中从单菌落生长到OD600为约1.0(按照上文的描述)。然后,将各3mL培养物加入含有50mL MM的500mL烧瓶中,在振荡孵育器中30℃下生长约24小时。通过离心收集细胞,重悬于Promega细胞裂解缓冲液中,用BIO 101 Biopulverizer system(Vista,CA)裂解。离心后,除去上清液,放置在冰上。
类似地,将含有质粒pY5-30、pYAT-GUS、pYGPAT-GUS和pDMW214构建体的解脂耶氏酵母菌株Y2034 30℃下在10mL SD培养基中生长48小时,从单菌落生长到OD600为约5.0。收集每种培养物各2mL,用于按照下文所述进行GUS活性测定,而将每种培养物各5ML转换到HGM中。
具体地,通过离心收集来自5mL等分物的细胞,用5mL HGM洗涤一次,重悬于HGM中。然后,将HGM中的培养物在振荡孵育器中30℃下生长24小时。收集每种HGM培养物各2mL用于GUS活性测定,而使剩余的培养物再生长额外的96小时,然后收集每种培养物的额外的2mL用于测定。
将每份2mL SD培养基中的培养样品重悬于1ML 0.5X细胞培养物裂解试剂(Promega)中。将重悬的细胞在配备橡胶O形环的2.0mL螺帽试管中与0.6mL玻璃珠(0.5mm直径)混合。然后在Biospec minibeadbeater(Bartlesville,OK)中,以最高的设置将细胞匀浆90秒。14,000rpm下将匀浆混合物在Eppendof离心管中离心2分钟,以除去细胞碎屑和珠子。将上清液用于GUS测定和蛋白测定。
为了进行每个荧光测定,将100μl提取物加入700μlGUS测定缓冲液(2mM 4-甲基伞形酮基-β-D-葡糖苷酸(“MUG”),溶于提取缓冲液中)或将200μl提取物加入800μl GUS测定缓冲液中。将混合物置于37℃下。然后在0、30和60分钟的时间点取出100μl的等分物,加入900μl终止缓冲液(1M Na2CO3)。用CytoFluor Series4000 Fluorescence Multi-Well平板读数器(PerSeptive Biosystems,Framingham,MA)对每个时间点进行读数,所述读数器设置为激发波长是360nm,而发射波长是455nm。用10μl提取物和200μl BioRadBradford试剂或20μl提取物和980μl BioRad Bradford试剂确定每个样品的总蛋白浓度(Bradford,M.M.Anal.Biochem.72:248-254(1976))。GUS活性表示为每分钟每mg蛋白的4-MU纳摩尔数。
设计将这些荧光测定的结果用于比较解脂耶氏酵母ATCC #76982菌株中的TEF、GPD、GPM、FBA和FBAIN启动子,如图6A所示。具体地,FBA启动子在解脂耶氏酵母中比GPD强2.2倍。此外,FBAIN启动子的GUS活性比GPD启动子强约6.6倍。
这些设计用于比较解脂耶氏酵母菌株Y2034中的TEF、GPAT、YAT1和FBAIN启动子的荧光测定的结果如下表所示。
表12
各种生长条件下TEF、FBAIN、YAT1和GPAT启动子活性的比较
基于上述数据(其中对YAT1启动子活性的定量是基于细胞提取物的GUS活性),当细胞从SD培养基转换到HGM中并且生长24小时后,YAT1启动子的活性增加约37倍。在HGM中120小时后,活性一定程度地减少,但仍然比在SD培养基中的活性高25倍。相反,当从SD培养基转换到HGM中24小时后,FBAIN启动子和GPAT启动子的活性分别减少30%和40%。TEF启动子的活性在HGM中24小时后增加2.3倍。因此,YAT1启动子在含油条件下是可诱导的。
通过GUS表达的定量PCR分析对耶氏酵母启动子进行的比较分析
通过定量PCR分析,确定含有pY5-30、pYZGDG、pDMW222、pDMW212和pDMW214构建体的解脂耶氏酵母中TEF、GPD、GPDIN、FBA和FBAIN启动子的转录活性。这要求分离RNA和实时RT-PCR。
更具体地,使含有pY5-30、pYZGDG、pDMW222、pDMW212和pDMW214构建体的解脂耶氏酵母ATCC #76982菌株在25mLErlenmeyer烧瓶中的6mL MM中30℃下从单菌落生长16小时。然后,将每种各6mL的起始培养物加入各个含有140mL HGM的500mL烧瓶中,30℃下温育4天。以24小时的每个间隔,从每个烧瓶取出每种培养物各1mL,以测量光密度,取出27mL,用于进行荧光测定(如上文的描述),取出两个1.5mL的等分物,进行RNA分离。对RNA分离的培养物进行离心,产生细胞沉淀。
根据改进的Qiagen RNeasy mini方案(Qiagen,San Diego,CA),从耶氏酵母菌株分离RNA。简言之,在每个样品的每个时间点,用340μL Qiagen’s缓冲液RLT重悬两种细胞沉淀的每一种。将来自两个试管的每一个试管的缓冲液RLT/细胞悬浮液混合物在珠子搅拌试管(Bio101,San Diego,CA)中混合。将0.5mL玻璃珠中的大约500μL加入试管中,通过在设置5(BioPulverizer,Bio101 Company,SanDiego,CA)进行珠子搅拌2分钟,破碎细胞。然后通过在14,000rpm离心1分钟而沉淀破碎的细胞,将350μl上清液转移到新的微量离心管中。将乙醇(350μL 70%)加入每种匀浆的裂解物。轻柔搅拌后,将整个样品加入2mL收集管中的Rneasy微型柱。将样品以10,000rpm离心15秒。将缓冲液RW1(350μL)加入Rneasy微型柱,将柱子以10,000rpm离心15秒,以洗涤细胞。弃去洗脱液。将Qiagen’s DNase1储液(10μL)加入70μl缓冲液RDD,轻柔混合。将完整的DNA酶溶液加入Rneasy微型柱,室温下温育15分钟。温育步骤后,将350μL缓冲液RW1加入微型柱,将柱子以10,000rpm离心15秒。将柱子用700μL缓冲液RW1洗涤两次。将不含RNA酶的水(50μL)加入柱子。以10,000rpm将柱子离心1分钟,以洗脱RNA。
采用两步RT-PCR方案,其中首先将耶氏酵母总RNA转化为cDNA,然后用实时PCR分析cDNA。用Applied Biosystems’HighCapacity cDNA Archive试剂盒(PN#4322171;Foster City,CA)和来自MediaTech,Inc.(PN# 46-000-Con;Holly Hill,FL)的分子生物级水进行向cDNA的转化。通过与10μl RT缓冲液、4μl 25XdNTPs、10μl 10X Random Hexamer引物、5μl Multiscribe逆转录酶和0.005μl RNA酶混合,并且用水将中反应体积补充为100μl,将来自耶氏酵母的总RNA(100ng)转化为cDNA。在热循环仪中温育反应,25℃下10分钟,然后是37℃下2小时。将cDNA在-20℃储存,然后进行实时分析。
用来自Applied Biosystems(PN# 4309155)的SYBR Green PCR主混合物进行实时分析。将逆转录反应物(2μl)加入10μl 2X SYBRPCR混合物、0.2μl 100μM针对URA(即引物YL-URA-16F和YL-URA-78R[SEQ ID NOs:198和199])或GUS(即引物GUS-767F和GUS-891R[SEQ ID NO:200和201])的正向和反向引物和7.2μl水。使反应在95℃下热循环10分钟,然后在ABI 7900 Sequence DetectionSystem仪器中进行40个循环的95℃下5秒和60℃下1分钟。在每个周期,在60℃延伸过程中收集实时荧光数据。
按照用户手册#2:“Relative Quantitation of Gene Expression”,Applied Biosystems,2001年10月,用ΔΔCT方法进行相对定量。将URA基因用于对GUS表达进行标准化。为了证实将URA用作标准化基因,比较GUS和URA的PCR效率,发现它们分别是1.04和0.99(其中1.00等于100%效率)。由于PCR效率都接近于100%,证实了将URA作为GUS表达的标准化物,以及将ΔΔCT方法用于表达定量。标准化的量称作ΔCT。
将每种不同菌株(即含有pYZGDG、pDMW222、pDMW212和pDMW214构建体的解脂耶氏酵母ATCC #76982菌株)中的GUSmRNA定量到含有pY5-30(TEF::GUS)的菌株的mRNA水平。因此,用含有TEF::GUS的菌株的mRNA水平作为参照样品,计算表达的相对定量。将GPD::GUS、GPDIN::GUS、FBA::GUS和FBAIN::GUS的标准化的值与TEF::GUS参照的标准化值进行比较。这种量称作ΔΔCT。然后利用公式2-ΔΔCT,将ΔΔCT值转化为绝对值。这些值表示包含嵌合GPD::GUS、GPDIN::GUS、FBA::GUS和FBAIN::GUS基因的菌株相对于包含嵌合TEF::GUS基因的菌株中的GUS的mRNA水平的增加倍数。采用该方法,可以比较TEF启动子的活性与GPD、GPDIN、FBA和FBAIN启动子的活性。
每种GUS嵌合基因的mRNA的相对定量结果示于图6B。更具体地,该测定显示,在HGM中24小时后,FBA和FBAIN启动子的转录活性分别比TEF启动子强大约3.3和6倍。类似地,GPD和GPDIN启动子的转录活性分别比TEF启动子强大约2和4.4倍。尽管FBA::GUS、FBAIN::GUS、GPD::GUS和GPDIN::GUS基因融合体的转录活性在4天的实验期中减少,FBAIN和GPDIN启动子的转录活性在实验的最后一天仍然比TEF启动子强大约3和2.6倍。
实施例2
鉴定用于增加解脂耶氏酵母中的基因转录的增强子
基于FBAIN和GPDIN的强启动子活性(其中活性分别大于FBA和GPD启动子的活性)和每个启动子区内的内含子的鉴定,进行了目前的工作,以确定每个内含子中是否存在增强子。
具体地,制备了由GPM::FBAIN启动子融合体和GPM::GPDIN启动子融合体组成的两个嵌合启动子,以驱动GUS报道基因的表达。嵌合启动子(包含“成分1”和“成分2”)描述于下表13。
表13
包含嵌合启动子::GUS::XPR基因内的嵌合启动子的质粒的构建
| 嵌合启动子 | 成分1 | 成分2 | 质粒名称 |
| GPM::FBAIN(SEQ ID NO:167) | GPM的-1bp至-843bp区 | FBAIN的+1bp至+171bp区其中内含子位于+62bp至+165bp的位置 | pDMW224 |
| GPM::GPDIN(SEQ ID NO:168) | GPM的-1bp至-843bp区 | GPDIN的+1bp至+198bp区其中内含子位于+49bp至+194bp的位置 | pDMW225 |
嵌合启动子的位置使每一种嵌合启动子都驱动质粒pDMW224和pDMW225中的GUS报道基因的表达。
通过基于组织化学测定(如实施例1的描述)比较包含pDMW224和pDMW225的解脂耶氏酵母菌株中的GUS活性与包含pY5-30、pYZGDG、pYZGMG和pDMW214构建体的解脂耶氏酵母菌株中的GUS活性,将GPM::FBAIN启动子和GPM::GPDIN启动子的活性与TEF、FBAIN、GPDIN和GPM启动子的活性进行比较。如上文所确定的,FBAIN启动子是最强的启动子。但是,嵌合GPM::FBAIN启动子和嵌合GPM::GPDIN启动子都比GPM启动子强得多,并且似乎活性与GPDIN启动子相同。因此,证明了在GPDIN和FBAIN启动子中都存在增强子。
本领域技术人员能够容易地用GPDIN内含子或FBAIN内含子构建相似的嵌合启动子。
实施例3
磺酰脲选择
由于缺乏合适的非抗生素选择转化标记,阻碍了耶氏酵母的遗传改进。本实施例描述了基于磺酰脲抗性开发显性的、解脂耶氏酵母非抗生素标记,其也可以遗传上用于工业酵母菌株,这些菌株可以是单倍体、二倍体、非整倍体或杂合的。
理论和最初的敏感性筛选
乙酰羟酸合酶(AHAS)是生物合成支链氨基酸的途径中最常见的酶。它是磺酰脲和咪唑烷酮除草剂的靶。因此,在微生物和植物中都报道了磺酰脲除草剂抗性。例如,在啤酒糖酵母中,AHAS中的单个W586L突变赋予对磺酰脲除草剂的抗性(Falco,S.C.,et al.,Dev.Ind.Microbiol.30:187-194(1989);Duggleby,R.G.,et.al.Eur.J.Biochem.270:2895(2003))。
当对野生型AHAS解脂耶氏酵母(GenBank获取号XP_501277)和啤酒糖酵母(GenBank获取号P07342)酶的氨基酸序列进行比对时,啤酒糖酵母酶的586位的Trp氨基酸残基与解脂耶氏酵母酶的497位的Trp残基相同。因此,如果野生型细胞自身对磺酰脲敏感,则认为解脂耶氏酵母酶中的W497L突变可能赋予磺酰脲除草剂抗性。采用本领域技术人员公知的方法,确定基本培养基中浓度为100μg/mL的磺酰脲(chlorimuron ethyl)足够抑制野生型解脂耶氏酵母菌株ATCC #20362和ATCC #90812的生长。
突变的W497L AHAS基因的合成
在两步反应中从基因组DNA建立包含W497L突变的解脂耶氏酵母AHAS基因(SEQ ID NO:280)。首先,用Pfu UltraTM High-FidelityDNA聚合酶(Stratagene,目录号600380)和引物410和411[SEQ IDNOs:365和366]从基因组DNA扩增AHAS基因的5’部分;类似地用引物412和413[SEQ ID NOs:367和368]扩增基因的3’部分。两对引物是重叠的,使得重叠区含有W497L突变(其中该突变是‘CT’改变为‘TG’)。
对大小正确的5’和3’PCR产物进行凝胶纯化,并且用作第二轮PCR的模板,其中用引物414和415(SEQ ID NOs:369和370)以及来自两个主要PCR反应的产物的混合物扩增完整的突变基因。这种突变的基因携带其自身的天然启动子和终止子序列。凝胶纯化具有正确大小的第二轮PCR产物,用酶SalI/BsiWI消化后,通过框内融合技术克隆到质粒pY35[含有嵌合TEF::串珠镰孢Δ12去饱和酶(Fm2)基因、大肠杆菌复制起点、细菌氨苄青霉素抗性基因、耶氏酵母Leu2基因和耶氏酵母自主复制序列(ARS);其它细节参见WO2005/047485]的载体主链中。框内融合反应混合物转化到TOP10感受态细胞(Invitrogen,目录号C4040-10)中。在LB/Amp平板上选择1天后,通过DNA微量制备分析八(8)个菌落。通过限制酶消化,证明7个克隆是正确的。其中一个含有磺酰脲抗性基因和LEU基因的克隆命名为“pY57”(或“pY57.Yl.AHAS.w4971”;图7A)。
通过标准醋酸锂方法用pY57和‘空’LEU转化野生型解脂耶氏酵母菌株ATCC #90812和#20362。也利用了包含‘No-DNA’的转化对照。将转化体铺板到MM+磺酰脲(SU;100μg/mL)琼脂平板上,生长4天后评估菌落是否存在。
表14
解脂耶氏酵母中的AHAS选择
基于上述结果,AHAS W497L是解脂耶氏酵母ATCC #90812和#20362中的良好非抗生素选择标记。随后,申请人采用了150μg/mL的磺酰脲浓度。这种新的标记对于转化解脂耶氏酵母是有利的,因为它不依赖于外源基因,而是依赖于突变的天然基因,并且它不需要营养缺陷,也不导致营养缺陷。除草剂对于人和动物无毒。
如果以类似此处所述的方法建立突变的AHAS酶,预期该选择方法可以普遍适用于其它工业酵母菌株,它们可能是单倍体、二倍体、非整倍体或杂合的。
实施例4
Δ6去饱和酶/Δ6延伸酶途径:制备生产占总脂质的约10-11%的ARA的Y2034和Y2047菌株
本实施例描述了来源于解脂耶氏酵母ATCC #20362的Y2034和Y2047菌株的构建,所述菌株分别能够生产占总脂质的约10和11%的ARA(图4)。这些菌株经过工程化,表达Δ6去饱和酶/Δ6延伸酶途径。因此,预期菌株Y2034和Y2047的完整脂质谱表明共合成大约25-29%GLA。
菌株Y2034和Y2047的开发首先需要构建菌株M4(生产8%DGLA)。
制备生产占总脂质的约8%的DGLA的M4菌株
制备构建体pKUNF12T6E(图7B;SEQ ID NO:114),将4种嵌合基因(包含Δ12去饱和酶、Δ6去饱和酶和两种C18/20延伸酶)整合到野生型耶氏酵母菌株ATCC #20362的Ura3基因座,从而能够生产DGLA。pKUNF12T6E质粒含有以下成分:
表15
质粒pKUNF12T6E(SEQ ID NO:114)的说明
| SEQ ID NO:114内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| AscI/BsiWI(9420-8629) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的784bp 5’部分 |
| SphI/PacI(12128-1) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的516bp 3’部分 |
| SwaI/BsiWI(6380-8629) | FBAIN::EL1S::Pex20,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●EL1S:密码子优化的延伸酶1基因(SEQ IDNO:19),来源于高山被孢霉(GenBank获取号AX464731)●Pex20:Pex20终止子序列,来源于耶氏酵母Pex20基因(GenBank获取号AF054613) |
| BglII/SwaI(4221-6380) | TEF::Δ6S::Lip1,包含:●TEF:TEF启动子(GenBank获取号AF054508)●Δ6S:密码子优化的Δ6去饱和酶基因(SEQ IDNO:3),来源于高山被孢霉(GenBank获取号AF465281)●Lip1:Lip1终止子序列,来源于耶氏酵母Lip1基因(GenBank获取号Z50020) |
| PmeI/ClaI(4207-1459) | FBA::F.Δ12::Lip2,包含:●FBA:FBA启动子(SEQ ID NO:161)●F.Δ12:串珠镰孢Δ12去饱和酶基因(SEQ IDNO:27)●Lip2:Lip2终止子序列,来源于耶氏酵母Lip2基因(GenBank获取号AJ012632) |
| ClaI/PacI(1459-1) | TEF::EL2S::XPR,包含:●TEF:TEF启动子(GenBank获取号AF054508)●EL2S:密码子优化的延伸酶基因(SEQ ID NO:22),来源于金黄色破囊壶菌(U.S.6,677,145)●XPR:耶氏酵母Xpr基因(GenBank获取号M17741)的3’区的约100bp |
用AscI/SphI消化pKUNF12T6E质粒,然后用于根据一般方法转化野生型解脂耶氏酵母ATCC #20362。将转化的细胞铺板到FOA选择培养基平板上,在30℃维持2-3天。挑选FOA抗性菌落,划线到MM和MMU选择平板上。能够在MMU平板上生长但是不能在MM平板上生长的菌落选为Ura-菌株。然后30℃下将Ura-菌株的单菌落接种到液体MMU中,以250rpm/min振荡2天。
通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示DGLA存在于含有pKUNF12T6E的4种嵌合基因的转化体中,但是不存在于野生型耶氏酵母对照菌株中。选择的32个Ura-菌株中的大多数产生占总脂质的约6%的DGLA。存在两种产生占总脂质的约8%的DGLA的菌株(即菌株M4和13-8)。
制备生产占总脂质的约10%的ARA的Y2034和Y2047菌株
制备构建体pDMW232(图7C;SEQ ID NO:115)和pDMW271(图7D;SEQ ID NO:116),分别将两种Δ5嵌合基因整合到耶氏酵母菌株M4的Leu2基因。质粒pDMW232和pDMW271分别含有以下成分,如表16和17的描述:
表16
质粒pDMW232(SEQ ID NO:115)的说明
| SEQ ID NO:115内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| AscI/BsiWI(5550-4755) | 耶氏酵母Leu2基因(GenBank获取号AF260230)的788bp 5’部分 |
| SphI/PacI(8258-8967) | 耶氏酵母Leu2基因(GenBank获取号AF260230)的703bp 3’部分 |
| SwaI/BsiWI(2114-4755) | FBAIN::MAΔ5::Pex20,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●MAΔ5:高山被孢霉Δ5去饱和酶基因(SEQ IDNO:6)(GenBank获取号AF067654)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| SwaI/ClaI(2114-17) | TEF::MAΔ5::Lip1,包含:●TEF:TEF启动子(GenBank获取号AF054508)●MAΔ5:SEQ ID NO:6(参见上文)●Lip1:耶氏酵母Lip1基因(GenBank获取号Z50020)的Lip1终止子序列 |
| PmeI/CIaI(5550-4755) | 耶氏酵母Ura3基因(GenBank获取号AJ306421) |
表17
质粒pDMW271(SEQ ID NO:116)的说明
| SEQ ID NO:116内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| AscI/BsiWI(5520-6315) | 耶氏酵母Leu2基因(GenBank获取号AF260230)的788bp 5’部分 |
| SphI/PacI(2820-2109) | 耶氏酵母Leu2基因(GenBank获取号AF260230)的703bp 3’部分 |
| SwaI/BsiWI(8960-6315) | ●FBAIN::MAΔ5::Pex20,如对pDMW232的描述(上文) |
| SwaI/ClaI(8960-11055) | ●TEF::MAΔ5::Lip1,如对pDMW232的描述(上文) |
| PmeI/ClaI(12690-11055) | 耶氏酵母Ura3基因(GenBank获取号AJ306421) |
| ClaI/PacI(1-2109) | TEF::HΔ5S::Pex16,包含:●TEF:TEF启动子(GenBank获取号AF054508)●HΔ5S:密码子优化的Δ5去饱和酶基因(SEQID NO:13),来源于人(GenBank获取号NP_037534)●Pex16:耶氏酵母Pex16基因(GenBank获取号U75433)的Pex16终止子序列 |
用AscI/SphI消化质粒pDMW232和pDMW271,然后用于根据一般方法转化菌株M4。转化后,将细胞铺板到MMLe平板上,在30℃维持2-3天。挑选在MMLe平板上生长的来自每次转化的各个菌落,划线到MM和MMLe平板上。能够在MMLe平板上生长但是不能在MM平板上生长的菌落选为Leu2-菌株。然后30℃下将Leu2-菌株的单菌落接种到液体MMLe中,以250rpm/min振荡2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示pDMW232和pDMW271转化体中存在ARA,但亲本M4菌株中不存在。具体地,在48个选择的具有pDMW232的Leu2-转化体中,34个菌株生产占工程化的耶氏酵母中的总脂质的5%以下的ARA,11个菌株生产6-8%的ARA,3个菌株生产约10%的ARA。生产10% ARA的菌株之一命名为“Y2034”。
同时,在48个选择的具有pDMW271的Leu2-转化体中,35个菌株生产占工程化的耶氏酵母中的总脂质的5%以下的ARA,12个菌株生产6-8%的ARA,1个菌株生产约11%的ARA。生产11% ARA的菌株命名为“Y2047”。
实施例5
制备具有Ura-基因型并且生产占总脂质的45%的LA的中间菌株Y2031
通过将质粒pKUNT2(图8A)的TEF::Y.Δ12::Pex20嵌合基因整合到野生型耶氏酵母菌株ATCC #20362的Ura3基因座中,制备菌株Y2031,从而产生Ura-基因型。
具体地,质粒pKUNT2含有以下成分:
表18
质粒pKUNT2的说明(SEQ ID NO:117)
| SEQ ID NO:117内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| AscI/BsiWI(3225-3015) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的784bp 5’部分 |
| SphI/PacI(5933-13) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的516bp 3’部分 |
| EcoRI/BsiWI(6380-8629) | TEF::Y.Δ12::Pex20,包含:●TEF:TEF启动子(GenBank获取号AF054508)●Y.Δ12:耶氏酵母Δ12去饱和酶基因(SEQ IDNO:23)●Pex20:来自耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子 |
根据一般方法,用AscI/SphI消化质粒pZKUT2,然后用于转化野生型解脂耶氏酵母ATCC #20362。将转化的细胞铺板到FOA选择培养基平板上,在30℃维持2-3天。挑选了FOA抗性菌落,划线到MM和MMU选择平板上。能够在MMU平板上生长,但是不能在MM平板上生长的菌株选作Ura-菌株。然后30℃下将Ura-菌株的单菌落(5个)单独接种到液体MMU中,以250rpm/min振荡2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard6890GC进行分析。
GC分析显示在两个Ura-菌株(即2号和3号菌株)中含有约45%LA,而野生型ATCC #20362中含有约20% LA。2号转化菌株称作菌株“Y2031”。
实施例6
密码子优化的Δ9延伸酶基因在解脂耶氏酵母中的合成和功能性表达
对球等鞭金藻的Δ9延伸酶基因(GenBank获取号AF390174)的密码子选择进行优化,以便在解脂耶氏酵母中表达,该优化的方式类似于WO 2004/101753中的描述。具体地,根据耶氏酵母密码子使用模式、ATG翻译起始密码子周围的共有序列和RNA稳定性的一般规则(Guhaniyogi,G.and J.Brewer,Gene 265(1-2):11-23(2001)),基于球等鞭金藻基因的DNA序列(SEQ ID NO:39),设计了密码子优化的Δ9延伸酶基因(SEQ ID NO:41)。除了修饰翻译起始位点,还修饰了792bp编码区的126bp,优化了123个密码子。密码子优化的基因中的修饰都没有改变编码的蛋白的氨基酸序列(GenBank获取号AF390174;SEQ ID NO:40)。
用于耶氏酵母的密码子优化的Δ9延伸酶基因的体外合成
按照下文合成了密码子优化的Δ9延伸酶基因。首先,设计了8对寡核苷酸,以延伸密码子优化的球等鞭金藻Δ9延伸酶基因的编码区的全长(如IL3-1A、IL3-1B、IL3-2A、IL3-2B、IL3-3A、IL3-3B、IL3-4A、IL3-4B、IL3-5A、IL3-5B、IL3-6A、IL3-6B、IL3-7A、IL3-7B、IL3-8A和IL3-8B、相应于SEQ ID NOs:187-202)。每对有义(A)和反义(B)寡核苷酸都是互补的,例外的是每个5’末端的4bp突出端。此外,分别在引物IL3-1F、IL3-4R、IL3-5F和IL3-8R(SEQ IDNOs:203-206)中引入了NcoI、PstI、PstI和Not1限制位点,用于随后进行亚克隆。
37℃下在含有50mM Tris-HCl(pH 7.5)、10mM MgCl2、10mMDTT、0.5mM亚精胺、0.5mM ATP和10U T4多核苷酸激酶的20μl体积中对每种寡核苷酸(100ng)进行磷酸化。混合每对有义和反义寡核苷酸,采用以下参数在热循环仪中退火:95℃(2min),85℃(2min),65℃(15min),37℃(15min),24℃(15min)和4℃(15min)。因此,将IL3-1A(SEQ ID NO:187)与IL3-1B(SEQID NO:188)退火,产生双链产物“IL3-1AB”。类似地,将IL3-2A(SEQ ID NO:189)与IL3-2B(SEQ ID NO:190)退火,产生双链产物“IL3-2AB”等。
然后将退火的双链寡核苷酸的两个独立的集合连接到一起,如下所示:集合1(包含IL3-1AB、IL3-2AB、IL3-3AB和IL3-4AB);和集合2(包含IL3-5AB、IL3-6AB、IL3-7AB和IL3-8AB)。在20μl的体积中将每个退火的寡核苷酸的集合与10U的T4 DNA连接酶混合,16℃下温育连接反应。
然后将每个连接反应的产物用作模板,通过PCR扩增设计的DNA片段。具体地,采用连接的“集合1”混合物(即IL3-1AB、IL3-2AB、IL3-3AB和IL3-4AB)作为模板,寡核苷酸IL3-1F和IL3-4R(SEQ IDNOs:203和204)作为引物,通过PCR扩增密码子优化的Δ9延伸酶基因的第一部分。按照一般方法的描述,在50μl总体积中进行PCR扩增。按照如下方法进行扩增:最初在95℃变性3分钟,然后进行35个循环的以下反应:95℃ 1分钟,56℃ 30秒,72℃ 40秒。72℃进行最终的延伸循环10分钟,然后在4℃终止反应。将417bp PCR片段亚克隆到pGEM-T easy载体(Promega)中,得到pT9(1-4)。
采用连接的“集合2”混合物(即IL3-5AB、IL3-6AB、IL3-7AB和IL3-8AB)作为模板,寡核苷酸IL3-5F和IL3-8R(SEQ ID NOs:205和206)作为引物,通过PCR类似地扩增密码子优化的Δ9延伸酶基因的第二部分,克隆到pGEM-T-easy载体中,得到pT9(5-8)。
分别用pT9(1-4)和pT9(5-8)转化大肠杆菌,从氨苄青霉素抗体转化体中分离质粒DNA。纯化质粒DNA,用合适的限制内切酶消化,以释放pT9(1-4)的417bp NcoI/PstI片段(SEQ ID NO:207)和pT9(5-8)的377bp PstI/Not1片段(SEQ ID NO:208)。然后混合这两个片段,按照方向与Nco1/Not1消化的pZUF17(SEQ IDNO:118;图8B)连接在一起,得到pDMW237(图8C;SEQ ID NO:119)。pDMW237中得到的合成Δ9延伸酶基因(“IgD9e”)的DNA序列与最初针对耶氏酵母设计的密码子优化的基因(即SEQ ID NO:41)完全相同。
密码子优化的Δ9延伸酶基因在解脂耶氏酵母中的表达
按照一般方法,将构建体pDMW237(图8C)转化到解脂耶氏酵母Y2031菌株中(实施例4),pDMW237是一种包含FBAIN::IgD9e::Pex20嵌合基因的自主复制质粒。使具有pDMW237的三种Y2031转化体单独在MM培养基中生长2天,通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示在具有pDMW237的这些转化体中分别生产了7.1%、7.3%和7.4% EDA。这些数据证明合成的、密码子优化的IgD9e能够将C18:2转化为EDA。密码子优化的基因的“底物转化百分比”确定为大约13%。
实施例7
在解脂耶氏酵母中合成密码子优化的Δ8去饱和酶基因
按照与WO 2004/101753和实施例6(上文)描述的相似方式,对纤细眼虫的Δ8去饱和酶基因(GenBank获取号AAD45877)的密码子选择进行了优化,以便在耶氏酵母中表达。尽管合成了三种不同的密码子优化的基因(即“D8S-1”、“D8S-2”和“D8S-3”),这些基因都不能将EDA去饱和为DGLA。因此,假定以前公开的Δ8去饱和酶序列都不正确,必须按照mRNA分离、cDNA合成和PCR直接从纤细眼虫分离Δ8去饱和酶。这得到了两个相似的序列,在此鉴定为Eg5(SEQ ID NOs:44和45)和Eg12(SEQ ID NOs:46和47)。
通过将基因克隆到啤酒糖酵母酵母表达载体并进行底物补料试验而进行每种基因序列的功能分析。尽管Eg5和Eg12都能够使EDA和EtrA去饱和,分别得到DGLA和ETA,但Eg5相对于Eg12具有显著更高的活性。
基于证实的Eg5的Δ8去饱和酶活性,为在解脂耶氏酵母中表达而对序列进行了密码子优化,从而导致合成了合成的功能性的密码子优化的Δ8去饱和酶,命名为“D8SF”(SEQ ID NOs:48和49)。
密码子优化的Δ8去饱和酶基因的初步体外合成
根据耶氏酵母密码子使用模式、“ATG”翻译起始密码子周围的共有序列和RNA稳定性的一般规则(Guhaniyogi,G.and J.Brewer,Gene 265(1-2):11-23(2001)),基于公开的纤细眼虫的序列(SEQID NOs:42和43),设计了密码子优化的Δ8去饱和酶基因(命名为“D8S-1”;SEQ ID NO:209)。除了修饰翻译起始位点,还修饰了1260bp编码区的200bp(15.9%)。除了第二个氨基酸从“K”变为“E”,从而在翻译起始密码子周围添加了NcoI位点,密码子优化的基因中的修饰都没有改变编码的蛋白的氨基酸序列(SEQ ID NO:43)。
具体地,按照下文合成了密码子优化的Δ8去饱和酶基因。首先,设计了13对寡核苷酸,以延伸密码子优化的纤细眼虫Δ8去饱和酶基因的编码区的全长(如D8-1A、D8-1B、D8-2A、D8-2B、D8-3A、D8-3B、D8-4A、D8-4B、D8-5A、D8-5B、D8-6A、D8-6B、D8-7A、D8-7B、D8-8A、D8-8B、D8-9A、D8-9B、D8-10A、D8-10B、D8-11A、D8-11B、D8-12A、D8-12B、D8-13A和D8-13B、相应于SEQ ID NOs:210-235)。每对有义(A)和反义(B)寡核苷酸都是互补的,例外的是每个5’末端的4bp突出端。此外,分别在引物D8-1A、D8-3B、D8-7A、D8-9B和D8-13B(SEQ ID NOs:210、215、222、227和235)中引入了NcoI、BglII、Xho1、SacI和Not1限制位点,用于随后进行亚克隆。
按照实施例6的描述对寡核苷酸(每种100ng)进行磷酸化,然后混合每对有义和反义寡核苷酸,并且退火到一起[如D8-1A(SEQ IDNO:210)退火于D8-1B(SEQ ID NO:211),得到双链产物“D8-1AB”和D8-2A,(SEQ ID NO:212)退火于D8-2B(SEQ ID NO:213),得到双链产物“D8-2AB”等]。
然后将退火的双链寡核苷酸的4个独立的集合连接到一起,如下所示:集合1(包含D8-1AB、D8-2AB和D8-3AB);集合2(包含D8-4AB、D8-5AB和D8-6AB);集合3(包含D8-7AB、D8-8AB和D8-9AB);和集合4(包含D8-10AB、D8-11AB、D8-12AB和D8-13AB)。在20μl的体积中将每个退火的寡核苷酸的集合与10U的T4 DNA连接酶混合,16℃下温育连接反应。
然后将每个连接反应的产物用作模板,通过PCR扩增设计的DNA片段。具体地,采用连接的“集合1”混合物(即D8-1AB、D8-2AB和D8-3AB)作为模板,寡核苷酸D8-1F和D8-3R(SEQ ID NOs:236和237)作为引物,通过PCR扩增密码子优化的Δ8去饱和酶基因的第一部分。按照实施例6的描述,在50μl总体积中进行PCR扩增。将309bp PCR片段亚克隆到pGEM-T easy载体(Promega)中,得到pT8(1-3)。
采用连接的“集合2”混合物(即D8-4AB、D8-5AB和D8-6AB)作为模板,寡核苷酸D8-4F和D8-6R(SEQ ID NOs:238和239)作为引物,通过PCR类似地扩增密码子优化的Δ8去饱和酶基因的第二部分,克隆到pGEM-T-easy载体中,得到pT8(4-6)。采用连接的“集合3”混合物(即D8-7AB、D8-8AB和D8-9AB)作为模板,寡核苷酸D8-7F和D8-9R(SEQ ID NOs:240和241)作为引物,通过PCR类似地扩增密码子优化的Δ8去饱和酶基因的第三部分,克隆到pGEM-T-easy载体中,得到pT8(7-9)。最后,采用“集合4”连接混合物(即D8-10AB、D8-11AB、D8-12AB和D8-13AB作为模板,寡核苷酸D8-10F和D8-13R(SEQ ID NOs:242和243)作为引物,通过PCR类似地扩增密码子优化的Δ8去饱和酶基因的第四部分,克隆到pGEM-T-easy载体中,得到pT8(10-13)。
分别用pT8(1-3)、pT8(4-6)、pT8(7-9)和pT8(10-13)转化大肠杆菌,从氨苄青霉素抗体转化体中分离质粒DNA。纯化质粒DNA,用合适的限制内切酶消化,以释放pT8(1-3)的309bpNcoI/BglII片段(SEQ ID NO:244)、pT8(4-6)的321bp BglII/XhoI片段(SEQ ID NO:245)、pT8(7-9)的264bp XhoI/SacI片段(SEQID NO:246)和pT8(10-13)的369bp Sac1/Not1片段(SEQ ID NO:247)。然后组合这些片段,按照方向与Nco1/Not1消化的pY54PC(SEQID NO:120;WO2004/101757)连接在一起,得到pDMW240(图8D)。这在pDMW240中得到了合成的Δ8去饱和酶基因(“D8S-1”,SEQ IDNO:209)。
与公开的纤细眼虫Δ8去饱和酶氨基酸序列(SEQ ID NO:43)相比,D8S-1的第二个氨基酸从‘K’改变为‘E’,以便在翻译起始密码子周围添加NcoI位点。采用pDMW240作为模板以及寡核苷酸ODMW390和ODMW391(SEQ ID NOs:248和249)作为引物,通过体外诱变(Stratagene,San Diego,CA)构建了具有公开的纤细眼虫Δ8去饱和酶序列(SEQ ID NO:43)的准确氨基酸序列的另一种形式的合成基因。得到的质粒命名为pDMW255。pDMW255中的合成Δ8去饱和酶基因命名为“D8S-2”,其氨基酸序列与SEQ ID NO:43中描述的序列完全相同。
非功能性的密码子优化的Δ8去饱和酶基因
按照一般方法中的描述,分别用pDMW240(图8D)和pDMW255转化解脂耶氏酵母菌株ATCC #76982(Leu-)。含有重组构建体的酵母在补加了EDA[20:2(11,14)]的MM中生长。具体地,使含有pDMW240(含D8S-1)或pDMW255(含D8S-2)的转化解脂耶氏酵母的单菌落30℃下在3mL MM生长至OD600为约1.0。为了进行底物补料,随后将100μl细胞30℃下在含有10μg EDA底物的3mL MM中传代培养约24小时。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
两种转化体都没有从EDA生产DGLA,因此,D8S-1和D8S-2都不是功能性的,并且不能使EDA去饱和。嵌合D8S-1::XPR和D8S-2::XPR基因分别示于SEQ ID NOs:250和251。
发现GenBank中保藏的Δ8去饱和酶(获取号AAD45877[SEQ IDNO:43])与WO 00/34439或Wallis et al.(Archives of Biochem.Biophys,365:307-316(1999))(本文的SEQ ID NO:252)描述的Δ8去饱和酶的蛋白序列之间存在3个氨基酸的差异。具体地,发现GenBank获取号AAD45877缺少3个氨基酸。采用pDMW255作为模板,ODMW392和ODMW393(SEQ ID NOs:253和254)作为引物,通过体外诱变(Stratagene,San Diego,CA)在合成的D8S-2基因中加入了9bp,由此产生了与WO 00/34439和Wallis et al.(上文)(SEQ ID NO:252)中描述的序列相同的蛋白。得到的质粒命名为“D8S-3”(SEQ ID NO:255)。将pDMW261转化到耶氏酵母中之后,按照上文进行了采用EDA的补料实验。用D8S-3没有观察到EDA去饱和为DGLA。
分离纤细眼虫Δ8去饱和酶基因
纤细眼虫获自密西根州立大学(East Lansing,MI)的RichardTriemer博士的实验室。从10mL活跃生长的培养物中将1mL等分物转移到500mL玻璃瓶中的250mL纤细眼虫(Eg)培养基中。Eg培养基是通过在970mL水中混合以下成分而制备的:1g醋酸钠、1g牛肉提取物(目录号U126-01,Difco Laboratories,Detroit,MI)、2g 胰蛋白胨(目录号0123-17-3,Difco Laboratories)和2g酵母提取物(目录号0127-17-9,Difco Laboratories)。过滤灭菌后,无菌加入30mL土壤-水上清液(目录号15-3790,CarolinaBiological Supply Company,Burlington,NC),得到最终的Eg培养基。使纤细眼虫培养物在23℃,16小时光、8小时暗周期中无搅拌条件下生长2周。
2周后,取出10mL培养物进行脂质分析,并且在1,800xg离心5分钟。用水洗涤沉淀物一次,重新离心。真空中将得到的沉淀物干燥5分钟,重悬于100μL三甲基氢氧化锍(TMSH),振荡下在室温中温育15分钟。此后,加入0.5mL己烷,振荡下将管形瓶室温下温育15分钟。分离脂肪酸甲酯(从己烷层注射的5μL),用装有Omegawax 320熔融二氧化硅毛细管柱(目录号24152,Supelco Inc.)的Hewlett-Packard 6890气相色谱进行定量。对烤箱温度进行程序化,在220℃保持2.7分钟,以20℃/min增加到240℃,然后再保持2.3分钟。通过Whatman氢发生器提供载体气体。将保留时间与商购的标准物(目录号U-99-A,Nu-Chek Prep,Inc.)的甲酯进行比较,得到的色谱图示于图9。
通过1,800xg离心10分钟,沉淀剩余的2周培养物(240mL),用水洗涤1次,再次离心。采用RNA STAT-60TM试剂(TEL-TEST,Inc.,Friendswood,TX),根据提供的制造商的方案(采用5mL试剂,0.5mL水中的溶解的RNA)提取总RNA。以此方式,从沉淀物获得了1mg总RNA(2mg/mL)。用mRNA纯化试剂盒(AmershamBiosciences,Piscataway,NJ),按照提供的制造商的方案从1mg总RNA分离mRNA。以此方式,获得了85μg mRNA。
根据制造商的方案,采用用于cDNA合成的SuperScriptTM Choice系统(InvitrogenTM Life Technologies,Carlsbad,CA),用提供的寡聚(dT)引物,从765ng mRNA合成cDNA。将合成的cDNA溶解于20μL水中。
采用下文所述条件,用寡核苷酸引物Eg5-1和Eg3-3(SEQ IDNOs:256和257)从cDNA扩增纤细眼虫Δ8去饱和酶。具体地,将cDNA(1μL)与50pmol Eg5-1、50pmol Eg5-1、1μL PCR核苷酸混合物(10mM,Promega,Madison,WI)、5μL 10X PCR缓冲液(Invitrogen)、1.5μL MgCl2(50mM,Invitrogen)、0.5μ Taq聚合酶(Invitrogen)和水混合到50μL。反应条件是94℃ 3分钟,然后是35个循环的94℃ 45秒,55℃ 45秒和72℃ 1分钟。72℃下使PCR终止7分钟,然后保持在4℃。用5μL通过琼脂糖凝胶电泳分析PCR反应,观察到分子量为大约1.3kB的DNA条带。通过琼脂糖凝胶电泳分离剩余的45μL产物,按照制造商的方案,用ZymocleanTM Gel DNA Recovery试剂盒(Zymo Research,Orange,CA)纯化DNA条带。按照制造商的方案将得到的DNA克隆到 Easy载体(Promega)中。用T7、M13-28Rev、Eg3-2和Eg5-2(分别是SEQ ID NOS:258-261)对多个克隆进行测序。
由此获得了两类DNA序列,即Eg5(SEQ ID NO:44)和Eg12(SEQ ID NO:46),它们的差别仅仅是几个bp。Eg5和Eg12的翻译得到了差别仅仅是1个氨基酸的蛋白序列SEQ ID NO:45和47。因此,Eg5的DNA和蛋白序列分别示于SEQ ID NO:44和SEQ IDNO:45;Eg12的DNA和蛋白序列分别示于SEQ ID NO:46和SEQ IDNO:47。
分离的纤细眼虫Δ8去饱和酶序列与公开的纤细眼虫Δ8去饱和酶序列的比较
图10中示出了SEQ ID NO:45(Eg5)和SEQ ID NO:47(Eg12)中所示蛋白序列与GenBank获取号AAD45877的蛋白序列(gi:5639724;本文的SEQ ID NO:43)以及与Wallis et al.(Archives ofBiochem.Biophys.,365:307-316(1999);WO 00/34439)公开的蛋白序列[本文的SEQ ID NO:252]的比对。在所有4个序列中保守的氨基酸用星号(*)表示。程序中用虚线使序列的比对最大化。推定的细胞色素b5结构域加下划线。推定的His盒用粗体表示。同一性百分比计算发现Eg5 Δ8去饱和酶蛋白与SEQ ID NO:43具有95.5%同一性,与SEQ ID NO:252具有96.2%同一性,其中“同一性百分比”定义为两种蛋白之间相同的氨基酸的百分比。用LASERGENE生物信息学计算程序组(DNASTAR Inc.,Madison,WI)的Megalign程序进行序列比对和同一性百分比计算。用具有缺省参数(GAPPENALTY=10,GAP LENGTH PENALTY=10)的Clustal比对法(Higgins and Sharp,CABIOS.5:151-153(1989))进行序列的多重比对。采用Clustal方法的逐对比对的缺省参数是KTUPLE1,GAPPENALTY=3,WINDOW=5和DIAGONALS SAVED=5。对于各种纤细眼虫Δ8去饱和酶序列之间的差异的更复杂分析,参见共同未决的美国专利申请No.11/166993。
在啤酒糖酵母中进行的纤细眼虫Δ8去饱和酶序列的功能分析
酵母附加体质粒(YEp)型载体pRS425(Christianson et al.,Gene,110:119-22(1992))含有来自啤酒糖酵母2μ内源质粒的序列、LEU2选择标记和基于多功能噬菌粒pBluescript II SK+的主链的序列。按照与Jia et al.(Physiological Genomics,3:83-92(2000))中描述的相同方法在pRS425的SacII和SpeI位点之间克隆啤酒糖酵母的强组成型甘油醛-3-磷酸脱氢酶(GPD)启动子,以制备pGPD-425。将NotI位点导入pGPD-425的BamHI位点(由此产生侧翼于BamHI位点的NotI位点),从而得到质粒pY-75。按照上文的描述用NotI消化,从 Easy载体释放Eg5(SEQ ID NO:44)和Eg12(SEQ ID NO:46),并且克隆到pY-75的NotI位点,以分别制备pY89-5(保藏为ATCC #PTA-6048)和pY89-12。以此方式,将Δ8去饱和酶(即Eg5[SEQ ID NO:44]和Eg12[SEQ ID NO:46])克隆在强启动子后面,以便在啤酒糖酵母中表达。pY89-5的图谱示于图8E。
用标准醋酸锂转化程序将质粒pY89-5、pY89-12和pY-75转化到啤酒糖酵母BY4741(ATCC #201388)中。在补加了CSM-leu的DOBA培养基(Qbiogene,Carlsbad,CA)上选择转化体。将来自每个平板的转化体接种到2mL补加了CSM-leu的DOB培养基(Qbiogene)上,30℃下生长1天,此后将0.5mL转移到补加了EDA或EtrA到1mM的相同培养基中。将它们在30℃,250rpm温育过夜,通过离心获得沉淀物并且真空下干燥。用50μL TMSH对沉淀物进行酯交换,按照一般方法中的描述通过GC进行分析。分析了pY-75(即克隆75-1和75-2)和pY89-5(即克隆5-6-1和5-6-2)的两个克隆,同时分析了来自两个独立转化体的两组pY89-12的克隆(即克隆12-8-1、12-8-2、12-9-1和12-9-2)。
通过EDA补料的克隆的GC分析获得的脂质谱示于表19;通过EtrA补料的克隆的GC分析获得的脂质谱示于表20。脂肪酸鉴定为16:0(棕榈酸)、16:1(棕榈油酸)、18:0、18:1(油酸)、20:2[EDA]、20:3(8,11,14)[DGLA]、20:3(11,14,17)[ETrA]和20:4(8,11,14,17)[ETA];每一种的组成表示为总脂肪酸的百分比。
表19
超量表达纤细眼虫Δ8去饱和酶的转化啤酒糖酵母的脂质分析:EDA底物补料
| 克隆 | 16:0 | 16:1 | 18:0 | 18:1 | 20:2 | 20:3(8,11,14) | % 20:2转化 |
| 75-1(对照) | 14 | 32 | 5 | 38 | 10 | 0 | 0 |
| 75-2(对照) | 14 | 31 | 5 | 41 | 9 | 0 | 0 |
| 5-6-1(Eg5) | 14 | 32 | 6 | 40 | 6 | 2 | 24 |
| 5-6-2(Eg5) | 14 | 30 | 6 | 41 | 7 | 2 | 19 |
| 12-8-1(Eg12) | 14 | 30 | 6 | 41 | 9 | 1 | 7 |
| 12-8-2(Eg12) | 14 | 32 | 5 | 41 | 8 | 1 | 8 |
| 12-9-1(Eg12) | 14 | 31 | 5 | 40 | 9 | 1 | 8 |
| 12-9-2(Eg12) | 14 | 32 | 5 | 41 | 8 | 1 | 7 |
表20
超量表达纤细眼虫Δ8去饱和酶的转化啤酒糖酵母的脂质分析:ETrA底物补料
| 克隆 | 16:0 | 16:1 | 18:0 | 18:1 | 20:3(11,14,17) | 20:4(8,11,14,17) | %20:3转化 |
| 75-1(对照) | 12 | 25 | 5 | 33 | 24 | 0 | 0 |
| 75-2(对照) | 12 | 24 | 5 | 36 | 22 | 1 | 5 |
| 5-6-1(Eg5) | 13 | 25 | 6 | 34 | 15 | 7 | 32 |
| 5-6-2(Eg5) | 13 | 24 | 6 | 34 | 17 | 6 | 27 |
| 12-8-1(Eg12) | 12 | 24 | 5 | 34 | 22 | 2 | 8 |
| 12-8-2(Eg12) | 12 | 25 | 5 | 35 | 20 | 2 | 9 |
| 12-9-1(Eg12) | 12 | 24 | 5 | 34 | 22 | 2 | 9 |
| 12-9-2(Eg12) | 12 | 25 | 6 | 35 | 20 | 2 | 9 |
表19和20中的数据表明克隆的眼虫Δ8去饱和酶能够使EDA和EtrA去饱和。SEQ ID NO:47所示序列与SEQ ID NO:45所示序列相比具有一个氨基酸的改变,并且Δ8去饱和酶活性降低。
表30中的克隆75-2产生的小量20:4(8,11,14,17)与20:4(8,11,14,17)的标准相比具有略微不同的保留时间。这个峰更有可能是该实验中野生型酵母产生的小量的不同脂肪酸。
为解脂耶氏酵母进行密码子优化的Δ8去饱和酶基因的进一步修饰
根据有功能的眼虫Δ8去饱和酶(SEQ ID NOs:44和45)的氨基酸序列校正pDMW261中的合成D8S-3基因的氨基酸序列。采用作为模板的pDMW261并且采用核苷酸ODMW404(SEQ ID NO:262)和D8-13R(SEQ ID NO:243),扩增了编码合成的D8S-3去饱和酶基因的DNA片段。用Bio101的Geneclean试剂盒纯化得到的片段,随后用Kpn1和Not1消化(引物ODMW404导入KpnI位点,而引物D8-13R导入NotI位点)。将Kpn1/Not1片段(SEQ ID NO:263)克隆到Kpn1/Not1消化的pKUNFmKF2(图11A;SEQ ID NO:121)中,得到pDMW277(图11B)。
将设计用于扩增和校正D8S-3基因的5’末端的寡核苷酸YL521和YL522(SEQ ID NOs:264和265)用作另一个PCR中的引物,所述另一个PCR反应中将pDMW277用作模板。引物分别在PCR片段的5’和3’末端导入Nco1位点和BglII位点。用Bio101的GeneClean试剂盒纯化318bp PCR产物,随后用Nco1和BglII消化。将消化的片段和来自pDMW277的954bp BglII/NotI片段一起用于交换pZF5T-PPC的NcoI/NotI片段(图11C;SEQ ID NO:122),形成pDMW287。除了校正合成D8S-3基因的5’末端,该克隆反应也将合成的Δ8去饱和酶基因置于解脂耶氏酵母FBAIN启动子(SEQ ID NO:162)的控制下。
然后在pDMW287上进行最后一系列定点诱变反应中的第一个反应。设计第一组引物YL525和YL526(SEQ ID NOs:266和267),以便将pDMW287中的合成D8S-3基因的氨基酸从F校正到S(50号位置)。然后,该诱变反应得到的质粒成为下一次定点诱变反应的模板,引物是YL527和YL528(SEQ ID NOs:268和269)。这些引物设计用于将D8S-3基因的氨基酸从F校正到S(67号位置),导致产生质粒pDMW287/YL527。
为了完成基因的第二个四分之一中的序列校正,与基因的第一个四分之一上的突变同时进行以下反应。采用pDMW287作为模板,并且采用寡核苷酸YL529和YL530(SEQ ID NOs:270和271)作为引物,进行体外诱变反应,将合成D8S-3基因的氨基酸从C校正到W(177号位置)。将该诱变反应的产物(即pDMW287/Y529)用作随后反应的模板,该反应用引物YL531和YL532(SEQ ID NOs:272和273)将氨基酸从P校正为L(213号位置)。该反应的产物称作pDMW287/YL529-31。
与基因的第一个和第二个四分之一上的突变同时,类似地在基因的3’末端上进行反应。每个随后的诱变反应都采用来自前一个反应的质粒产物。在pDMW287上用引物YL533和YL534(SEQ ID NOs:274和275)将氨基酸从C校正为S(244号位置),得到pDMW287/YL533。用引物YL535和YL536(SEQ ID NOs:276和277)将pDMW287/YL533的合成D8S-3基因中的氨基酸从A校正为T(280号位置),形成pDMW287/YL533-5。最后,采用pDMW287/YL533-5作为模板,YL537和YL538(SEQ ID NOs:278和279)作为引物,将合成D8S-3基因中333号位置的氨基酸P校正为S。得到的质粒称作pDMW287/YL533-5-7。
用pDMW287/YL529-31的BglII/XhoI片段和pDMW287/YL533-5-7的XhoI/NotI片段改变pDMW287/YL257的BglII/NotI片段,得到pDMW287F(图11D)。SEQ ID NO:49示出了SEQ ID NO:48的核苷酸2-1270编码的氨基酸序列,其与SEQ ID NO:45所示序列基本相同,仅仅是在起始甲硫氨酸后有一个额外的缬氨酸。
实施例8
密码子优化的Δ9延伸酶基因和密码子优化的Δ8去饱和酶在解脂耶氏酵母中的功能性表达
本实施例描述了经过转化而共表达来自实施例6和7的密码子优化的Δ9延伸酶和密码子优化的Δ8去饱和酶的解脂耶氏酵母中的DGLA生物合成。本实施例从而证实了两种基因的活性和解脂耶氏酵母表达Δ9延伸酶/Δ8去饱和酶途径的能力。
具体地,将包含构建体pDMW287F(实施例7)的嵌合FBAIN::D8SF::Pex16基因的ClaI/PacI片段插入pDMW237(实施例6)的ClaI/PacI位点,得到构建体pDMW297(图11E;SED ID NO:123)。
质粒pDMW297含有以下成分:
表21
质粒pDMW297(SEQ ID NO:123)的说明
| SEQ ID NO:123内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| EcoRI/ClaI(9053-10448) | ARS18序列(GenBank获取号A17608) |
| ClaI/PacI(1-2590) | FBAIN::D8SF::Pex16,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●D8SF:密码子优化的Δ8去饱和酶基因(SEQ IDNO:48),来源于纤细眼虫(GenBank获取号AF139720)●Pex16:耶氏酵母Pex16基因(GenBank获取号U75433)的Pex16终止子序列 |
| PacI/SalI(2590-4082) | 耶氏酵母Ura3基因(GenBank获取号AJ306421) |
| SalI/BsiWI(4082-6257) | FBAIN::IgD9e::Pex20,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●IgD9e:密码子优化的Δ9延伸酶基因(SEQ IDNO:41),来源于球等鞭金藻(GenBank获取号390174)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
然后用构建体pDMW297根据一般方法转化菌株Y2031(实施例5)。将转化的细胞铺板到MM平板上,在30℃维持2-3天。挑选了总共8个在MM平板上生长的转化体,重新划线到新的MM平板上。一旦生长,30℃下将这些菌株单独接种到液体MM中,以250rpm/min振荡2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC进行分析显示在所有分析的转化体中都生产DGLA。1个菌株生产生产约3.2%,4个菌株生产4.3-4.5%,2个菌株生产5.5-5.8%,1个菌株生产6.4% DGLA(在此命名为菌株“Y0489”)。菌株Y0489中密码子优化的D8SF基因的“底物转化百分比”是75%。
实施例9
Δ9延伸酶/Δ8去饱和酶途径:制备生产占总脂质的约14%的ARA的Y2214菌株
本实施例描述了来源于解脂耶氏酵母ATCC #20362的菌株Y2214的构建,所述菌株能够生产占总脂质的14%的ARA(图4)。该菌株经过工程化,表达Δ9延伸酶/Δ8去饱和酶途径;因此,预期菌株Y2214的完整脂质谱分析表明最终的含ARA的油中没有共合成GLA。
此处菌株Y2214的开发需要构建菌株Y2152和Y2153(生产约3.5% DGLA)、菌株Y2173和Y2175(生产14-16%DGLA)和菌株Y2183和Y2184(生产5%ARA)。
制备生产占总脂质的约3.5%的DGLA的菌株Y2152和Y2153
用构建体pZP2C16M899(图12A,SEQ ID NO:124)整合含有四种嵌合基因(包括两种Δ9延伸酶、合成的C16/18脂肪酸延伸酶和Δ8去饱和酶)和含有单个氨基酸突变的耶氏酵母AHAS基因(乙酰羟酸合酶)的簇。耶氏酵母中的突变的AHAS酶赋予对磺酰脲的抗性,其用作阳性筛选标记。质粒pZP2C16M899设计用于整合到耶氏酵母菌株ATCC #20362的Pox2基因位点中,从而含有以下成分:
表22
质粒pZP2C16M899(SEQ ID NO:124)的说明
| SEQ ID NO:124内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| BsiWI/AscI(6152-6962) | 耶氏酵母Aco2基因(GenBank获取号AJ001300)的810bp 5’部分 |
| SphI/EcoRI(9670-10325) | 耶氏酵母Aco2基因(GenBank获取号AJ001300)的655bp 3’部分 |
| BsiWI/PmeI和EcoRV(929-3195) | GPM/FBA内含子::rELO2S::Oct,包含:●GPM/FBAIN:GPM::FBAIN嵌合启动子(SEQID NO:167)●rELO2S:密码子优化的rELO2延伸酶基因(SEQ ID NO:52),来源于大鼠(GenBank获取号AB071986)●OCT:耶氏酵母OCT基因(GenBank获取号X69988)的OCT终止子序列 |
| BsiWI/EcoRI(929-14447,反向) | GPAT::IgD9e::Pex20,包含:●GPAT:GPAT启动子(SEQ ID NO:164)●IgD9e:密码子优化的Δ9延伸酶基因(SEQ IDNO:41),来源于球等鞭金藻■Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| EcoRI/SwaI(14447-12912) | TEF::IgD9e::Lip1,包含:●TEF:TEF启动子(GenBank获取号AF054508)●IgD9e:SEQ ID NO:41,来源于球等鞭金藻●Lip1:耶氏酵母Lip1基因(GenBank获取号Z50020)的Lip1终止子序列 |
| SwaI/PacI(12912-10325) | FBAIN::D8SF::Pex16,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●D8SF:密码子优化的Δ8去饱和酶基因(SEQ IDNO:48),来源于纤细眼虫(GenBank获取号AF139720)●Pex16:耶氏酵母Pex16基因(GenBank获取号U75433)的Pex16终止子序列 |
| PmeI和EcoRV/BsiWI(3195-6152) | 包含W497L突变的解脂耶氏酵母AHAS基因(SEQ ID NO:280) |
用SphI/AscI消化质粒pZP2C16M899,然后用于根据一般方法转化ATCC #20362。转化后,将细胞铺板到含有150mg磺酰脲的MM平板上,在30℃维持2-3天。挑选磺酰脲抗性菌落,划线到磺酰脲选择平板的MM上。然后30℃下将总共96个转化体接种到具有磺酰脲的液体MM中,250rpm/min振荡2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示在含有pZP2C16M899的4种嵌合基因的转化体中存在DGLA,但在野生型耶氏酵母对照菌株中不存在。选择的96个菌株中的大多数生产占总脂质的2%以下的DGLA。28个菌株生产占总脂质的2-2.9%的DGLA。2个菌株生产占总脂质的3.5%的DGLA。65号和73号菌株在此分别称作菌株“Y2152”和“Y2153”。
制备生产占总脂质的约14-16%的DGLA的菌株Y2173和Y2175
用构建体pDMW314(图12B,SEQ ID NO:125)将含有四种嵌合基因(包括两种Δ9延伸酶、Δ8去饱和酶和Δ12去饱和酶)的簇整合到耶氏酵母菌株Y2152和Y2153的Ura3基因位点,从而增强DGLA的生产。质粒pDMW314含有以下成分:
表23
质粒pDMW314(SEQ ID NO:125)的说明
| SEQ ID NO:125内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| AscI/BsiWI(10066-9275) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的784bp 5’部分 |
| SphI/PacI(12774-1) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的516bp 3’部分 |
| SwaI/BsiWI(6582-9275) | FBAIN::F.D12S::Pex20,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●F.Δ12:串珠镰孢Δ12去饱和酶基因(SEQ IDNO:27)●Pex20:来自耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| ClaI/EcoRI(6199-4123) | GPAT::IgD9E::Pex20:如对pZP2C16M899的描述(上文) |
| EcoRI/SwaI(4123-2588) | TEF::IgD9E::Lip1:如对pZP2C16M899的描述(上文) |
| SwaI/PacI(2588-1) | FBAIN::D8SF::Pex16:如对pZP2C16M899的描述(上文) |
用AscI/SphI消化质粒pDMW314,然后用于根据一般方法转化解脂耶氏酵母菌株Y2152和Y2153。将转化的细胞铺板到FOA选择培养基平板上,在30℃维持2-3天。挑选FOA抗性菌落,划线到MM和MMU选择平板上。能够在MMU平板上生长但是不能在MM平板上生长的菌落选为Ura-菌株。然后30℃下将Ura-菌株的单菌落接种到液体MMU中,以250rpm/min振荡2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890GC进行分析。
GC分析显示几乎所有含有pDMW314的4种嵌合基因的转化体的DGLA生产都增加。选择的具有pDMW314的Y2152的48个Ura-菌株的大多数都生产占总脂质的约6-8%的DGLA。一个菌株(即47号,在此称作“Y2173”)生产占总脂质的约13.9%的DGLA。
类似地,选择的具有pDMW314的Y2153的24个Ura-菌株中的大多数生产占总脂质的约6-8%的DGLA。两个菌株(即6号和11号,在此命名为菌株“Y2175”和“Y2176”)分别生产占总脂质的约16.3%和17.2%的DGLA。
制备生产占总脂质的约5%的ARA的菌株Y2183和Y2184
用制备构建体pDMW322(图12C;SEQ ID NO:126)将两种Δ5嵌合基因整合到耶氏酵母Y2173和2175菌株的Leu2基因位点。质粒pDMW322含有以下成分:
表24
质粒pDMW232(SEQ ID NO:126)的说明
| SEQ ID NO:126内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| AscI/BsiWI(3437-2642) | 耶氏酵母Leu2基因(GenBank获取号AF260230)的788bp 5’部分 |
| SphI/PacI(6854-6145) | 耶氏酵母Leu2基因(GenBank获取号AF260230)的703bp 3’部分 |
| SwaI和PmeI/BsiWI(1-2642) | FBAIN::MAΔ5::Pex20,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●MAΔ5:高山被孢霉Δ5去饱和酶基因(SEQ IDNO:6)(GenBank获取号AF067654)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| EcoRI/SwaIwith PmeI(8833-1) | GPM/FBAIN::I.Δ5S::Oct,包含:●GPM/FBAIN:GPM::FBAIN嵌合启动子(SEQ IDNO:167)●I.Δ5S:密码子优化的Δ5去饱和酶基因(SEQ IDNO:10),来源于球等鞭金藻(WO 2002/081668)●耶氏酵母OCT基因(GenBank获取号X69988)的OCT:OCT终止子序列 |
| EcoRI/PmeI(8833-7216) | 耶氏酵母Ura3基因(GenBank获取号AJ306421) |
用AscI/SphI消化质粒pDMW322,然后用于根据一般方法转化菌株Y2173和Y2175。转化后,将细胞铺板到MMLe平板上,在30℃维持2-3天。挑选在MMLe平板上生长的各个菌落,划线到MM和MMLe平板上。能够在MMLe平板上生长但是不能在MM平板上生长的菌落选为Leu2-菌株。然后30℃下将Leu2-菌株的单菌落接种到液体MMLe中,以250rpm/min振荡的条件下培养2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示pDMW322转化体中存在ARA,但亲本Y2173和Y2175菌株中不存在。具体地,在48个选择的具有pDMW322的Y2173的Leu2-转化体中,大多数菌株生产占总脂质的4.4%以下的ARA;但是,有两个菌株(即1号和42号,在此称作菌株“Y2181”和“Y2182”)分别生产占总脂质的约4.5%和5.8%的ARA。
平行地,在48个选择的具有pDMW322的Y2175的Leu2-转化体中,大多数菌株生产占总脂质的4.5%以下的ARA。有3个菌株(即22号、42号和47号,在此称作菌株“Y2183”、“Y2184”和“Y2185”)分别生产占工程化的耶氏酵母中总脂质的约4.9%、4.6%和4.7%的ARA。
制备生产占总脂质的约14%的ARA的菌株Y2214
用构建体pZKSL5598(图12D,SEQ ID NO:127)将含有四种嵌合基因(包括Δ9延伸酶、Δ8去饱和酶和两种Δ5去饱和酶)的簇整合到耶氏酵母Y2183和Y2185菌株的Lys5基因(GenBank获取号M34929)位点,从而增强ARA的生产。质粒pZKSL5598含有以下成分:
表25
质粒pZKSL5598(SEQ ID NO:127)的说明
| SEQ ID NO:127内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| AscI/BsiWI(10409-9573) | 耶氏酵母Lys5基因(GenBank获取号M34929)的794bp 5’部分 |
| SphI/PacI(13804-13117) | 耶氏酵母Lys5基因(GenBank获取号M34929)的687bp 3’部分 |
| BsiWI/SwaI(7150-9573) | NT::I.D5S::Lip1,包含:●NT:YAT1启动子(SEQ ID NO:165)●I.Δ5S:密码子优化的Δ5去饱和酶基因(SEQ IDNO:10),来源于球等鞭金藻(WO 2002/081668)●Lip1:耶氏酵母Lip1基因(GenBank获取号Z50020)的Lip1终止子序列 |
| SalI/BsiWI(4537-7150) | GPAT::MAΔ5::Pex20,包含:●GPAT:GPAT启动子(SEQ ID NO:164)●MAΔ5:高山被孢霉Δ5去饱和酶基因(SEQ IDNO:6)(GenBank获取号AF067654)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| SwaI/PmeI(2381-348) | FBAINm::IgD9e::OCT,包含:●FBAINm:FBAINm启动子(SEQ ID NO:163)●IgD9e:密码子优化的Δ9延伸酶基因(SEQ IDNO:41),来源于球等鞭金藻●OCT:耶氏酵母OCT基因(GenBank获取号X69988)的OCT终止子序列 |
| ClaI/PacI(1-13804) | GPD::D8SF::Pex16,包含:●GPD:GPD启动子(SEQ ID NO:158)●D8SF:密码子优化的Δ8去饱和酶基因(SEQ IDNO:48),来源于纤细眼虫(GenBank获取号AF139720)●Pex16:耶氏酵母Pex16基因(GenBank获取号U75433)的Pex16终止子序列 |
| SalII/PmeI(4537-2417) | 耶氏酵母Leu2基因(GenBank获取号AF260230) |
用AscI/SphI消化质粒pZKSL5598,然后用于根据一般方法分别转化菌株Y2183和Y2184。转化后,将细胞铺板到MMLys平板上,在30℃维持2-3天。挑选在MMLys平板上生长的来自每次转化的各个菌落,划线到MM和MMLys平板上。能够在MMLys平板上生长但是不能在MM平板上生长的菌落选为Lys-菌株。然后30℃下将Lys-菌株的单菌落接种到液体MMLy中,以250rpm/min振荡2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示pZKSL5598转化体中的ARA生产增加。在48个选择的具有pZKSL5598的Y2183的Lys-转化体中,大多数菌株生产占总脂质的4-9.5%的ARA。3个菌株(即7号、12号和37号,在此命名为“Y2209”、“Y2210”和“Y2211”)分别生产占总脂质的约9.9%、10.3%和9.6%的ARA。
在48个选择的具有pZKSL5598的Y2184的Lys-转化体中,大多数菌株生产占总脂质的4-11%的ARA。2个菌株(即3号和22号,在此命名为“Y2213”和“Y2214”)分别生产占总脂质的约11.9%和14%的ARA。
实施例10
制备生产占总脂质的14%的EPA的中间菌株2067U
本实施例描述了来源于解脂耶氏酵母ATCC #20362的菌株Y2067U的构建,该菌株能够生产占总脂质的显著浓度的EPA(图4)。在这种生产EPA的菌株中,基于TAG含量和/或组成的分析,检验了高山被孢霉LPAAT2、DGAT1和DGAT2以及解脂耶氏酵母CPT1基因超量表达的作用,分别如实施例14、15、16和21的描述(参见下文)。
菌株Y2067U(生产14% EPA)的开发需要构建菌株M4(生产8%DGLA,描述于实施例4)、菌株Y2034(生产10% ARA,描述于实施例4)、菌株E(生产10% EPA)、菌株EU(生产10% EPA)和菌株Y2067(生产15% EPA)。
制备生产占总脂质的约10%的EPA的E菌株
制备构建体pZP3L37(图13A;SEQ ID NO:128),将三种合成的Δ17去饱和酶嵌合基因整合到Y2034菌株的酰基-CoA氧化酶3基因,如实施例4的描述。质粒pZP3L37含有以下成分:
表26
质粒pZP3L37(SEQ ID NO:128)的说明
| SEQ ID NO:128内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| AscI/BsiWI(6813-6043) | 耶氏酵母Pox3基因(GenBank获取号AJ001301)的763bp 5’部分 |
| SphI/PacI(9521-10345) | 耶氏酵母Pox3基因(GenBank获取号AJ001301)的818bp 3’部分 |
| ClaI/BsiWI(4233-6043) | TEF::Δ17S::Pex20,包含:●TEF:TEF启动子(GenBank获取号AF054508)●Δ17S:密码子优化的Δ17去饱和酶基因(SEQ IDNO:16),来源于异丝水酶(US 2003/0196217 A1)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| ClaI/PmeI(4233-1811) | FBAIN::Δ17S::Lip2,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●Δ17S:SEQ ID NO:16(参见上文)●Lip2:耶氏酵母Lip2基因(GenBank获取号AJ012632)的Lip2终止子序列 |
| PmeI/SwaI(1811-1) | 耶氏酵母Leu2基因(GenBank获取号AF260230) |
| PacI/SwaI(10345-1) | FBAINm::Δ17S::Pex16,包含:●FBAINm:FBAINm启动子(SEQ ID NO:163)●Δ17S:SEQ ID NO:16(参见上文)●Pex16:耶氏酵母Pex16基因(GenBank获取号U75433)的Pex16终止子序列 |
用AscI/SphI消化质粒pZP3L37,然后用于根据一般方法转化菌株Y2034。转化后,将细胞铺板到MM平板上,在30℃维持2-3天。挑选在MM平板上生长的总共48个转化体,重新划线到新的MM平板上。一旦生长,30℃下将这些菌株单独接种到液体MM中,以250rpm/min振荡的条件下培养2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示大多数具有pZP3L37的转化体中存在EPA,但亲本菌株(即Y2034)中不存在。在48个选择的具有pZP3L37的转化体中,18个菌株生产占工程化的耶氏酵母中总脂质的2%以下的EPA,14个菌株生产2-3%的EPA,1个菌株生产约7%的EPA。
在按照一般方法中的描述用“两阶段生长条件(即48hrs MM,72 hrs HGM)”培养生产7% EPA的菌株后,进一步分析该菌株。GC分析显示工程化的菌株在两阶段生长后生产占总脂质的约10%的EPA。该菌株命名为“E”菌株。
制备具有Ura-表型的生产占总脂质的约10%的EPA的EU菌株
通过鉴定具有5-FOA抗性的菌株E的突变细胞,制备了菌株EU(Ura-)。具体地,将1环的耶氏酵母E菌株细胞接种到3mL YPD培养基中,30℃下以250rpm振荡的条件下生长24小时。用YPD将培养物稀释到OD600为0.4,然后再温育4小时。将培养物铺板(100μl/平板)到MM+FOA平板上,30℃下维持2-3天。总共挑选了16个FOA抗性菌落,划线到MM和MM+FOA选择平板上。从中挑选出10个在FOA选择平板上生长,但是不在MM平板上生长的菌落,选作可能的Ura-菌株。
将这些菌株之一用作pY37/F15转化的宿主,pY37/F15包含嵌合GPD::串珠镰孢Δ15::XPR2基因和作为选择标记的Ura3基因(图13B;SEQ ID NO:129)。在MM平板上选择3天后,数百个菌落在平板上生长,不携带质粒的转化对照没有菌落生长。这种5-FOA抗性菌株命名为“EU”。
然后将EU菌株的单菌落接种到额外含有0.1g/L尿苷的液体MMU中,30℃下以250rpm振荡的条件下生长2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard6890GC进行分析。GC分析显示EU菌株生产占总脂质的约10%的EPA。
制备生产占总脂质的约15%的EPA的Y2067菌株
制备质粒pKO2UF2PE(图13C;SEQ ID NO:130),将含有两种嵌合基因(包括异源Δ12去饱和酶和C18/20延伸酶)和Ura3基因的簇整合到菌株EU的天然耶氏酵母Δ12去饱和酶基因中。质粒pKO2UF2PE含有以下成分:
表27
质粒pKO2UF2PE(SEQ ID NO:130)的说明
| SEQ ID NO:130内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| AscI/BsiWI(3382-2645) | 耶氏酵母Δ12去饱和酶基因(SEQ ID NO:23)的730bp 5’部分 |
| SphI/EcoRI(6090-6646) | 耶氏酵母Δ12去饱和酶基因(SEQ ID NO:23)的556bp 3’部分 |
| SwaI/BsiWI/(1-2645) | FBAINm::F.Δ12DS::Pex20,包含:●FBAINm:FBAINm启动子(SEQ ID NO:163)●F.Δ12:串珠镰孢Δ12去饱和酶基因(SEQ IDNO:27)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| SwaI/PmeI(1-8525) | GPAT::EL1S::OCT,包含:●GPAT:GPAT启动子(SEQ ID NO:164)●EL1S:密码子优化的延伸酶1基因(SEQ IDNO:19),来源于高山被孢霉(GenBank获取号AX464731)●OCT:耶氏酵母OCT基因(GenBank获取号X69988)的OCT终止子序列 |
| EcoRI/PacI(6646-8163) | 耶氏酵母Ura3基因(GenBank获取号AJ306421) |
用AscI/SphI消化质粒pKO2UF2PE,然后用于根据一般方法转化菌株EU(但将菌株EU划线到YPD平板上并且生长大约36小时[而不是18小时],然后悬浮在转化缓冲液中)。转化后,将细胞铺板到MM平板上,在30℃维持2-3天。挑选在MM平板上生长的总共72个转化体,独立地重新划线到新的MM平板上。一旦生长,30℃下将这些菌株单独接种到液体MM中,以250rpm/min振荡的条件下培养2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示几乎所有的具有pKO2UF2PE的转化体中都存在EPA。更具体地,在72个选择的转化体中,17个菌株生产占工程化的耶氏酵母中总脂质的8-9.9%的EPA,27个菌株生产10-10.9%EPA,16个菌株生产11-11.9% EPA,7个菌株生产12-12.7% EPA。按照一般方法中的描述,用“两阶段生长条件(即48hrs MM,72hrsHGM)”进一步分析生产12.7% EPA的菌株。GC分析显示工程化的菌株在两阶段生长后生产占总脂质的约15%的EPA。该菌株命名为“Y2067”菌株。
制备具有Ura-表型的生产占总脂质的约14%的EPA的Y2067U菌株
为了破坏Y2067菌株中的Ura3基因,制备了构建体pZKUT16(图13D;SEQ ID NO:131),将TEF::rELO2S::Pex20嵌合基因整合到菌株Y2067的Ura3基因中。rELO2S是密码子优化的rELO基因,其编码将16:0延伸为18:0的大鼠肝酶(即C16/18 延伸酶)。质粒pZKUT16含有以下成分:
表28
质粒pZKUT16(SEQ ID NO:131)的说明
| SEQ ID NO:131内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| BsiWI/PacI(1-721) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的721bp 5’部分 |
| SalI/ClaI(3565-4289) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的724bp 3’部分 |
| ClaI/BsiWI(4289-1) | TEF::rELO2S::Pex20,包含:●TEF:TEF启动子(GenBank获取号AF054508)●rELO2S:密码子优化的rELO2延伸酶基因(SEQID NO:52),来源于大鼠(GenBank获取号AB071986)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
用SalI/PacI消化质粒pZKUT16,然后用于根据一般方法转化Y2067菌株。转化后,将细胞铺板到MM+5-FOA选择平板上,在30℃维持2-3天。
挑选了总共24个在MM+5-FOA平板上生长的转化体,分别重新划线到MM平板和MM+5-FOA平板上。能够在MM+5-FOA平板上生长,但是不能在MM平板上生长的菌株选作Ura-菌株。30℃下将总共10个Ura-菌株单独接种到液体MMU培养基中,在250rpm/min振荡的条件下生长1天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示在MMU培养基中生长1天后,所有具有pZKUT16的转化体中都存在5-7% EPA。用两阶段生长条件(即48hrs MM,96hrs HGM)进一步分析生产6.2% EPA的菌株。GC分析显示工程化的菌株生产占总脂质的约14%的EPA。该菌株命名为菌株“Y2067U”。
该菌株相对于野生型解脂耶氏酵母ATCC #20362的最终基因型是Ura3-,Pox3-,Y.Δ12-,FBA::F.Δ12::Lip2,FBAINm::F.Δ12::Pex20,TEF::Δ6S::Lip1,FBAIN::E1S::Pex20,GPAT::E1S::Oct,TEF::E2S::Xpr,FBAIN::Δ5::Pex20,TEF::Δ5::Lip1,FBAIN::Δ17S::Lip2,FBAINm::Δ17S::Pex16,TEF::Δ17S和TEF::rELO2S::Pex20。
实施例11
本实施例描述了来源于解脂耶氏酵母ATCC #20362的菌株Y2107U1的构建,该菌株能够生产占总脂质的显著浓度的EPA(图4)。基于TAG含量和/或组成的分析,在这种生产EPA的菌株中检验高山被孢霉GPAT基因超量表达的影响,如实施例17的描述(下文)。
菌株Y2107U1(生产16% EPA,并且具有Ura-表型)的开发需要构建菌株M4(生产8% DGLA,并且描述于实施例4)、菌株2047(生产11% ARA,并且描述于实施例4)、菌株Y2048(生产11%EPA)、菌株Y2060(生产13% EPA)、菌株Y2072(生产15% EPA)、菌株Y2072U1(生产14% EPA)和菌株Y2089(生产18% EP)。
制备生产占总脂质的约11%的EPA的Y2048菌株
制备构建体pZP3L37(图13A;SEQ ID NO:128;实施例10),将三种合成的Δ17去饱和酶嵌合基因整合到Y2047菌株(实施例4)的酰基-CoA氧化酶3基因。具体地,用SphI/AscI消化质粒pZP3L37,然后用于根据一般方法转化菌株Y2047。转化后,将细胞铺板到MM平板上,在30℃维持2-3天。挑选了总共96个在MM平板上生长的转化体,重新划线到新的MM平板上。一旦生长,30℃下将这些菌株单独接种到液体MM中,以250rpm/min振荡的条件下培养2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示大多数具有pZP3L37的转化体中存在EPA,但亲本Y2047菌株中不存在。在96个选择的具有pZP3L37的转化体中,20个菌株生产占工程化的耶氏酵母中总脂质的2%以下的EPA,23个菌株生产2-3%的EPA,5个菌株生产3-4%的EPA,2个菌株(即71号菌株和94号菌株)生产约6%的EPA。
选择71号菌株(生产6% EPA),通过以两阶段生长条件(即48hrs MM,72hrs HGM)培养而进一步分析。GC分析显示71号菌株生产占总脂质的约11%的EPA。该菌株命名为菌株“Y2048”。
制备具有Ura-表型的生产占总脂质的约13%的EPA的Y2060菌株
为了破坏菌株Y2048中的Ura3基因,将构建体pZKUT16(图13D;SEQ ID NO:131,参见实施例10)用于将TEF::rELO2S::Pex20嵌合基因整合到Y2048菌株的Ura3基因中。具体地,用SalI/PacI消化质粒pZKUT16,然后用于根据一般方法转化菌株Y2048。转化后,将细胞铺板到MM+5-FOA选择平板上,在30℃维持2-3天。
挑选了总共40个在MM+5-FOA平板上生长的转化体,分别重新划线到MM平板和MM+5-FOA平板上。能够在MM+5-FOA平板上生长,但是不能在MM平板上生长的菌株选作Ura-菌株。30℃下将这40个Ura-菌株的每一个单独接种到液体MMU培养基中,在250rpm/min振荡的条件下生长2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示在MMU培养基中生长2天后,14个菌株生产占总脂质的5%以下的EPA,9个菌株生产5-5.9%的EPA,15个菌株生产6-6.9%的EPA,7个菌株生产7-8%的EPA。用两阶段生长条件(即48hrs MM,96hrs HGM)进一步分析生产7-8% EPA的菌株。GC分析显示所有这些菌株都生产占总脂质的10%以上的EPA;其中之一生产约13%的EPA。该菌株命名为菌株“Y2060”。
制备生产占总脂质的约15%的EPA的Y2072菌株
用构建体pKO2UM25E(图14A;SEQ ID NO:132)将含有三种嵌合基因(包括C18/20延伸酶、Δ12去饱和酶和Δ5去饱和酶)和Ura3基因的簇整合到菌株Y2060的天然耶氏酵母Δ12去饱和酶基因中。质粒pKO2UM25E含有以下成分:
表29
质粒pKO2UM25E(SEQ ID NO:132)的说明
| SEQ ID NO:132内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| HindIII/AscI(1-728) | 耶氏酵母Δ12去饱和酶基因(SEQ ID NO:23)的728bp 5’部分 |
| SphI/EcoRI(3436-3992) | 耶氏酵母Δ12去饱和酶基因(SEQ ID NO:23)的556bp 3’部分 |
| BsiWI/HindIII(10437-1) | GPAT::EL1S::XPR,包含:●GPAT:GPAT启动子(SEQ ID NO:164)●EL1S:密码子优化的延伸酶1基因(SEQ IDNO:19),来源于高山被孢霉(GenBank获取号AX464731)●XPR:耶氏酵母Xpr基因(GenBank获取号M17741)的3’区的约100bp |
| BglII/BsiWI(7920-10437) | FBAIN::M.Δ12::Pex20,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●M.Δ12:深黄被孢霉Δ12去饱和酶基因(GenBank获取号AF417245;SEQ ID NO:25)■Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| SalI/PacI(6046-7544) | 耶氏酵母Ura3基因(GeneBank获取号AJ306421) |
| EcoRI/SalI(3992-6046) | TEF::I.Δ5S::Pex20,包含:●TEF:TEF启动子(GenBank获取号AF054508)●I.Δ5S:密码子优化的Δ5去饱和酶基因(SEQ IDNO:10),来源于球等鞭金藻(WO 2002/081668)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
用SphI/AscI消化质粒pKO2UM25E,然后用于根据一般方法转化菌株Y2060。转化后,将细胞铺板到MM平板上,在30℃维持2-3天。
挑选了总共63个在MM平板上生长的转化体,重新划线到新的MM平板上。一旦生长,30℃下将这些菌株单独接种到液体MM中,以250rpm/min振荡的条件下培养2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示在MM培养基中生长2天后,几乎所有的具有pKO2UM25E的转化体中都存在EPA。在63个选择的转化体中,26个菌株生产6-8.9%的EPA,46个菌株生产9%以上的EPA。选择生产9%以上的EPA的菌株,用于用两阶段生长程序(即48hrs MM,96hrs HGM)进一步分析。GC分析显示在两阶段生长后,46个选择的菌株中的45个菌株生产占总脂质的11-14.5%的EPA,而2号培养物生产15.1%的EPA。该菌株(即2号)命名为菌株“Y2072”。
制备具有Ura-表型的生产占总脂质的约14%的EPA的Y2072U1菌株
建立构建体pZKUGPI5S(图14B;SEQ ID NO:133),用于将GPAT::I.Δ5S::Pex20嵌合基因整合到Y2072菌株的Ura3基因中。更具体地,质粒pZKUGPI5S包含以下成分:
表30
质粒pZKUGPI5S(SEQ ID NO:133)的说明
| SEQ ID NO:133内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| BsiWI/PacI(318-1038) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的721bp 5’部分 |
| SalI/ClaI(3882-4606) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的724bp 3’部分 |
| ClaI/BsiWI(4606-318) | GPAT::I.Δ5S::Pex20,包含:●GPAT:GPAT启动子(SEQ ID NO:164)●I.Δ5S:密码子优化的Δ5去饱和酶基因(SEQ IDNO:10),来源于球等鞭金藻(WO 2002/081668)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
用SalI/PacI消化质粒pZKUGPI5S,然后用于根据一般方法转化菌株Y2072。转化后,将细胞铺板到MM+5-FOA选择平板上,在30℃维持3-4天。
挑选了总共24个在MM+5-FOA平板上生长的转化体,分别重新划线到MM平板和MM+5-FOA平板上。能够在MM+5-FOA平板上生长,但是不能在MM平板上生长的菌株选作Ura-菌株。将这24个Ura-菌株的每一个单独接种到液体MMU培养基中,30℃下在250rpm/min振荡的条件下生长2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示在MMU中生长2天后,8个菌株生产占总脂质的7.3-8.9%的EPA,14个菌株生产9-9.9%的EPA,1个菌株生产10.5%的EPA(即1号),1个菌株生产10.7%的EPA(即23号)。用两阶段生长条件(即48hrs MM,96hrs HGM)进一步分析1号和23号菌株。GC分析显示在两阶段生长后这两个菌株都生产占总脂质的约14%的EPA。1号菌株命名为菌株“Y2072U1”。
制备生产占总脂质的约18%的EPA的Y2089菌株
用构建体pDMW302T16(图14C;SEQ ID NO:134)将含有四种嵌合基因(包括C16/18 延伸酶、C18/20延伸酶、Δ6去饱和酶和Δ12去饱和酶)和Ura3基因的簇整合到Y2072U1菌株的耶氏酵母脂肪酶1基因位点中。质粒pDMW302T16含有以下成分:
表31
质粒pDMW302T16(SEQ ID NO:134)的说明
| SEQ ID NO:134内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| BsiWI/AscI(1-817) | 耶氏酵母脂肪酶1基因(GenBank获取号Z50020)的817bp 5’部分 |
| SphI/PacI3525-4294 | 耶氏酵母脂肪酶1基因(GenBank获取号Z50020)的769bp 3’部分 |
| EcoRI/BsiWI(13328-1) | TEF::rELO2S::Pex20,包含:●TEF:TEF启动子(GenBank获取号AF054508)●rELO2S:密码子优化的rELO2延伸酶基因(SEQ ID NO:52),来源于大鼠(GenBank获取号AB071986)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| BglII/EcoRI(10599-13306) | FBAIN::D6S::Lip1,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●Δ6S:密码子优化的Δ6去饱和酶基因(SEQ IDNO:3),来源于高山被孢霉(GenBank获取号AF465281)■Lip1:来自耶氏酵母Lip1基因(GenBank获取号Z50020)的Lip1终止子序列 |
| ClaI/PmeI(8078-10555) | GPDIN::EL1S::Lip2,包含:■GPDIN:GPDIN启动子(SEQ ID NO:159)●EL1S:密码子优化的延伸酶1基因(SEQ IDNO:19),来源于高山被孢霉(GenBank获取号AX464731)●Lip2:耶氏酵母脂肪酶2基因(GenBank获取号AJ012632)的Lip2终止子 |
| EcoRI/ClaI(6450-8078) | 耶氏酵母Ura 3基因(GeneBank获取号AJ306421) |
| PacI/EcoRI(4294-6450) | TEF::F.Δ12::Pex16,包含:●TEF:TEF启动子(GenBank获取号AF054508)●F.Δ12:串珠镰孢Δ12去饱和酶基因(SEQ IDNO:27)■Pex16:耶氏酵母Pex16基因(GenBank获取号U75433)的Pex16终止子 |
用SphI/AscI消化质粒pDMW302T16,然后用于根据一般方法转化菌株Y2072U1。转化后,将细胞铺板到MM平板上,在30℃维持3-4天。挑选了总共48个在MM平板上生长的转化体,重新划线到新的MM平板上。一旦生长,将这些菌株单独接种到液体MM中,30℃下在250rpm/min振荡的条件下生长2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890GC进行分析。
GC分析显示在MM培养基中生长2天后,几乎所有的具有pDMW302T16的Y2072U1的转化体都生产EPA。在48个选择的转化体中,27个菌株生产10%以下的EPA,14个菌株生产10-12.9%的EPA,5个菌株生产13-13.9%的EPA。选择34号菌株(生产13.9%EPA)用于用两阶段生长程序(即48hrs MM,96hrs HGM)进一步分析。GC分析显示34号菌株生产占总脂质的大约18%的EPA。34号菌株命名为菌株“Y2089”。
该菌株相对于野生型解脂耶氏酵母ATCC #20362的最终基因型是:Pox3-,LIP1-,Y.Δ12-,FBA::F.Δ12::Lip2,TEF::F.Δ12::Pex16,FBAIN::MΔ12::Pex20,TEF::Δ6S::Lip1,FBAIN::Δ6S::Lip1,FBAIN::E1S::Pex20,GPAT::E1S::Oct,GPDIN::E1S::Lip2,TEF::E2S::Xpr,FBAIN::MAΔ5::Pex20,TEF::MAΔ5::Lip1,TEF::HΔ5S::Pex16,TEF::IΔ5S::Pex20,GPAT::IΔ5S::Pex20,FBAIN::Δ17S::Lip2,FBAINm::Δ17S::Pex16,TEF::Δ17S::Pex16和2X TEF::rELO2S::Pex20。
制备具有Ura-表型的生产占总脂质的约16%的EPA的Y2107U1菌株
建立构建体pZKUGPE1S(图14D;SEQ ID NO:135),用于将GPAT::EL1S::Pex20嵌合基因整合到菌株Y2089的Ura3基因中。更具体地,质粒pZKUGPE1S包含以下成分:
表32
质粒pZKUGPE1S(SEQ ID NO:135)的说明
| SEQ ID NO:135内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| BsiWI/PacI(318-1038) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的721bp 5’部分 |
| SalI/ClaI(3882-4606) | 耶氏酵母Ura3基因(GenBank获取号AJ306421)的724bp 3’部分 |
| ClaI/BsiWI(4606-318) | GPAT::E1S::Pex20,包含:●GPAT:GPAT启动子(SEQ ID NO:164)●EL1S:密码子优化的延伸酶1基因(SEQ IDNO:19),来源于高山被孢霉(GenBank获取号AX464731)●Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
用SalI/PacI消化质粒pZKUGPE1S,然后用于根据一般方法转化菌株Y2089。转化后,将细胞铺板到MM+5-FOA选择平板上,在30℃维持3-4天。
挑选了总共8个在MM+5-FOA平板上生长的转化体,分别重新划线到MM平板和MM+5-FOA平板上。能够在MM+5-FOA平板上生长,但是不能在MM平板上生长的菌株选作Ura-菌株。将这8个Ura-菌株的每一个单独接种到液体MMU中,30℃下在250rpm/min振荡的条件下生长2天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示在MMU中生长2天后,6个菌株生产占总脂质的6.6-8.7%的EPA,2个菌株生产9.4-10%的EPA(即4号和5号)。用两阶段生长条件(即48hrs MM,96hrs HGM)进一步分析4号和5号菌株。GC分析显示这两个菌株在两阶段生长后生产占总脂质的约16%的EPA。4号菌株命名为菌株“Y2107U1”。
实施例12
制备生产占总脂质的9-12%的EPA的中间菌株MU
本实施例描述了来源于解脂耶氏酵母ATCC #20362的菌株MU的构建,该菌株能够生产占总脂质的显著浓度的EPA(图4)。基于TAG含量和/或组成的分析,在这种生产EPA的菌株中检验多个天然解脂耶氏酵母酰基转移酶敲除的影响,如实施例24的描述(下文)。
菌株MU(生产9-12% EPA)的开发需要构建菌株M4(生产8%DGLA,并且描述于实施例4)、菌株Y2034(生产10% ARA,并且描述于实施例4)、菌株E(生产10% EPA,并且描述于实施例10)、菌株EU(生产10% EPA,并且描述于实施例10)和菌株M26(生产14% EPA)。
制备生产占总脂质的14%的EPA的M26菌株
用构建体pZKO2UM26E(SEQ ID NO:136;图15A)将含有三种嵌合基因(包括C18/20延伸酶、Δ6去饱和酶和Δ12去饱和酶)和Ura3基因的簇整合到EU菌株(实施例10)的耶氏酵母Δ12去饱和酶基因位点中。质粒pKO2UM26E含有以下成分:
表33
质粒pKO2UM26E(SEQ ID NO:136)的说明
| SEQ ID NO:136内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| HindIII/AscI(1-728) | 耶氏酵母Δ12去饱和酶基因(SEQ ID NO:23)的728bp 5’部分 |
| SphI/EcoRI(3436-3992) | 耶氏酵母Δ12去饱和酶基因(SEQ ID NO:23)的556bp 3’部分 |
| BsiWI/HindIII(11095-1) | GPAT::EL1S::XPR,包含:GPAT:GPAT启动子(SEQ ID NO:164)EL1S:密码子优化的延伸酶1基因(SEQ ID NO:19),来源于高山被孢霉(GenBank获取号AX464731)XPR:耶氏酵母Xpr基因(GenBank获取号M17741)的3’区的约100bp |
| BglII/BsiWI(8578-11095) | FBAIN::M.Δ12::Pex20,包含:FBAIN:FBAIN启动子(SEQ ID NO:162)M.Δ12:深黄被孢霉Δ12去饱和酶基因(GenBank获取号AF417245;SEQ ID NO:25)Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
| SalI/PacI(6704-8202) | 耶氏酵母Ura3基因(GenBank获取号AJ306421) |
| EcoRI/SalI(3992-6704) | FBAIN::M.Δ6B::Pex20,包含:TEF:TEF启动子(GenBank获取号AF054508)M.Δ6B:高山被孢霉Δ6去饱和酶基因“B”(GenBank获取号AB070555;SEQ ID NO:4)Pex20:耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
用SphI/AscI消化质粒pKO2UM26E,然后用于根据一般方法转化EU菌株(实施例10)。转化后,将细胞铺板到MM平板上,在30℃维持2-3天。
挑选了总共48个在MM平板上生长的转化体,重新划线到新的MM平板上。一旦生长,30℃下将这些菌株单独接种到液体MM中,以250rpm/min振荡的条件下生长1天。通过离心收集细胞,提取脂质,通过酯交换制备脂肪酸甲酯,随后用Hewlett-Packard 6890 GC进行分析。
GC分析显示在MM培养基中生长1天后,几乎所有的具有pKO2UM26E的转化体中都存在EPA。在48个选择的转化体中,5个菌株生产占工程化的耶氏酵母中的总脂质的4%以下的EPA,23个菌株生产4-5.9% EPA,9个菌株生产6-6.9% EPA,11个菌株生产7-8.2% EPA。选择生产8.2% EPA的菌株用于通过两阶段生长程序(即48hrs MM,96hrs HGM)进一步分析。GC分析显示工程化的菌株生产占总脂质的约14%的EPA。该菌株称作菌株“M26”。
菌株M26相对于野生型解脂耶氏酵母ATCC #20362的最终基因型是:Pox3-,Y.Δ12-,FBA::F.Δ12::Lip2,FBAIN::MΔ12::Pex20,TEF::Δ6S::Lip1,FBAIN::Δ6B::Pex20,FBAIN::E1S::Pex20,GPAT::E1S::Xpr,TEF::E2S::Xpr,FBAIN::MAΔ5::Pex20,TEF::MAΔ5::Lip1,TEF::H Δ 5S::Pex16,FBAIN::Δ 17S::Lip2,FBAINm::Δ17S::Pex16,TEF::Δ 17S::Pex16和TEF::rELO2S::Pex20。
制备生产占总脂质的约14%的EPA的MU菌株
菌株MU是菌株M26的Ura营养缺陷型。该菌株是通过用PacI和HincII消化过的5μg质粒pZKUM(SEQ ID NO:137)转化菌株M26而制备的。用Frozen-EZ酵母转化试剂盒(Zymo ResearchCorporation,Orange,CA)进行转化,通过将100μl转化的细胞混合物在以下培养基中铺板在琼脂板上,选择转化体:6.7g/L酵母氮基(DIFCO Laboratories,Detroit,MI)、20g/右旋糖、50mg/L尿嘧啶和800mg/L FOA。7天后,将出现的小菌落铺板在MM和MMU琼脂板上。所有都是URA营养缺陷型。菌株之一命名为“MU”。
实施例13
高山被孢霉基因组DNA和cDNA的制备
本实施例描述了高山被孢霉(ATCC #16266)的基因组DNA和cDNA的制备。这使得能够分别按照实施例14、15、16、17和18的描述分离高山被孢霉LPAAT2、DGAT1、DGAT2、GPAT和ELO3。
制备高山被孢霉基因组DNA
使用QiaPrep Spin微量制备试剂盒(Qiagen,目录号627106),从高山被孢霉(ATCC #16266)分离基因组DNA。刮下在YPD琼脂平板(2%细菌用酵母提取物,3%细菌用肽胨,2%葡萄糖,2%细菌用琼脂)上生长的细胞,重新悬浮于1.2mL试剂盒缓冲液P1。将重新悬浮的细胞置于2个2.0mL螺旋帽试管中,每个试管装有0.6mL玻璃珠(0.5mm直径)。在Biospec(Bartlesville,OK)微珠搅拌机上,在HOMOGENIZE设置,匀浆细胞2min。然后在Eppendorf微量离心机中在14,000rpm离心试管2min。将上清液(0.75mL)转移至3个1.5mL微量离心试管。向每个试管中加入等体积的试剂盒缓冲液P2。通过反转混合试管3次后,将0.35mL缓冲液N3加入每个试管。通过反转混合试管5次,再次混合物内容物。在Eppendorf微量离心机中在14,000rpm离心混合物5min。将每个试管的上清液转移进分开的试剂盒离心柱中。然后对柱子进行以下步骤:离心(14,000rpm 1min),用缓冲液PE洗涤一次,离心(14,000rpm 1min),然后最后离心(14,000rpm 1min)。将缓冲液EB(50μl)加入每个柱中,静置1min。然后通过在14,000rpm离心1min,洗脱基因组DNA。
制备高山被孢霉cDNA根据制造商的方案,使用BD-Clontech Creator cDNA文库试剂盒(Mississauga,ON,Canada),制备了高山被孢霉cDNA。具体地,在23℃,将高山被孢霉菌株ATCC #16266在60mL YPD培养基(2%细菌用酵母提取物,3%细菌用肽胨,2%葡萄糖)中培养3天。通过在Beckman GH3.8转子中3750rpm离心10min,沉淀细胞,并重新悬浮于6X 0.6mL Trizole试剂(Invitrogen)。将重新悬浮的细胞转移到6个2mL螺旋帽试管中,每个试管装有0.6mL 0.5mm玻璃珠。在Biospec(Bartlesville,OK)微珠搅拌机上,在HOMOGENIZE设置,匀浆细胞2min。短时间离心试管,沉淀珠子。将液体转移至4个新的1.5mL微量离心试管,向每个试管中加入0.2mL氯仿/异戊醇(24:1)。用手摇动试管1min,静置3min。然后在4℃,在14,000rpm离心试管10min。将上层转移至4个新试管。将异丙醇(0.5mL)加入每个试管中。在室温温育试管15min,然后在14,000rpm、4℃离心10min。各用1mL 75%乙醇(由无RNA酶的水制备)洗涤沉淀,并在空气中干燥。然后,将总RNA样品重新溶于500μl水,使用1:50稀释的RNA样品,在A260nm测量RNA的量。得到共计3.14mg RNA。
根据制造商的方案,用Qiagen Rneasy总RNA Midi试剂盒进一步纯化该总RNA样品。从而,将总RNA样品稀释至2mL,并与含有80μl β-巯基乙醇和5.6mL 100%乙醇的8mL缓冲液RLT相混合。将样品分成4份,装载4 RNeasy midid柱。然后,在4500Xg离心柱5min。为了洗涤柱,装载2mL缓冲液RPE,在4500Xg离心柱2min。重复洗涤步骤1次,例外是,将离心延长至5min。如下洗脱总RNA:向每个柱中应用250μl无RNA酶的水,等待1min,在4500Xg离心3min。
按照Pharmacia的试剂盒手册,从上面的总RNA样品分离聚A(+)RNA。简而言之,使用2个寡-dT-纤维素柱。各用1mL高盐缓冲液,洗涤柱2次。将来自前一步骤的总RNA样品稀释至2mL总体积,并调节至10mM Tris/HCl,pH 8.0,1mM EDTA。在65℃加热样品5min,然后置于冰上。加入样品缓冲液(0.4mL),然后在重力进料下,将样品装载到2个寡-dT-纤维素柱上。在350Xg离心柱2min,各用0.25mL高盐缓冲液洗涤2次,每次洗涤后在350Xg离心2min。在相同的离心程序后,再用低盐缓冲液洗涤柱3次。通过各用0.25mL预热至65℃的洗脱缓冲液洗涤柱4次,洗脱聚(A)+RNA,随后进行相同的离心程序。重复整个纯化过程一遍。得到的纯化的聚(A)+RNA的浓度为30.4ng/μl。
使用BD-Clontech描述的LD-PCR方法和0.1μg聚A(+)RNA样品,制备cDNA。更具体地,就第1链cDNA合成而言,将3μl聚(A)+RNA样品与1μl SMART IV寡核苷酸(SEQ ID NO:281)和1μl CDSIII/3’PCR引物(SEQ ID NO:282)相混合。在72℃加热混合物2min,在冰上冷却2min。向试管中加入下述物质:2μl第1链缓冲液,1μl 20mM DTT,1μl 10mM dNTP混合物和1μlPowerscript逆转录酶。在42℃温育混合物1小时,然后在冰上冷却。
将第1链cDNA合成混合物用作PCR反应的模板。更具体地,反应混合物含有下述物质:2μl第1链cDNA混合物,2μl5’-PCR引物(SEQ ID NO:283),2μl CDSIII/3’-PCR引物(SEQ ID NO:282),80μl水,10μl 10X Advantage 2 PCR缓冲液,2μl 50X dNTP混合物和2μl 50X Advantage 2聚合酶混合物。将热循环仪条件设定为:95℃ 20sec,然后是在GenAmp 9600仪器上的14个95℃ 5sec和68℃ 6min的循环。通过琼脂糖凝胶电泳和溴化乙锭染色,定量PCR产物。
将75μl上述PCR产物(cDNA)与3μl 20μg/μl蛋白酶K(与试剂盒一起提供)相混合。在45℃温育混合物20min,然后加入75μl水,用150μl苯酚:氯仿:异戊醇混合物(25:24:1)提取混合物。用150μl氯仿:异戊醇(25:1)进一步提取水相。然后,将水相与15μl 3M醋酸钠,2μl 20μg/μl糖原和400μl 100%乙醇相混合。立即在离心机中在室温在14000rpm离心混合物20min。用150μl 80%乙醇洗涤沉淀一次,在空气中干燥,溶于79μl水。
随后用SfiI消化溶解的cDNA(将79μl cDNA与10μl 10X SfiI缓冲液,10μl SfiI酶和1μl 100X BSA相混合,在50℃温育混合物2小时)。加入二甲苯蓝染料(2μl 1%)。然后完全按照制造商的方案,在与试剂盒一起提供的Chroma Spin-400柱上分离混合物。通过琼脂糖凝胶电泳,分析从柱收集的级分。合并含有cDNA的前3个级分,并用乙醇沉淀cDNA。将沉淀的cDNA重新溶于7μl水,并连接进试剂盒-提供的pDNR-LIB。
文库测序
将连接产物用于转化大肠杆菌XL-1 Blue电穿孔感受态细胞(Stratagene)。得到估计共2 x 106菌落。使用M13正向引物(SEQ IDNO:284),通过Agencourt Bioscience Corporation(Beverly,MA),进行cDNA文库的测序。
实施例14
高山被孢霉LPAAT2表达增加PUFAs百分比
本实施例描述经过转化而共表达高山被孢霉LPAAT2(SEQ IDNOs:69和70)的解脂耶氏酵母菌株Y2067U(实施例10)中EPA生物合成和积累的增加。认为如果类似地共表达高山被孢霉LPAAT2(如在菌株Y2034、Y2047和/或Y2214中),则经过工程化以通过Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径生产ARA的解脂耶氏酵母宿主菌株能够证明ARA生物合成和积累的增加。
按照如下方法克隆高山被孢霉LPAAT2 ORF。通过PCR,用引物MLPAT-F和MLPAT-R(SEQ ID NOs:285和286)扩增来自高山被孢霉的cDNA(实施例13)的LPAAT2 ORF。反应混合物含有1μlcDNA、每种引物各1μl、22μl水和25μl ExTaq预混合2X Taq PCR溶液(TaKaRa Bio Inc.,Otsu,Shiga,520-2193,Japan)。按照如下方法进行扩增:最初在94℃变性150sec,然后是30个循环的94℃下变性30sec、55℃下退火30sec和72℃下延伸90sec。72℃进行最终的延伸循环10分钟,然后在4℃终止反应。从PCR反应获得了约950bp DNA片段。根据制造商的方案,用Qiagen(Valencia,CA)PCR纯化试剂盒对其进行纯化。用NcoI和NotI消化纯化的PCR产物,克隆到Nco I-Not I切割的pZUF17载体(SEQ ID NO:118;图8B)中,使得基因处于自主复制的载体中的解脂耶氏酵母FBAIN启动子和PEX20-3’终止子区控制下,以便在解脂耶氏酵母中表达。通过微量制备DNA的限制分析证实正确的转化体,得到的质粒命名为“pMLPAT-17”(SEQ ID NO:138)。
为了将高山被孢霉LPAAT2整合到解脂耶氏酵母的基因组中,建立了质粒pMLPAT-Int。用引物LPAT-Re-5-1和LPAT-Re-5-2(SEQID NOs:287和288)扩增1129bp DNA片段YLPAT-5’(SEQ ID NO:289),该片段含有解脂耶氏酵母LPAAT1(SEQ ID NO:71)的AUG上游紧邻的解脂耶氏酵母基因组1103bp片段。反应混合物含有1μl解脂耶氏酵母基因组DNA、每种引物各1μl、22μl水和25μl ExTaq预混合2X Taq PCR溶液(TaKaRa)。按照上文进行扩增。从PCR反应获得了约1130bp DNA片段。根据制造商的方案,用Qiagen的PCR纯化试剂盒对其进行纯化。用SalI和ClaI消化纯化的PCR产物,克隆到SalI-ClaI切割的pBluescript SK(-)载体中,得到质粒“pYLPAT-5’”。
然后用引物LPAT-Re-3-1和LPAT-Re-3-2(SEQ ID NOs:290和291)扩增938bp片段YLPAT-3’(SEQ ID NO:292),该片段含有解脂耶氏酵母LPAAT1的终止密码子后紧邻的解脂耶氏酵母基因组903bp片段。用ClaI和XhoI消化纯化的PCR产物,克隆到ClaI-XhoI消化的pYLPAT-5’中。通过微量制备分析证实正确的转化体,得到的质粒命名为“pYLPAT-5’-3’”。
然后用ClaI和NotI消化pMLPAT-17(SEQ ID NO:138),根据制造商的方案,用Qiagen QiaexII凝胶纯化试剂盒分离含有解脂耶氏酵母Ura3基因的约3.5kb片段、解脂耶氏酵母FBAIN启动子和高山被孢霉LPAAT2基因。将该片段克隆到ClaI-NotI消化的pYLPAT-5’-3’中。通过微量制备和限制分析证实正确的转化体。得到的质粒命名为“pMLPAT-Int”(SEQ ID NO:139)。
如下制备“对照”载体pZUF-MOD-1(SEQ ID NO:140;图15B)。首先,用引物pzuf-mod1和pzuf-mod2(SEQ ID NOs:293和294)扩增252bp“填充”DNA片段。用Qiagen QiaQuick PCR纯化试剂盒纯化扩增的片段,使用标准条件,用NcoI和NotI消化,然后再用QiaQuick PCR纯化试剂盒进行纯化。将该片段连接进类似地消化的NcoI-/NotI-切割的pZUF17载体(SEQ ID NO:118;图8B),并将得到的连接混合物用于转化大肠杆菌Top10细胞(Invitrogen)。使用Qiagen QiaPrep Spin微量制备试剂盒,从4个得到的菌落纯化质粒DNA。用NcoI和NotI消化纯化的质粒,证实约250bp片段的存在。得到的质粒称作“pZUF-MOD-1”(SEQ ID NO:140)。
根据一般方法,分别用质粒pMLPAT-17、质粒pZUF-MOD-1(对照)和SpeI/XbaI消化的质粒pMLPAT-Int转化解脂耶氏酵母菌株Y2067U(来自实施例10,生产占总脂质的14%的EPA)。使转化体在添加了氨基酸的合成MM中生长2天,然后在HGM中生长4天。基于GC分析(如一般方法中所述),在下表中示出了两个含有pZUF-MOD-1的转化体、两个含有pMLPAT-17的转化体,和两个具有整合到基因组中的pMLPAT-Int的转化体的脂肪酸谱。脂肪酸鉴定为18:0、18:1(油酸)、18:2(LA)、GLA、DGLA、ARA、ETA和EPA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表34
经过工程化而超量表达高山被孢霉LPAAT2的耶氏酵母菌株Y2067U中的脂质组成
如上文所证明的,从pMLPAT-17表达高山被孢霉LPAAT2,使“对照”菌株中的EPA百分比从约14%增加到15.5-16%。当用pMLPAT-Int将高山被孢霉LPAAT2整合到基因组中时,EPA额外增加到16.6-17.3%。如果天然解脂耶氏酵母LPAAT1(SEQ ID NOs:71和72)和/或LPAAT2(SEQ ID NOs:74和75)从例如菌株Y2067U+pMLPAT-Int中敲除,可以预期进一步的增加。
实施例15
高山被孢霉DGAT1表达增加PUFAs百分比
本实施例描述经过转化而共表达高山被孢霉DGAT1 cDNA(SEQID NO:83)的解脂耶氏酵母菌株Y2067U(实施例10)中EPA生物合成和积累的增加。认为如果类似地共表达高山被孢霉DGAT1(如在菌株Y2034、Y2047和/或Y2214中),则经过工程化以通过Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径生产ARA的解脂耶氏酵母宿主菌株能够证明ARA生物合成和积累的增加。
如下克隆高山被孢霉DGAT1 ORF。首先,为了辅助克隆cDNA,将DGAT1的第2个密码子的序列从‘ACA’改变成‘GCA’,导致从苏氨酸至丙氨酸的氨基酸改变。这通过用引物MACAT-F1和MACAT-R(SEQ ID NO:295和296)扩增高山被孢霉DGAT1 ORF的完整编码区来实现。更具体地,PCR反应混合物含有1μl各20μM引物MACAT-F1和MACAT-R溶液,1μl高山被孢霉cDNA(同上,实施例13),22μl水和25μl ExTaq预混合2X Taq PCR溶液(TaKaRaBio Inc.,Otsu,Shiga,520-2193,Japan)。如下进行扩增:在94℃初步变性150sec,随后30个下述循环:在94℃变性30sec,在55℃退火30sec,在72℃延伸90sec。进行在72℃ 10min的最终延伸循环,然后在4℃终止反应。从PCR反应得到约1600bp DNA片段。根据制造商的方案,使用Qiagen的PCR纯化试剂盒,进行纯化。
将高山被孢霉DGAT1 ORF插入Nco I-和Not I-消化的质粒pZUF17(SEQ ID NO:118;图8B),使得ORF克隆在FBAIN启动子和PEX20-3’终止子区域控制下。但是,由于DGAT1 ORF含有内部NcoI位点,克隆必需进行2个分开的限制酶消化。首先,用BamHI和Nco I消化约2μg纯化的PCR产物。反应混合物含有20U每种酶(Promega)和6μl限制缓冲液D,总体积为60μl。在37℃温育混合物2小时。通过琼脂糖凝胶电泳分离约320bp片段,并使用QiagenQiaex II凝胶纯化试剂盒进行纯化。单独地,使用与上面相同的反应条件,例外是用Not I替代Nco I,用BamHI和Not I消化约2μg纯化的PCR产物,如上分离和纯化约1280bp片段。最后,用Nco I和Not I消化约3μg pZUF17,并如上纯化,制备约7kB片段。
在三向连接中,将约7kB Nco I/Not I pZUF17片段、约320bp NcoI/BamHI DGAT1片段和约1280bp BamHI/Not I DGAT1片段连接到一起,在室温温育过夜。连接混合物含有100ng 7kB片段以及320bp和1280bp片段各200ng、2μl连接酶缓冲液和2U T4 DNA连接酶(Promega),总体积为20μl。根据制造商的方案,将连接产物用于转化大肠杆菌Top10化学感受态细胞(Invitrogen)。
将转化的单个菌落(共12个)用于接种培养物,用于微量制备分析。限制图谱和测序证实,12个菌落中的5个菌落携带需要的质粒,其称作“pMDGAT1-17”(图15C;SEQ ID NO:141)。
根据一般方法,分别用pMDGAT1-17和pZUF-MOD-1(上文,实施例14)转化解脂耶氏酵母菌株Y2067U(来自实施例10)。使转化体在添加了氨基酸的合成MM中生长2天,然后在HGM中生长4天。基于GC分析(如一般方法中所述),在下表35中示出了两个含有pMDGAT1-17的转化体和两个含有pZUF-MOD-1的转化体的脂肪酸谱。脂肪酸鉴定为18:0、18:1(油酸)、18:2(LA)、GLA、DGLA、ARA、ETA和EPA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表35
经过工程化而超量表达高山被孢霉DGAT1的耶氏酵母菌株Y2067U中的脂质组成
如上文证明的,从质粒pMDGAT1-17表达高山被孢霉DGAT1,分别使“对照”菌株中的EPA百分比从约13.3%增加到约14%(“Y2067U+pMDGAT1-17 #1”)和约15.1%(“Y2067U+pMDGAT1-17#2”)。如果天然解脂耶氏酵母DGAT1(SEQ ID NOs:81和82)从例如菌株Y2067U+pMDGAT1-17中敲除,可以预期EPA的进一步增加。
实施例16
高山被孢霉DGAT2增加PUFAs百分比
本实施例描述经过转化而共表达高山被孢霉DGAT2 cDNA(SEQID NO:95)的解脂耶氏酵母菌株Y2067U(实施例10)中EPA生物合成和积累的增加。认为如果类似地共表达高山被孢霉DGAT2(如在菌株Y2034、Y2047和/或Y2214中),则经过工程化以通过Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径生产ARA的解脂耶氏酵母宿主菌株能够证明ARA生物合成和积累的增加。
如下将高山被孢霉DGAT2 ORF克隆到质粒pZUF17中。首先,用来自高山被孢霉cDNA(上文实施例13)的引物MDGAT-F和MDGAT-R1(SEQ ID NOs:297和298)PCR扩增ORF。分离预期的1015bp片段,纯化,用Nco I和Not I消化,克隆到Nco I-Not I切割的pZUF17载体(SEQ ID NO:118;图8B)中,使得基因在解脂耶氏酵母FBAIN启动子和PEX20-3’终止子区域控制下。通过微量制备DNA的限制分析证实正确的转化体,得到的质粒命名为“pMDGAT2-17”(SEQ ID NO:142)。
根据一般方法,分别用pMDGAT2-17和pZUF-MOD-1(上文,实施例14)转化解脂耶氏酵母菌株Y2067U(来自实施例10)。使转化体在添加了氨基酸的合成MM中生长2天,然后在HGM中生长4天。基于GC分析(如一般方法中所述),下文示出了两个含有pMDGAT2-17的转化体和两个含有pZUF-MOD-1的转化体的脂肪酸谱。脂肪酸鉴定为18:0、18:1(油酸)、18:2(LA)、GLA、DGLA、ARA、ETA和EPA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表36
经过工程化而超量表达高山被孢霉DGAT2的耶氏酵母菌株Y2067U中的脂质组成
如上文证明的,从质粒pMDGAT2-17表达高山被孢霉DGAT2,使“对照”菌株中的EPA百分比从约13.3%增加到约15.25%(“Y2067U+pMDGAT2-17)。如果天然解脂耶氏酵母DGAT2(SEQID NOs:89-94)从例如菌株Y2067U+pMDGAT2-17中敲除,可以预期EPA的进一步增加。
实施例17
高山被孢霉GPAT增加PUFAs百分比
本实施例描述经过转化而共表达高山被孢霉GPAT ORF(SEQ IDNO:97)的解脂耶氏酵母菌株Y2107U1(实施例11)中DGLA生物合成和积累的增加(以及18:1的量减少)。认为如果类似地共表达高山被孢霉GPAT(如在菌株Y2034、Y2047和/或Y2214中),则经过工程化以通过Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径生产ARA的解脂耶氏酵母宿主菌株能够证明ARA生物合成和积累的增加。
基于来自构巢曲霉(GenBank获取号EAA62242)和粗糙链孢霉(GenBank获取号XP_325840)的GPAT的序列,设计了以下引物进行简并PCR:
MGPAT-N1(SEQ ID NO:299)CCNCAYGCNAAYCARTTYGT
MGPAT-NR5(SEQ ID NO:300)TTCCANGTNGCCATNTCRTC
[注:用于SEQ ID NOs:299和300的核酸简并密码如下:
R=A/G;Y=C/T;并且N=A/C/T/G。]
用TaKaRa ExTaq预混合Taq聚合酶(TaKaRa Bio Inc.,Otsu,Shiga,Japan),在Perkin Elmer GeneAmp 9600 PCR仪中进行PCR扩增。扩增如下进行:30个循环的94℃下变性30sec、55℃下退火30sec和72℃下延伸90sec。然后72℃进行最终的延伸循环7分钟。
获得了约1.2kB的片段(SEQ ID NO:99)。用Qiagen QiaQuickPCR纯化试剂盒纯化该片段,克隆到克隆载体pCR2.1-TOPO(Invitrogen)中,并且测序。得到的序列翻译后基于BLAST程序分析与已知的GPATs具有同源性。
基于1212bp cDNA片段的序列,通过PCR扩增和基因组步移技术克隆了高山被孢霉GPAT的5’和3’区。这使得能够组装相应于高山被孢霉GPAT(SEQ ID NO:100)的-1050bp至+2885bp区的重叠群。该重叠群包括GPAT的整个编码区和四个内含子(SEQ IDNOs:104,105,106和107)。
具体地,将描述于实施例13(1μl)的高山被孢霉cDNA样品用作扩增GPAT的3’末端的模板。MGPAT-5N1(SEQ ID NO:301)和CDSIII/3’(SEQ ID NO:282)用作模板。用TaKaRa ExTaq预混合Taq聚合酶(TaKaRa Bio Inc.,Otsu,Shiga,Japan),在PerkinElmer GeneAmp 9600 PCR仪中进行PCR扩增。扩增如下进行:30个循环的94℃下变性30sec、55℃下退火30sec和72℃下延伸90sec。然后72℃进行最终的延伸循环7分钟。
1:10稀释PCR产物,将1μl稀释的PCR产物用作第二轮扩增的模板,该第二轮扩增采用MGPAT-5N2(SEQ ID NO:302)和CDSIII/3’作为引物。条件与上文描述的完全相同。再次将第二轮PCR产物1:10稀释,将1μl稀释的PCR产物用作第三轮PCR的模板,该第三轮PCR采用MGPAT-5N3(SEQ ID NO:303)和CDSIII/3’作为引物。PCR条件再次相同。
第三轮PCR产生约1kB的片段。用Qiagen QiaQuick PCR纯化试剂盒纯化该片段,克隆到pCR2.1-TOPO载体中,进行序列分析。序列分析结果表明该965bp片段(SEQ ID NO:101)相应于GPAT基因的3’末端。
用Clontech Universal GenomeWalkerTM试剂盒获得一段相应于高山被孢霉GPAT的5’末端区的基因组DNA。简言之,用DraI、EcoRV、PvuII或StuI单独地消化2.5μg每个高山被孢霉基因组DNA,使用Qiagen Qiaquick PCR纯化试剂盒,纯化消化的DNA样品,并用每种试剂盒缓冲液EB各30μl洗脱,并用如下所示的基因组步移接头(SEQ ID NO:304[上链]和305[下链]),连接纯化的样品:
5’-GTAATACGACTCACTATAGGGCACGCGTGGTCGACGGCCCGGGCTGGT-3’
3’-H2N-CCCGACCA-5’
每种连接反应混合物含有1.9μl 25μM基因组步移接头,1.6μl10X连接缓冲液,0.5μl T4 DNA连接酶和4μl纯化的消化的基因组DNA样品之一。在16℃温育反应混合物过夜。通过在70℃温育5min,终止反应。然后,将72μl 10mM TrisHCl,1mM EDTA,pH 7.4缓冲液加入每种连接反应混合物。
进行了四个独立的PCR反应,每个反应采用四种连接混合物之一作为模板。PCR反应混合物含有1μl连接混合物,0.5μl 20μMMGPAT-5-1A(SEQ ID NO:306),1μl 10μM试剂盒引物AP1(SEQID NO:307),22.5μl μl水和25μl ExTaq预混合Taq 2X PCR溶液(TaKaRa)。采用以下条件进行32轮PCR扩增:30个循环的94℃变性30sec、55℃退火30sec和72℃延伸180sec。72℃最终延伸7min,随后在4℃终止反应。
分别以1:50稀释每个PCR反应的产物,用作第二轮PCR的模板。每个反应混合物含有1μl稀释的PCR产物之一作为模板,0.5μl 20μM MGPAT-3N1(SEQ ID NO:308),21μl 10μM试剂盒引物AP2(SEQ ID NO:309),22.5μl水和25μl of ExTaq预混合Taq 2X PCR溶液(TaKaRa)。用与上文所述相同的热循环条件进行32轮PCR反应。
从第二轮PCR获得DNA片段。纯化该片段,克隆到pCR2.1-TOPO中并测序。测序分析显示1908bp片段(SEQ ID NO:102)是高山被孢霉GPAT基因的5’末端。
类似地,按照上文所述,通过两轮基因组步移获得966 bp片段(SEQ ID NO:103),不同的是采用引物MGPAT-5N1作为基因特异性引物进行第一轮PCR,用引物MGPAT-5N2作为基因特异性引物进行第二轮。也纯化该片段,克隆到pCR2.1-TOPO中并测序。序列分析显示它含有GPAT基因的一部分;但是,该片段的长度不足以延伸基因的任何一个末端。与3’cDNA序列(SEQ ID NO:101)的比较显示没有包括ORF的最后171bp。
组装来自高山被孢霉的全长GPAT序列
从上文描述的最初的部分cDNA片段(SEQ ID NO:99)、3’cDNA片段(SEQ ID NO:101)、内部基因组片段(SEQ ID NO:103)和5’基因组片段(SEQ ID NO:102)组装含有完整GPAT基因(包括在GPAT翻译起始‘ATG’密码子上游延伸1050个碱基,并且延伸超过GPAT终止密码子22个碱基的区域)的3935bp序列(SEQ ID NO:100)(图示于图16)。该区域中包括了2151bp GPAT ORF。从‘ATG’到终止密码子‘TAG’的高山被孢霉GPAT ORF的完整核苷酸序列提供为SEQ ID NO:97(相应于SEQ ID NO:100的碱基1050-2863,排除4个内含子(即内含子1[SEQ ID NO:104],相应于SEQ ID NO:100的碱基1195-1469;内含子2[SEQ ID NO:105],相应于SEQ IDNO:100的碱基1585-1839;内含子3[SEQ ID NO:106],相应于SEQID NO:100的碱基2795-2877和内含子4[SEQ ID NO:107],相应于SEQ ID NO:100的碱基2940-3038)。翻译的氨基酸序列(SEQ IDNO:98)显示出与许多真菌、植物和动物GPATs的同源性。
更具体地,通过进行BLAST(Basic Local Alignment SearchTool;Altschul,S.F.,et al.,J.Mol.Biol.215:403-410(1993))检索确定序列同一性。此处描述为SEQ ID NO:98的氨基酸片段与推定的玉蜀黍黑粉菌的GPAT(GenBank获取号EAK84237)的蛋白序列具有47%同一性和65%相似性,预期值是1e-152;此外,SEQ IDNO:111与烟曲霉的GPAT(GenBank获取号EAL20089)具有47%同一性和62%相似性,预期值是1e-142。
构建包含FBAIN::MGPAT::PEX20-3’嵌合基因的质粒pMGPAT-17
如下克隆高山被孢霉GPAT ORF。用引物MGPAT-cDNA-5和MGPAT-cDNA-R(SEQ ID NOs:310和311)通过PCR从cDNA扩增GPAT ORF。反应混合物包含1μl cDNA、每种引物各1μl、22μl水和25μl ExTaq预混合2X Taq PCR溶液(TaKaRa Bio Inc.,Otsu,Shiga,520-2193,Japan)。如下进行扩增:在94℃初步变性150sec,随后30个下述循环:在94℃变性30sec,在55℃退火30sec,在72℃延伸90sec。进行在72℃ 10min的最终延伸循环,然后在4℃终止反应。从PCR反应得到约2.2kB DNA片段。根据制造商的说明,用Qiagen PCR纯化试剂盒纯化该片段。
用BamHI和EcoRI消化纯化的PCR产物,通过凝胶琼脂糖电泳分离约470bp的片段,用Qiagen凝胶纯化试剂盒纯化。独立地,也用EcoRI和NotI切割PCR产物,按照上文分离和纯化1.69kB片段。将两个片段连接到BamHI和NotI切割的pZUF-MOD-1载体(SEQID NO:140;图15B)中,使得基因处于自主复制的载体中解脂耶氏酵母FBAIN启动子和PEX20-3’终止子区的控制下,以便在解脂耶氏酵母中表达。通过微量制备DNA的限制分析证实正确的转化体,得到的质粒命名为“pMGPAT-17”(SEQ ID NO:143;图15D)。
分析超量表达高山被孢霉GPAT的转化的解脂耶氏酵母中的脂质组成
根据一般方法,分别用质粒pMGPAT-17和pZUF-MOD-1(上文,实施例14)转化解脂耶氏酵母菌株Y2107U1(来自实施例11)。使转化体在添加了氨基酸的合成MM中生长2天,然后在HGM中生长4天。基于GC分析(如一般方法中所述),下文示出了两个含有pZUF-MOD-1的转化体和两个含有pMGPAT-17的转化体的脂肪酸谱。脂肪酸鉴定为18:0、18:1(油酸)、18:2(LA)、GLA、DGLA、ARA、ETA和EPA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表37
经过工程化而超量表达高山被孢霉GPAT的耶氏酵母菌株Y2107U1中的脂质组成
如上文证明的,从pMGPAT-17表达高山被孢霉GPAT,使“对照”菌株中的DGLA百分比从约2.5%增加到约6.5%。18:1的水平从约23%减少到约16%。如果天然解脂耶氏酵母GPAT从表达pMGPAT-17的转化菌株中敲除,可以预期DGLA(或任何其它下游PUFAs)的进一步增加。
实施例18
高山被孢霉脂肪酸延伸酶“ELO3”增加PUFAs百分比
本实施例描述经过转化而共表达高山被孢霉C16/18脂肪酸延伸酶(“ELO3”;SEQ ID NOs:53和54)的解脂耶氏酵母菌株Y2031(实施例5)中生产35%以上的C18脂肪酸(18:0、18:1、18:2和GLA)和31%以下的C16脂肪酸。认为ELO3(其可以任选是密码子优化的,以便增加表达)可以促使碳流入工程化的Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径,作为增加需要的PUFA即ARA的生产的手段。例如,包含该C16/18脂肪酸延伸酶的嵌合基因可以容易地导入例如菌株Y2034、Y2047或Y2214中。
高山被孢霉C16/18脂肪酸延伸酶的序列鉴定
从9,984种高山被孢霉cDNA序列(实施例13)鉴定编码一部分高山被孢霉脂肪酸延伸酶的cDNA片段(SEQ ID NO:55)。该cDNA片段与许多脂肪酸延伸酶具有显著同源性,从而鉴定为延伸酶。
根据同一性百分比、相似性百分比和预期值报道了概括与SEQ IDNO:55具有最多相似性的序列的BLAST比较的结果。具体地,SEQID NO:55的翻译的氨基酸序列与来自白色假丝酵母SC5314的可能的脂肪酸延伸酶(GenBank获取号EAL04510.1,其中标注为与啤酒糖酵母EUR4、FEN1和ELO1相似的三种可能的脂肪酸延伸酶基因之一)的蛋白序列具有32%同一性和46%相似性,预期值是4e-13。此外,SEQ ID NO:55与来自啤酒糖酵母的ELO1(GenBank获取号NC_001142,X染色体的碱基67849-68781)具有35%同一性和53%相似性。啤酒糖酵母ELO1描述为中等链酰基延伸酶,其催化不饱和C12-C16脂肪酰基-CoAs在羧基末端延伸为C16-C18脂肪酸。
基于上文报道的同源性,SEQ ID NO:55的解脂耶氏酵母基因产物在此命名为“延伸酶3”或“ELO3”。
分析部分脂肪酸延伸酶cDNA序列(SEQ ID NO:55)表明,5’和3’末端都是不完整的。为了获得缺失的高山被孢霉ELO3的3’区,采用了Clontech Universal GenomeWalkerTM试剂盒(如实施例17的描述)。具体地,在第一轮PCR中采用了与上文所述相同的一组四种连接混合物,采用了相同的成分和条件,不同之处是将MA Elong 3’1(SEQ ID NO:312)和AP1用作引物(即代替引物MGPAT-5-1A和AP1)。第二轮PCR采用MA Elong 3’2(SEQ ID NO:313)和AP2作为引物。从第二轮PCR获得了1042bp DNA片段(SEQ ID NO:56)。纯化该片段,克隆到pCR2.1-TOPO中并测序。序列分析表明该片段含有ELO3的3’末端,包括基因的‘TAA’终止密码子下游的约640bp。
也采用与用于Clontech 3’-末端RACE(上文)中相同的一组四种连接混合物获得了高山被孢霉ELO3的5’末端区。具体地,进行了采用与上文所述相同的成分和条件的第一轮PCR,不同之处是将MAElong 5’1(SEQ ID NO:314,在5’末端嵌套)和AP1用作引物(即代替引物MA Elong 3’1和AP1)。第二轮PCR采用MA Elong 5’2(SEQID NO:315,在5’末端嵌套)和AP2作为引物。获得了2223bp DNA片段(SEQ ID NO:57)。对其进行纯化,克隆到pCR2.1-TOPO中并测序。序列分析显示它含有ELO3基因的5’区。
这样,通过组合最初的部分cDNA序列(SEQ ID NO:55)和通过基因组步移获得的5’和3’序列(分别是SEQ ID NOs:57和56,图示于图17),获得了高山被孢霉ELO3(SEQ ID NO:53)的完整cDNA序列。这产生了3557bp的序列,在此鉴定为SEQ ID NO:58,其包含ELO3的推定的‘ATG’翻译起始密码子上游的2091bp;828bpELO3 ORF(即SEQ ID NO:53,相应于SEQ ID NO:58的碱基2092-2919);和ELO3终止密码子的下游的638 bp(相应于SEQ IDNO:58的碱基2920-3557)。
高山被孢霉ELO3的相应基因组序列比SEQ ID NO:58中提供的cDNA片段长。具体地,发现在ORF的318bp处含有ELO3基因的基因组DNA中存在542bp内含子(SEQ ID NO:59);因此,在此提供于SEQ ID NO:60中的基因组DNA片段是4,099bp(图17)。
基于BLAST程序分析,翻译的ELO3蛋白序列(SEQ ID NO:54)具有以下同源性:与来自白色假丝酵母SC5314的可能的脂肪酸延伸酶(GenBank获取号EAL04510.1)具有37%同一性和51%相似性,预期值是4e-43。此外,翻译的ELO3与来自粗糙链孢霉的XP_331368(在此标注为“假定蛋白”)的蛋白序列具有33%同一性和44%相似性,预期值是3e-44。
构建包含FBAIN::ELO3::PEX16-3’嵌合基因的质粒pZUF6S-E3WT
如下克隆高山被孢霉脂肪酸ELO3 ORF。用引物MA Elong 5’NcoI 3和MA Elong 3’NotI(SEQ ID NOs:316和317)通过PCR从高山被孢霉cDNA(实施例13)扩增ELO3 ORF。反应混合物包含1μl cDNA、每种引物各1μl、22μl水和25μl ExTaq预混合2X TaqPCR溶液(TaKaRa Bio Inc.,Otsu,Shiga,520-2193,Japan)。如下进行扩增:在94℃初步变性30sec,随后32个下述循环:在94℃变性30sec,在55℃退火30sec,在72℃延伸120sec。进行在72℃ 10min的最终延伸循环,然后在4℃终止反应。从PCR反应得到约830bp DNA片段。根据制造商的说明,用Qiagen(Valencia,CA)PCR纯化试剂盒纯化该片段。将纯化的PCR产物分成两个等分物,用NcoI和NspI消化一个等分物,用NspI和NotI消化另一个等分物。通过三段连接,将约270bp NcoI-NspI和约560bp NspI-NotI片段克隆到Nco I-Not I切割的pZF5T-PPC载体(图11C;SEQ ID NO:122)中,使得基因处于自主复制的载体中解脂耶氏酵母FBAIN启动子和PEX16-3’终止子区(GenBank获取号U75433)的控制下,以便在解脂耶氏酵母中表达。通过微量制备物分析证实正确的转化体,得到的质粒命名为“pZF5T-PPC-E3”(SEQ ID NO:144)。
用ClaI和PacI消化质粒pZF5T-PPC-E3,用Qiagen凝胶提取试剂盒从琼脂糖凝胶纯化约2.2kB的条带(即FBAIN::ELO3::PEX16-3’片段)。将该片段克隆到ClaI-PacI切割的pZUF6S(图18A;SEQ ID NO:145)中,其是一种自主复制的质粒,含有FBAIN启动子和Pex20-3’终止子(即FBAIN::D6S::Pex20嵌合基因)控制下的高山被孢霉Δ6去饱和酶ORF(“D6S”;GenBank获取号AF465281)和Ura3基因。通过微量制备物分析证实正确的转化体,得到的质粒命名为“pZUF6S-E3WT”(图18B;SEQ ID NO:146)。
分析超量表达高山被孢霉ELO3的转化的解脂耶氏酵母中的脂质组成
根据一般方法,用质粒pZUF6S(对照,包含FBAIN::D6S::Pex20嵌合基因)和质粒pZUF6S-E3WT(包含FBAIN::D6S::Pex20嵌合基因和FBAIN::ELO3::PEX16嵌合基因)转化解脂耶氏酵母菌株Y2031(实施例53)。使转化体在添加了氨基酸的合成MM中生长2天,然后在HGM中生长4天。基于GC分析(如一般方法中所述),下表38示出了6个含有pZUF6S的克隆(1-6号克隆,来自单次转化)和22个含有pZUF6S-E3WT的克隆(来自4次不同的转化[即3、5、6和7号])的脂肪酸谱。脂肪酸鉴定为16:0(棕榈酸)、16:1(棕榈油酸)、18:0、18:1(油酸)、18:2(LA)和GLA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表38
经过工程化而超量表达高山被孢霉ELO3的耶氏酵母菌株Y2031中的脂质组成
一些样品(以粗体和斜体标出)偏离预期读数。具体地,Y2031+pZUF6S-E3WT #3-3和Y2031+pZUF6S-E3WT #5-6都不生产GlA。类似地,Y2031+pZUF6S-E3WT #7-1、#7-3和#7-4具有GC误差,其中GC读出的16:0和16:1峰是单峰。由于这些变异结果,表39报道了对照和表达ELO3的转化菌株中的平均脂质。具体地,表39示出了表38中的脂肪酸谱的平均值,当计算这些平均值时,没有包括表38中用粗体和斜体表示的有错误的行。“总C16”表示16:0和16:1的平均值的和,而“总C18”表示18:0、18:1、18:2和GLA的平均值的和。
表39
经过工程化而超量表达高山被孢霉ELO3的耶氏酵母菌株Y2031中的脂质组成
基于上面报道的数据,当与解脂耶氏酵母菌株Y2031中的高山被孢霉Δ6去饱和酶共表达时,相对于仅仅超量表达高山被孢霉Δ6去饱和酶的Y2031的对照菌株,高山被孢霉ELO3的超量表达导致C18的百分比增加,C16的百分比减少。这表明高山被孢霉ELO3实际上是C16/18脂肪酸延伸酶。
实施例19
耶氏酵母C16/18脂肪酸延伸酶“YE2”增加PUFAs百分比
本实施例描述经过转化而共表达解脂耶氏酵母C16/18脂肪酸延伸酶(“YE2”;SEQ ID NO:62)的解脂耶氏酵母菌株Y2031(实施例5)中GLA生物合成和积累的增加。认为YE2延伸酶可以促使碳流入工程化的Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径,作为增加需要的PUFA即ARA的生产的手段。例如,包含该C16/18脂肪酸延伸酶的嵌合基因可以容易地导入例如菌株Y2034、Y2047或Y2214中。
解脂耶氏酵母C16/18脂肪酸延伸酶的序列鉴定
采用大鼠Elo2 C16/18脂肪酸延伸酶蛋白序列(GenBank获取号AB071986;SEQ ID NO:51)作为询问序列,通过序列比较鉴定了来自解脂耶氏酵母的新的脂肪酸延伸酶候选物。具体地,用这种rElo2询问序列检索GenBank和“Yeast project Genolevures”的公共解脂耶氏酵母蛋白数据库(Center for Bioinformatics,LaBRI,TalenceCedex,France)(也参见Dujon,B.et al.,Nature 430(6995):35-44(2004))。这导致鉴定了标注为“未命名蛋白产物”的同源序列,即GenBank获取号CAG77901(SEQ ID NOs:61和62)。该基因命名为YE2。
用BLAST算法(Altschul,S.F.,et al.,Nucleic Acids Res.25:3389-3402(1997))比较耶氏酵母YE2氨基酸序列与公共数据库,发现最相似的已知氨基酸序列是来自白色假丝酵母SC5314的氨基酸序列(SEQ ID NO:63,GenBank获取号EAL04510),其标注为可能的脂肪酸延伸酶。这种蛋白具有约40%同一性,评分为236,E值为7e-61。
分离耶氏酵母YE2基因
采用耶氏酵母基因组DNA作为模板,寡核苷酸YL597和YL598(SEQ ID NOs:318和319)作为引物,通过PCR扩增YE2基因的编码区。按照一般方法的描述,在50μl总体积中进行PCR反应。热循环仪条件设定为35个循环的95℃ 1min、56℃ 30sec、72℃ 1min,然后72℃下最终延伸10min。纯化YE2编码区的PCR产物,用NcoI/NotI消化,然后与NcoI/NotI消化的pZKUGPYE1-N(下文实施例20;也参见图18C,SEQ ID NO:147)连接在一起,得到pZKUGPYE2(图18D,SEQ ID NO:148)。在“ATG”翻译起始密码子周围加入NcoI位点,使YE2的第二个氨基酸从L改变为V。
按照方向将pZKUGPYE2(含有GPAT启动子和YE2编码区)的ClaI/NotI片段和含有Aco终止子的NotI/PacI片段(通过用引物YL325和YL326[SEQ ID NOs:371和372]对ACO 3’终止子进行PCR扩增,然后用NotI/PacI消化而制备)与ClaI/PacI消化的载体pZUF6S连接在一起,得到pZUF6YE2。随后将pZKUT16的ClaI/NcoI片段(含有TEF启动子)和pZUF6YE2的NcoI/PacI片段(含有YE2的编码区和Aco终止子)按照方向与ClaI/PacI消化的载体pZUF6S连接在一起,得到pZUF6TYE2(SEQ ID NO:149)。
分析超量表达YE2的转化的解脂耶氏酵母中的脂质组成
分别用质粒pZUF6S(图18A,SEQ ID NO:145)和pZUF6TYE2(SEQ ID NO:149)转化耶氏酵母菌株Y2031。这两种质粒的成分描述于表40和41。
表40
质粒pZUF6S(SEQ ID NO:145)的说明
| SEQ ID NO:145内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| EcoRI/ClaI(3114-4510) | 耶氏酵母自主复制序列18(ARS18;GenBank获取号M91600) |
| SalI/PacI(6022-4530) | 耶氏酵母Ura3基因(GenBank获取号AJ306421) |
| EcoRI/BsiWI(6063-318) | FBAIN::Δ6S::Pex20,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●Δ6S:密码子优化的Δ6去饱和酶基因(SEQ IDNO:3),来源于高山被孢霉(GenBank获取号AF465281)●Pex20:来自耶氏酵母Pex20基因(GenBank获取号AF054613)的Pex20终止子序列 |
表41
质粒pZUF6TYE2(SEQ ID NO:149)的说明
| SEQ ID NO:149内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| EcoRI/ClaI(7461-8857) | 耶氏酵母自主复制序列18(ARS18;GenBank获取号M91600) |
| SalI/PacI(1907-415) | 耶氏酵母Ura3基因(GenBank获取号AJ306421) |
| EcoRI/BsiWI(1948-4665) | FBAIN::Δ6S::Pex20:如对pZUF6的描述(上文) |
| ClaI/PacI(8857-415) | TEF::YE2::Aco,包含:●TEF:TEF启动子(GenBank获取号AF054508)●YE2:耶氏酵母YE2基因(SEQ ID NO:61;GenBank获取号CAG77901)的编码区●Aco:耶氏酵母Aco3基因(GenBank获取号AJ001301)的Aco3终止子序列 |
根据一般方法,用质粒pZUF6S(对照)和质粒pZUF6TYE2转化解脂耶氏酵母菌株2031(实施例5)。转化体在液体MM中生长2天。基于GC分析(如一般方法的描述),8个均含有pZUF6S或pZUF6YE2的菌落的脂肪酸谱示于下表42。脂肪酸鉴定为16:0(棕榈酸)、16:1(棕榈油酸)、18:0、18:1(油酸)、18:2(LA)和GLA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表42
用pZUF6S和pZUF6TYE2转化的耶氏酵母菌株Y2031中的脂肪酸组成的比较
GC分析显示在具有pZUF6S的Y2031转化体中生产了占总脂质的约27.1%的C16(C16:0和C16:1)和62.2%的C18(C18:0、C18:1、C18:2和GLA);在具有pZUF6TYE2的Y2031转化体中生产约21.3%的C16和73.6%的C18。因此,在pZUF6TYE2转化体中,C16的总量减少了约21.4%,C18的总量增加了约18%(与具有pZUF6S的转化体相比)。这些数据证明了TE2作为C16/18脂肪酸延伸酶起作用,在耶氏酵母中生产C18脂肪酸。此外,相对于在pZUF6S转化体中生产的GLA,在pZUF6TYE2转化体中生产多大约12.8%的GLA。这些数据表明YE2延伸酶能够促进碳流入工程化PUFA途径,生产更多的终产物(即GLA)。
实施例20
耶氏酵母C14/16脂肪酸延伸酶“YE1”增加PUFAs百分比
本实施例描述经过转化而共表达解脂耶氏酵母C14/16脂肪酸延伸酶(“YE1”;SEQ ID NO:65)的解脂耶氏酵母菌株Y2031(实施例5)中GLA生物合成和积累的增加。认为YE1延伸酶可以促使碳流入工程化的Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径,作为增加需要的PUFA即ARA的生产的手段。具体地,包含该C14/16脂肪酸延伸酶的嵌合基因可以容易地导入例如菌株Y2034、Y2047或Y2214中。
解脂耶氏酵母C14/16脂肪酸延伸酶的序列鉴定
采用大鼠Elo2 C16/18脂肪酸延伸酶蛋白序列(GenBank获取号AB071986;SEQ ID NO:51)作为询问序列,按照类似于实施例19的方法鉴定了来自解脂耶氏酵母的新的脂肪酸延伸酶候选物。这导致鉴定了标注为“未命名蛋白产物”的同源序列,即GenBank获取号CAG83378(SEQ ID NOs:64和65)。该基因命名为“YE1”。
用BLAST算法(Altschul,S.F.,et al.,Nucleic Acids Res.25:3389-3402(1997))比较耶氏酵母YE1氨基酸序列与公共数据库,发现最相似的已知序列是来自粗糙链孢霉的FEN1(GenBank获取号CAD70918;SEQ ID NO:66),它是一种与YE1具有约60%同一性的可能的脂肪酸延伸酶。
YE1基因的DNA序列(SEQ ID NO:64)具有内部NcoI位点。为了在YE1基因的“ATG”翻译起始密码子周围掺入耶氏酵母翻译基序,采用了两步策略对耶氏酵母的完整YE1基因进行PCR。具体地,采用耶氏酵母基因组DNA作为模板,采用寡核苷酸YL567和YL568(SEQ ID NOs:320和321)作为引物,通过PCR扩增YE1的前一半,而用寡核苷酸YL569和YL570(SEQ ID NOs:322和323)作为引物类似地扩增YE1基因的后一半。按照一般方法的描述,在50μl总体积中进行PCR反应。热循环仪条件设定为35个循环的95℃ 1min、56℃ 30sec、72℃ 1min,然后72℃下最终延伸10min。纯化相应于YE1的5’部分的PCR产物,然后用NcoI和SacI消化,得到YE1-1片段,同时纯化YE1的3’部分的PCR产物,用SacI和NotI消化,得到YE1-2片段。直接将YE1-1和YE1-2片段连接于NcoI/NotI消化的pZKUGPE1S(上文,实施例11),得到pZKUGPYE1(图19A,SEQ ID NO:150)。然后用pZKUGPYE1作为模板,寡核苷酸YL571和YL572(SEQ ID NOs:324和325)作为引物,通过定点诱变对YE1的内部NcoI位点进行突变,得到pZKUGPYE1-N(SEQ ID NO:147)。序列分析显示该突变没有改变YE1的氨基酸序列。在ATG翻译起始密码子周围加入NcoI位点,使YE1的第二个氨基酸从S改变为A。
将pZF5T-PPC的ClaI/NcoI片段(含有FBAIN启动子)和pZKUGPYE1-N的NcoI/PacI片段(含有YE1的编码区和Aco终止子)按照方向与ClaI/PacI消化的载体pZUF6S连接在一起,得到pZUF6FYE1(SEQ ID NO:151)。
分析超量表达YE1的转化的解脂耶氏酵母中的脂质组成
将包含解脂耶氏酵母YE1 ORF的嵌合基因克隆到质粒pZUF6中,使得可以通过转化的耶氏酵母菌株中脂肪酸组成的GC分析确定基因超量表达的作用。具体地,对照质粒pZUF6S(图18A;SEQ IDNO:145;包含FBAIN::D6S::Pex20嵌合基因)的成分描述于实施例19;而pZUF6FYE1(图19B;SEQ ID NO:151,包含FBAIN::D6S::Pex20嵌合基因和FBAIN::YE1::Aco嵌合基因)的成分描述于下表43。
表43
质粒pZUF6FYE1(SEQ ID NO:151)的说明
| SEQ ID NO:151内的RE位点和核苷酸 | 片段和嵌合基因成分的说明 |
| EcoRI/ClaI(7047-8445) | 耶氏酵母自主复制序列18(ARS18;GenBank获取号M91600) |
| SalI/PacI(1493-1) | 耶氏酵母Ura3基因(GenBank获取号AJ306421) |
| EcoRI/BsiWI(1534-4251) | FBAIN::Δ6S::Pex20:如对pZUF6的描述(上文实施例25) |
| ClaI/PacI(8443-1) | FBAIN::YE1::Aco,包含:●FBAIN:FBAIN启动子(SEQ ID NO:162)●YE1:耶氏酵母YE1基因(SEQ ID NO:64;GenBank获取号CAG83378)的编码区●Aco:来自耶氏酵母Aco3基因的(GenBank获取号AJ001301)Aco3终止子 |
转化后,使转化体在添加了氨基酸的合成MM中生长2天,然后在HGM中生长4天。基于GC分析(如一般方法中所述),下表55示出了6个含有pZUF6S的克隆和5个含有pZUF6FYE1的克隆的脂肪酸谱。脂肪酸鉴定为16:0(棕榈酸)、16:1(棕榈油酸)、18:0、18:1(油酸)、18:2(LA)和GLA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表44
用pZUF6S和pZUF6FYE1转化的耶氏酵母菌株Y2031中的脂肪酸组成的比较
GC分析测量出在具有pZUF6S的Y2031转化体中生产占总脂质的约31.1%的C16(C16:0+C16:1),而在具有pZUF6FYE1的Y2031转化体中生产约39.6%的C16。与具有pZUF6S的转化体相比,在pZUF6FYE1转化体中的C16总量增加约26.7%。因此,这些数据证明YE1作为C14/16脂肪酸延伸酶起作用,在耶氏酵母中生产C16脂肪酸。此外,在pZUF6FYE1转化体中生产的GLA比在pZUF6S转化体中多57%,表明YE1延伸酶能够促使碳流入工程化的途径,以生产更多的终产物(即GLA)。
实施例21
解脂耶氏酵母CPT1超量表达增加PUFAs的百分比
本实施例描述经过转化而超量表达解脂耶氏酵母CPT1 cDNA(SEQ ID NO:109)的解脂耶氏酵母菌株Y2067U(实施例10)中EPA生物合成和积累的增加。也增加了导致EPA合成的PUFAs。认为如果类似地共表达解脂耶氏酵母CPT1(如在菌株Y2034、Y2047或Y2214中),则经过工程化以通过Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径生产ARA的解脂耶氏酵母宿主菌株能够证明ARA生物合成和积累的增加。
用以下程序制备解脂耶氏酵母菌株ATCC #20326 cDNA。在30℃,使细胞在200mL YPD培养基(2%细菌用酵母提取物,3%细菌用肽胨,2%葡萄糖)中生长1天,然后通过在Beckman GH3.8转子中3750rpm离心10min而沉淀细胞,并用HGM洗涤两次。将洗涤的细胞重悬于200mL HGM中,30℃下再生长4hrs。然后通过在4x50mL试管中3750rpm离心10min,收获细胞。
用Qiagen Rneasy总RNA Midi试剂盒分离总RNA。为了破碎细胞,将收获的细胞重悬于4X600μl试剂盒缓冲液RLT(按照制造商的规定,补加了β-巯基乙醇)中,与等体积的0.5mm玻璃珠在4个2mL螺帽试管中混合。用Biospec微珠搅拌机在匀浆设置下将细胞破碎2min。加入额外的4 x 600μl缓冲液RLT。通过离心除去玻璃珠和细胞碎屑,根据制造商的方案用上清液分离总RNA。
按照制造商的方案,用Qiagen Oligotex mRNA纯化试剂盒从上面的总RNA样品分离聚A(+)RNA。用相同的试剂盒对分离的聚A(+)RNA再进行一轮纯化,以确保mRNA样品的纯度。最终的纯化的聚(A)+RNA的浓度为30.4ng/μl。
按照实施例13的描述用BD-Clontech规定的LD-PCR法和0.1μg聚A(+)RNA样品制备cDNA,不同之处是用于第一链cDNA合成的PCR热循环仪条件设定为95℃ 20sec,然后是20个循环的95℃ 5sec和68℃ 6min。通过琼脂糖凝胶电泳和溴化乙锭对PCR产物进行定量。
如下克隆解脂耶氏酵母CPT1 cDNA。用引物CPT1-5’-NcoI和CPT1-3’-NotI(SEQ ID NOs:326和327)通过PCR从解脂耶氏酵母的cDNA扩增解脂耶氏酵母ORF。反应混合物含有0.5μl cDNA、每种引物各0.5μl、11μl水和12.5μl ExTaq预混合2X Taq PCR溶液(TaKaRa Bio Inc.,Otsu,Shiga,520-2193,Japan)。按照如下方法进行扩增:最初在94℃变性300sec,然后是30个循环的94℃下变性30sec、55℃下退火30sec和72℃下延伸60sec。72℃进行最终的延伸循环10分钟,然后在4℃终止反应。从PCR反应获得了约1190bp DNA片段。根据制造商的方案,用Qiagen的PCR纯化试剂盒对其进行纯化。用NcoI和NotI消化纯化的PCR产物,克隆到Nco I-NotI切割的pZUF17载体(SEQ ID NO:118;图8B)中,使得基因处于解脂耶氏酵母FBAIN启动子和PEX20-3’终止子区控制下。通过微量制备物分析证实正确的转化体,得到的质粒命名为“pYCPT1-17”(SEQ ID NO:152)。
为了将嵌合FBAIN::CPT1::PEX20基因整合到解脂耶氏酵母的基因组中,通过用NcoI和NotI消化pYCPT1-17,并且分离含有CPT1ORF的约1190bp片段而建立质粒pYCPT1-ZP217。然后将该片段克隆到NcoI和NotI消化的pZP217+Ura(SEQ ID NO:153)中。如图19C所示,质粒pZP217+Ura是一种解脂耶氏酵母整合质粒,包含嵌合的TEF::合成Δ17去饱和酶(针对解脂耶氏酵母进行了密码子优化)::Pex20-3’基因和用作选择标记的Ura3基因。通过微量制备物分析证实正确的转化体,得到的质粒命名为“pYCPT1-ZP217”(SEQ IDNO:154)。
根据一般方法,分别用BssHII/BbuI消化的pYCPT1-ZP217和pZUF-MOD-1(上文实施例14)转化解脂耶氏酵母菌株Y2067U(来自实施例10)。使转化体在添加了氨基酸的合成MM中生长2天,然后在HGM中生长4天。基于GC分析(如一般方法中所述),下表示出了两个含有pZUF-MOD-1的转化体和基因组中整合了pYCPT1-ZP217的4个转化体的脂肪酸谱。脂肪酸鉴定为18:0、18:1(油酸)、18:2(LA)、GLA、DGLA、ARA、ETA和EPA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表45
经过工程化而超量表达解脂耶氏酵母CPT1的耶氏酵母菌株Y2067U中的脂质组成
如上所示,通过基因组整合,强FBAIN启动子控制下的解脂耶氏酵母CPT1的表达使EPA百分比从“对照”菌株中的13.4%增加到15.7-16%。此外,也增加了GLA、DGLA和ARA水平。
实施例22
啤酒糖酵母ISC1增加PUFAs百分比
本实施例描述经过转化而共表达啤酒糖酵母ISC1基因(SEQ IDNO:111)的解脂耶氏酵母菌株M4(实施例4)中EPA生物合成和积累的增加。认为如果类似地共表达啤酒糖酵母ISC1(如在菌株Y2034、Y2047或Y2214中),则经过工程化以通过Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径生产EPA的解脂耶氏酵母宿主菌株能够证明EPA生物合成和积累的增加。
如下将啤酒糖酵母ISC1ORF克隆到质粒pZP217+Ura中。首先,用来自啤酒糖酵母菌株S288C(Promega,Madison,WI)的基因组DNA以及引物对Isc1F和Isc1R(SEQ ID NOs:328和329)PCR扩增ORF。引物Isc1F将扩增的ORF中的ISC1的野生型5’序列从‘ATGTACAA’修饰为‘ATGGACAA’,因为必需掺入NcoI位点从而使ISC1符合读码框。按照如下方法进行扩增:最初在94℃变性120sec,然后是35个循环的94℃下变性30sec、50℃下退火30sec和68℃下延伸120sec。68℃进行最终的延伸循环10分钟,然后在4℃终止反应。从ISC1的PCR反应获得了1455bp DNA片段,用1%琼脂糖凝胶(120V,30min)和来自Invitrogen(Carlsbad,CA)的1kB DNA标准物通过电泳证实了PCR产物大小。
根据制造商的说明,用来自Zymo Research Corporation(Orange,CA)的DNA Clean & Concentrator-5试剂盒纯化DNA。然后将ISC1片段分别克隆到NcoI和NotI消化的pZP217+Ura(SEQ IDNO:153;图19C)中。通过凝胶电泳证实正确的转化体,得到的质粒命名为“pTEF::ISC1”(SEQ ID NO:155)。因此,该质粒包含DNA盒,该DNA盒包含3’-POX2、URA3、TEF::ISC1::Pex20和POX2启动子区。
如下制备“对照”载体。首先,用来自啤酒糖酵母菌株S288C的基因组DNA和引物对Pcl1F和Pcl1R(SEQ ID NOs:330和331)PCR扩增啤酒糖酵母pcl1 ORF(编码参与进入有丝分裂细胞周期和调节形态发生的蛋白)。按照上文的描述进行扩增。从pcl1的PCR反应获得861bp DNA片段(通过上文描述的电泳证实)。用DNA Clean &Concentrator-5试剂盒纯化DNA,然后用NcoI/NotI消化。然后将片段克隆到相似消化的pZP217+Ura中。通过凝胶电泳证实正确的转化体,得到的质粒命名为“pTEF::pcl1”。然后用HincII消化质粒pTEF::plc1,除去pcl1ORF。重新连接得到的质粒,使得用AscI/SphI消化后得到包含3’-POX2、URA3、TEF::Pex20和POX2启动子区的线性DNA盒。
分别用Asc1/Sph1消化的pTEF::ISC1和“对照”转化感受态解脂耶氏酵母菌株M4细胞(来自实施例4)(其中每种质粒各5μg进行了消化)。用Frozen EZ Yeast Transformation II试剂盒(ZymoResearch)完成转化,在包含不含氨基酸的YNB(6.7g/L;Becton,Dickinson and Co.,Sparks,MD[目录号291940])、葡萄糖(20g/L)和琼脂(20g/L)的平板上选择转化体。获得了几百个转化的菌落。采用来自ISC1的5个独立转化体的基因组DNA,通过PCR证实了每个DNA盒整合到解脂耶氏酵母POX2基因座中。
使转化体在不含氨基酸、含有2%葡萄糖的YNB中生长。通过离心收集细胞,重悬于包含100g/L右旋糖、2g/L MgSO4和50mM pH 6.5的磷酸缓冲液的培养基中,再生长5天。通过离心收集来自0.75mL每种培养物的细胞,分析它们的脂肪酸组成。基于GC分析(如一般方法中所述),下文示出了3个含有“对照”载体的转化体和5个含有pTEF::ISC1的转化体的脂肪酸谱。脂肪酸鉴定为16:0、16:1、18:0、18:1(油酸)、18:2(LA)、GLA、DGLA、ARA、ETA和EPA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表46
经过工程化而超量表达啤酒糖酵母ISC1的耶氏酵母菌株M4中的脂质组成
啤酒糖酵母ISC1基因的表达使EPA的百分比从“对照”菌株中的9.3%增加到10.7%(“M4+pTEF::ISC1”),代表增加了14.5%。
实施例23
制备解脂耶氏酵母酰基转移酶敲除体
本实施例描述了建立解脂耶氏酵母的单、双和三敲除菌株,所述菌株中破坏了PDAT、DGAT2、DGAT1、PDAT和DGAT2、PDAT和DGAT1、DGAT1和DGAT2,或PDAT、DGAT1和DGAT2基因。证实了每个敲除菌株中基因的破坏,通过实施例24中的总脂质的GC分析确定了每个破坏对脂肪酸含量和组成的影响。
靶向破坏解脂耶氏酵母DGAT2基因
通过用称作质粒pY21DGAT2的靶向盒对内源DGAT2基因的同源重组-介导的替换,进行解脂耶氏酵母ATCC #90812中的DGAT2基因的靶向破坏。pY21DGAT2源自质粒pY20(图19D;SEQ ID NO:156)。具体地,通过将570bp Hind III/Eco RI片段插入类似地线性化的pY20,制备pY21DGAT2。570bp DNA片段含有(按5’至3’方向):3’同源序列位置+1090至+1464(SEQ ID NO:89的编码序列(ORF)),Bgl II限制位点和5’同源序列位置+906至+1089(SEQ IDNO:89所示的编码序列(ORF))。分别使用两对PCR引物P95和P96(SEQ ID NO:332和333)和P97和P98(SEQ ID NO:334和335),通过PCR扩增制备片段。
通过Bgl II限制消化,将pY21DGAT2线性化,并转化进对数中期解脂耶氏酵母ATCC #90812细胞,如一般方法中所述。将细胞铺板到YPD潮霉素选择平板,并在30℃维持2-3天。
分离14个解脂耶氏酵母ATCC #90812潮霉素-抗性菌落,通过PCR筛选靶向破坏。一组PCR引物(P115和P116[分别是SEQ IDNO:336和337])用于在同源重组后扩增特异性的连接片段。另一对PCR引物(P115和P112[SEQ ID NO:338])用于检测天然基因。
ATCC #90812菌株的14个潮霉素-抗性菌落中的2个是对连接片段阳性的,且对天然片段阴性的。因而,在这2个菌株中证实了靶向整合,其中之一命名为“S-D2”。
靶向破坏解脂耶氏酵母PDAT基因
通过用称作pLV13(图19E;SEQ ID NO:157)的靶向盒对内源PDAT基因的同源重组-介导的替换,进行解脂耶氏酵母ATCC#90812中的PDAT基因的靶向破坏。pLV13源自质粒pY20(图19D;SEQ ID NO:156)。具体地,通过将992bp Bam HI/Eco RI片段插入类似地线性化的pLV5,制备pLV13。992bp DNA片段含有(按5’至3’方向):3’同源序列位置+877至+1371(SEQ ID NO:76的编码序列(ORF)),Bgl II限制位点和5’同源序列位置+390至+876(SEQID NO:76的编码序列(ORF))。分别使用PCR引物P39和P41(SEQ ID NO:339和340)和P40和P42(SEQ ID NO:341和342),通过PCR扩增制备片段。
通过Bgl II限制消化,将pLV13线性化,并根据一般方法转化进对数中期解脂耶氏酵母ATCC #90812细胞。将细胞铺板到Bio101DOB/CSM-Ura选择平板,并在30℃维持2-3天。
分离10个解脂耶氏酵母ATCC #90812菌落,通过PCR筛选靶向破坏。一组PCR引物(P51和P52[分别是SEQ ID NO:343和344])设计用于扩增靶向盒。另一组PCR引物(P37和P38[分别是SEQ IDNO:345和346])设计用于检测天然基因。10个菌株中的10个都是对连接片段阳性的,且10个菌株中的3个是对天然片段阴性的,因而,在这3个菌株中证实了成功的靶向整合。这些菌株之一命名为“S-P”。
解脂耶氏酵母DGAT1基因的靶向破坏
使用简并PCR引物P201和P203(分别是SEQ ID NO:347和348)和解脂耶氏酵母ATCC #76982基因组DNA作为模板,通过PCR克隆全长Yl DGAT1 ORF。需要简并引物,因为不知道编码Yl DGAT1的核苷酸序列。
在RoboCycler Gradient 40 PCR仪中进行PCR,按照如下进行扩增:95℃下初步变性1min,然后是30个循环的95℃下变性30sec、55℃下退火1min和72℃下延伸1min。72℃进行最终的延伸循环10分钟,然后在4℃终止反应。通过琼脂糖凝胶电泳检测预期的PCR产物(约1.6kB),分离,纯化,克隆进克隆载体(Invitrogen),并部分地测序,以证实它的身份。
通过靶向盒对内源DGAT1基因的同源重组-介导的替换,进行解脂耶氏酵母ATCC #90812中的推定DGAT1基因的靶向破坏(采用上文对DGAT2描述的方法)。具体地,将1.6kB分离的Yl DGAT1 ORF(SEQ ID NO:81)用作PCR模板分子,构建由下述组分组成的YlDGAT1靶向盒:5’同源Yl DGAT1序列(用引物P214和P215(SEQID NOs:349和350)扩增)、耶氏酵母亮氨酸2(Leu2;GenBank获取号AAA35244)基因,和3’同源Yl DGAT1序列(用引物P216和P217(SEQ ID NOs:351和352)扩增)。用Pfu Ultra聚合酶(Stratagene,目录号600630)和上文所述热循环仪条件扩增靶向盒的每个单独部分后,纯化每个片段。将3个正确大小的、纯化的片段混合到一起,作为使用PCR引物P214和P219(SEQ ID NO:353)的第二个PCR反应的模板分子,得到Yl DGAT1破坏盒。
凝胶纯化靶向盒,并用于转化对数中期野生型解脂耶氏酵母(ATCC #90812)。如一般方法中所述进行转化。将转化体铺板到Bio101 DOB/CSM-Leu选择平板上,并在30℃维持2-3天。通过PCR筛选几种亮氨酸原养型,以证实靶向的DGAT1破坏。具体地,一组PCR引物(P226和P227[SEQ ID NO:354和355])用于扩增破坏盒和天然靶基因之间的连接。另一组PCR引物(P214和P217[SEQ IDNO:349和352])用于检测天然基因。
所有亮氨酸原养型菌落都是对连接片段阳性的,且是对天然片段阴性的,因而,在这些菌株中证实了靶向整合,其中之一命名为“S-D1”。
建立含有PDAT和/或DGAT2和/或DGAT1基因破坏的解脂耶氏酵母双敲除和三敲除菌株
用质粒pLV13(含有PDAT破坏)转化解脂耶氏酵母ATCC #90812潮霉素抗性“S-D2”突变体(含有DGAT2破坏),按照对单个PDAT破坏的描述,通过PCR筛选转化体。证明12个转化体中的两个在DGAT2和PDAT基因中都有破坏。这些菌株之一命名为“S-D2-P”。
类似地,制备了在DGAT1和PDAT(“S-D1-P”)中、在DGAT2和DGAT1(“S-D2-D1”)中具有双敲除、以及在DGAT2、DGAT1和PDAT中具有三敲除的菌株(“S-D2-D1-P”)。
实施例24
解脂耶氏酵母酰基转移酶敲除减少脂质含量并增加PUFAs百分比
本实施例分析了酰基转移酶的单和/或双和/或三敲除在野生型解脂耶氏酵母和以前经过工程化而生产EPA的解脂耶氏酵母菌株中的影响,这是通过脂肪酸含量和组成而测量的。认为如果类似地操作宿主的天然酰基转移酶(如在菌株Y2034、Y2047或Y2214中),则经过工程化以通过Δ6去饱和酶/Δ6延伸酶途径或Δ9延伸酶/Δ8去饱和酶途径生产ARA的解脂耶氏酵母宿主菌株能够证明ARA生物合成和积累的增加。
具有酰基转移酶破坏的解脂耶氏酵母ATCC #90812中的TAG含量减少
首先,比较了野生型和突变的解脂耶氏酵母ATCC #90812中的TAG含量,所述突变的解脂耶氏酵母ATCC #90812含有:(1)PDAT、DGAT2和DGAT1中的单个破坏;(2)PDAT和DGAT2、DGAT1和PDAT,以及DGAT1和DGAT2中的双破坏;和(3)PDAT、DGAT2和DGAT1的三破坏。
具体地,分别将来自含有野生型和突变的解脂耶氏酵母ATCC#90812(即S-D1、S-D2、S-P、S-D1-D2、S-D1-P、S-D2-P和S-D1-D2-P)的一环细胞接种在3mL YPD培养基中,30℃下在摇床(300rpm)上生长过夜。收获细胞,在0.9% NaCl中洗涤一次,重悬于50mLHGM中。然后使细胞在摇床上生长48小时。在水中洗涤细胞,冻干细胞沉淀。将二十(20)mg干细胞量用于通过GC分析总脂肪酸和TLC(下文)和GC分析后的油级分。
在下面的5个步骤中,描述了TLC使用的方法:(1)将内部标准品15:0脂肪酸(10μl 10mg/mL)加入2-3mg干细胞团,然后使用甲醇/氯仿方法提取总脂质。(2)使用25-50μl微量吸管,将提取的脂(50μl)印迹到绘在离5 x 20cm硅胶60平板底部约1英寸的光束线上。(3)然后在N2下干燥TLC平板,并插入含有约100mL 80:20:1己烷:乙醚:醋酸溶剂的罐中。(4)分离带后,将碘蒸汽吹过平板的一侧,以鉴别带。这使得能够使用剃刀刀片刮掉在平板的另一侧上的样品,用于进一步分析。(5)如一般方法中所述,进行刮掉的样品的基础酯交换和GC分析。
GC结果示于下表47。将培养物描述为“S”菌株(野生型),“S-P”(PDAT敲除),“S-D1”(DGAT1敲除),“S-D2”(DGAT2敲除),“S-D1-D2”(DGAT1和DGAT2敲除),“S-P-D1”(PDAT和DGAT1敲除),“S-P-D2”(PDAT和DGAT2敲除)和“S-P-D1-D2”(PDAT,DGAT1和DGAT2敲除)。使用的缩写是:“WT”=野生型;“FAs”=脂肪酸;“dcw”=干细胞重量;并且,“FAs% dcw,% WT”=相对于野生型中的%的FAs% dcw,其中“S”菌株是野生型。
表47
在PDAT、DGAT2和DGAT1中具有单个、两个或三个破坏的耶氏酵母ATCC #90812菌株的脂含量
表47的结果表明3种DAG AT对油生物合成的相对贡献。DGAT2的贡献最大,而PDAT和DGAT1的贡献相等,但是小于DGAT2。三敲除菌株脂的约3%残留油含量可能是ARE2(SEQ ID NOs:78和79)编码的解脂耶氏酵母的酰基-CoA:甾醇-酰基转移酶的贡献。
具有破坏的DGAT2基因的菌株EU中的TAG含量减少,EPA百分比增加
检验野生型解脂耶氏酵母ATCC #90812中的多种酰基转移酶敲除(上文)的影响后,在EU菌株(即,经过工程化而生产10% EPA;参见实施例10)的DGAT2敲除菌株中检验TAG含量和脂肪酸组成。
具体地,按照实施例23中对S菌株(ATCC #90812)的描述,破坏菌株EU中的DGAT2基因。DGAT2破坏的菌株命名为EU-D2。收获EU和EU-D2菌株,在根据两种不同条件生长后进行分析。在下表中称作“3mL”的条件下,使细胞在3mL MM培养基中生长1天,洗涤,然后在3mL HGM中生长3天。或者,在下表中称作“51mL”的条件下,使细胞在51mL MM培养基中生长1天,洗涤,然后在51mL HGM中生长3天。在TLC分离后的51mL培养物的提取物(“级分”)中确定磷脂酰胆碱(PC)、磷脂酰乙醇胺(PE)和三酰基甘油(TAG或油)的脂肪酸组成。
GC结果示于下表48。培养物描述为“EU”菌株(野生型)和“EU-D2”菌株(DGAT2敲除)。脂肪酸鉴定为16:0、16:1、18:0、18:1(油酸)、18:2(LA)、GLA、DGLA、ARA、ETA和EPA;每种脂肪酸的组成表示为总脂肪酸的百分比。
表48
具有DGAT2破坏的耶氏酵母菌株EU的脂质含量和组成
结果表明DGAT2敲除导致% EPA(占总脂肪酸的百分比)的加倍和脂质含量的减半(表示为% dcw)。此外,脂含量中观察到的几乎所有改变都是由于TAG级分的改变。菌株EU的51mL培养物中的% EPA比预期的少,可能是由于不稳定性。
具有破坏的酰基转移酶基因的解脂耶氏酵母菌株中MU中TAG含量增加,EPA百分比减少
最后,基于菌株EU-D2中单个DGAT2敲除导致的EPA百分比增加和脂质含量减少,在菌株MU(经过工程化而生产14% EPA,参见实施例12)的多个酰基转移酶敲除菌株中研究了TAG含量和脂肪酸组成。具体地,在菌株MU中,建立了在PDAT、DGAT2和DGAT1中的单个破坏,和在PDAT和DGAT2中的双破坏。在4种不同生长条件下生长后,比较每个这样的菌株的脂含量和组成。
更具体地,使用实施例23的方法(不同之处是,DGAT1破坏的选择依赖于URA3基因),在菌株MU中建立PDAT、DGAT2、DGAT1中的单个破坏。这会产生单敲除菌株,分别称作“MU-D1”(破坏DGAT1)、“MU-D2”(破坏DGAT2)和“MU-P”(破坏PDAT)。通过PCR证实各个敲除菌株。另外,通过相同方法破坏MU-D2菌株的PDAT基因,并通过PCR证实破坏。得到的双敲除菌株称作“MU-D2-P”。
分析MU-D1、MU-D2、MU-P和M-D2-P敲除菌株,以确定每个敲除对脂含量和组成的影响,如下所述。而且,还使用促进含油性的生长条件,来确定它们对总脂含量的影响。因而,共进行了4个不同的实验,分别称作“实验A”、“实验B”、“实验C”和“实验E”。更具体地,将3环来自含有上述每种菌株的平板的细胞接种进MMU[实验B和C是3mL;实验A和E是50mL],并在30℃摇床中生长24小时(实验A,B和C)或48小时(实验E)。收获细胞,在HGM中洗涤一次,重新悬浮于HGM(实验A和E是50mL;且实验B是3mL)或含有尿嘧啶的HGM(“HGMU”)(实验C是3mL),并如上培养4天。将一份等分物(1mL)用于根据一般方法所述的GC脂分析,第二份等分物用于测定培养物在600nm的OD。收获实验A和E的剩余培养物,在水中洗涤一次,冻干,用于干细胞重量(dcw)测定。相反地,使用显示它们的关系的方程,从它们的OD600,确定实验B和C的dcw。还测定了实验A,B,C和E中的不同菌株各自的脂肪酸组成。
在下面的表49中显示了结果。将培养物描述为“MU”菌株(亲本EPA生产菌株),“MU-P”(PDAT敲除),“MU-D1”(DGAT1敲除),“MU-D2”(DGAT2敲除)和“MU-D2-P”(DGAT2和PDAT敲除)。使用的缩写是:“WT”=野生型(即,MU);“OD”=光密度;“dcw”=干细胞重量;“TFAs”=总脂肪酸;和,“TFAs% dcw,% WT”=相对于野生型(“MU”)菌株的TFAs% dcw。脂肪酸鉴定为16:0、16:1、18:0、18:1(油酸)、18:2(LA)、GLA、DGLA、ARA、ETA和EPA;每种脂肪酸的组成表示为总脂肪酸的百分比。
数据证实,转化细胞中的脂质含量随生长条件而变化。而且,每种酰基转移酶对脂质含量的贡献也变化。具体地,在实验B、C和E中,DGAT2对油生物合成的贡献大于PDAT或DGAT1。相反地,如实验A所证实的,在DGAT2、DGAT1和PDAT中的单敲除会导致大约相同的脂质含量减少(即,分别是减少48%、49%和42%[参见“TFAs% dcw,% WT”])。
至于脂肪酸组成,数据表明每个DAG AT基因的敲除导致油含量降低和EPA百分比增加。例如,DGAT2敲除导致大约一半的脂质含量以及EPA占脂肪酸的百分比大约加倍(类似于上文菌株EU-D2中观察到的结果)。DAGAT2和PDAT都敲除,导致最少的油和最多的EPA百分比。
基于此处报道的结果,认为天然DGAT2和/或DGAT1和/或PDAT的破坏是显著降低经过工程化而生产高浓度的PUFAs,包括ARA的解脂耶氏酵母菌株(如在菌株Y2034、Y2047或Y2214中)的PUFAs百分比的有用手段。实际上,菌株Y2214中天然DGAT2的破坏导致ARA的百分比增加1.7倍(数据未示出)。
Claims (36)
1.用于生产花生四烯酸的重组生产宿主细胞,包括背景耶氏酵母种,其中包含基因集合,所述集合包含ω-3/ω-6脂肪酸生物合成途径的以下基因:
a)至少一种编码Δ6去饱和酶的基因;
b)至少一种编码C18/20延伸酶的基因;和
c)至少一种编码Δ5去饱和酶的基因。
2.用于生产花生四烯酸的重组生产宿主细胞,包括背景耶氏酵母种,其中包含基因集合,所述集合包含ω-3/ω-6脂肪酸生物合成途径的以下基因:
a)至少一种编码Δ9延伸酶的基因;
b)至少一种编码Δ8去饱和酶的基因;和
c)至少一种编码Δ5去饱和酶的基因。
3.权利要求1或2的重组生产宿主,其中该基因集合任选包含至少一种编码Δ12去饱和酶的基因。
4.权利要求3的重组生产宿主,其中该背景耶氏酵母种不含任何编码具有Δ12去饱和酶活性的多肽的天然基因。
5.权利要求1或2的重组生产宿主,其中所述ω-3/ω-6脂肪酸生物合成途径基因的至少一种基因处于具有选自SEQ ID NOs:158-168和364的核酸序列的启动子序列的控制下。
6.权利要求3的重组生产宿主,其中所述Δ12去饱和酶具有选自SEQ ID NOs:24、26、28、30、31、32、34、36、38、358、359、361、362和363的氨基酸序列。
7.权利要求1的重组生产宿主细胞,其中所述Δ6去饱和酶具有选自SEQ ID NOs:2和5的氨基酸序列;并且其中所述C18/20延伸酶具有选自SEQ ID NOs:18和21的氨基酸序列。
8.权利要求2的重组生产宿主细胞,其中所述Δ9延伸酶具有选自SEQ ID NOs:40和18的氨基酸序列,并且其中所述Δ8去饱和酶具有选自SEQ ID NOs:45、47和49的氨基酸序列。
9.权利要求1或2的重组生产宿主细胞,其中所述Δ5去饱和酶具有选自SEQ ID NOs:7、9和12的氨基酸序列。
10.权利要求1或2的重组生产宿主,其中该基因集合任选包含选自下组的ω-3/ω-6脂肪酸生物合成途径基因:
a)至少一种编码Δ9去饱和酶的基因;
b)至少一种编码C16/18延伸酶的基因;和
c)至少一种编码C14/16延伸酶的基因。
11.权利要求10的重组生产宿主,其中所述C16/18延伸酶具有选自SEQ ID NOs:51、54和62的氨基酸序列,并且其中所述C14/16延伸酶具有SEQ ID NO:65所示的氨基酸序列。
12.权利要求1或2的重组生产宿主,其中该基因集合任选包含至少一种编码选自下组的酰基转移酶的基因:
(1)二酰基甘油酰基转移酶(DGAT1);
(2)二酰基甘油酰基转移酶(DGAT2);
(3)磷脂:二酰基甘油酰基转移酶(PDAT);
(4)酰基-CoA:1-酰基溶血磷脂酰胆碱酰基转移酶(LPCAT);
(5)甘油-3-磷酸酰基转移酶(GPAT);和
(6)溶血磷脂酸酰基转移酶(LPAAT)。
13.权利要求12的重组生产宿主,其中所述二酰基甘油酰基转移酶(DGAT1)具有选自SEQ ID NOs:82和84-88的氨基酸序列;其中所述二酰基甘油酰基转移酶(DGAT2)具有选自SEQ ID NOs:90、92、94和96的氨基酸序列;其中所述磷脂:二酰基甘油酰基转移酶(PDAT)具有SEQ ID NO:77所示的氨基酸序列;其中所述甘油-3-磷酸酰基转移酶(GPAT)具有SEQ ID NO:98所示的氨基酸序列;其中所述溶血磷脂酸酰基转移酶(LPAAT)具有选自SEQ ID NOs:68、70、72和75的氨基酸序列;并且其中所述酰基-CoA:1-酰基溶血磷脂酰胆碱酰基转移酶(LPCAT)具有SEQ ID NO:80所示的氨基酸序列。
14.用于生产花生四烯酸的重组生产宿主细胞,包括背景耶氏酵母种,其中包含基因集合,所述集合包含ω-3/ω-6脂肪酸生物合成途径的以下基因:
a)至少一种编码Δ6去饱和酶的基因;
b)至少一种编码C18/20延伸酶的基因;
c)至少一种编码Δ5去饱和酶的基因;
d)至少一种编码Δ17去饱和酶的基因;和
e)至少一种编码Δ12去饱和酶的基因;并且
其中所述背景耶氏酵母种不含任何编码苹果酸异丙酯脱氢酶(Leu2-)的天然基因。
15.用于生产花生四烯酸的重组生产宿主细胞,包括背景耶氏酵母种,其中包含基因集合,所述集合包含ω-3/ω-6脂肪酸生物合成途径的以下基因:
f)至少一种编码Δ9延伸酶的基因;
g)至少一种编码Δ8去饱和酶的基因;
h)至少一种编码Δ5去饱和酶的基因;
i)至少一种编码Δ12去饱和酶的基因;和
j)至少一种编码C16/18延伸酶的基因;并且
其中所述背景耶氏酵母种不含任何编码酵母氨酸脱氢酶(Lys5-)的天然基因。
16.用于生产花生四烯酸的重组生产宿主细胞,包括背景耶氏酵母种,其中包含基因集合,所述集合包含ω-3/ω-6脂肪酸生物合成途径的以下基因:
a)至少一组基因,选自:
(1)至少一种编码Δ6去饱和酶的基因;至少一种编码C18/20延伸酶的基因;和至少一种编码Δ5去饱和酶的基因;
(2)至少一种编码Δ9延伸酶的基因;至少一种编码Δ8去饱和酶的基因;和至少一种编码Δ5去饱和酶的基因;
b)至少一种编码选自下组的酶的基因:
(1)Δ12去饱和酶;
(2)Δ9去饱和酶;
(3)C14/16延伸酶;
(4)C16/18延伸酶;和
c)至少一种编码选自下组的酶的基因:
(i)选自DGAT1、DGAT2和PDAT的二酰基甘油酰基转移酶;
(ii)酰基-CoA:1-酰基溶血磷脂酰胆碱酰基转移酶(LPCAT);
(iii)甘油-3-磷酸酰基转移酶(GPAT);
(iv)溶血磷脂酸酰基转移酶(LPAAT);
(v)磷脂酶C;和
(vi)磷脂酶A2;
其中:
(1)该背景耶氏酵母种不含任何编码具有Δ12去饱和酶活性的多肽的天然基因,
(2)该背景耶氏酵母种不含任何编码选自脂肪酶1(Lip1-)、过氧化物酶体酰基CoA氧化酶ACO3(Pox3-)、酰基-CoA氧化酶2(Pox2-)、乳清苷-5’-磷酸脱羧酶(Ura3-)、酵母氨酸脱氢酶(Lys5-)、脂肪酶2(Lip2-)和苹果酸异丙酯脱氢酶(Leu2-)的酶的天然基因。
17.权利要求1或2的重组生产宿主,其中该宿主生产的微生物油包含占总脂肪酸的至少约5%的花生四烯酸。
18.权利要求1或2的重组生产宿主,其中该宿主生产包含花生四烯酸的微生物油,并且该微生物油不含任何γ-亚油酸。
19.生产包含花生四烯酸的微生物油的方法,包括:
a)培养权利要求1或2的宿主,所述宿主中生产包含花生四烯酸的微生物油;和
b)任选回收步骤(a)的微生物油。
20.由权利要求19的方法生产的微生物油。
21.权利要求20的微生物油,其中所述油含有至少约5%的花生四烯酸。
22.权利要求20的混合油,其中所述油包含选自亚油酸、γ-亚麻酸、二十碳二烯酸、二高-γ-亚油酸、花生四烯酸、α-亚麻酸、十八碳四烯酸、二十碳三烯酸、二十碳四烯酸、二十二碳五烯酸和二十二碳六烯酸的脂肪酸。
23.包含有效量的由权利要求19的方法生产的微生物油的食品。
24.权利要求23的食品,选自食物类似物、肉类产品、谷物产品、烘烤食品、快餐和乳制品。
25.选自医学食品、饮食补充物、婴儿配方和药品的产品,包含有效量的由权利要求19的方法生产的微生物油。
26.包含有效量的由权利要求19的方法生产的微生物油的动物饲料。
27.权利要求26的动物饲料,其中所述动物饲料选自宠物饲料、反刍动物饲料、禽类动物饲料和水产养殖饲料。
28.包含有效量的微生物油并且任选包含酵母生物物质的动物饲料,包含权利要求1或2的重组宿主。
29.权利要求28的动物饲料,其中该酵母生物物质包括选自蛋白、脂质、碳水化合物、维生素、矿物质和核酸的饲料营养物质。
30.制备补加了花生四烯酸的食品的方法,包括将权利要求19的方法生产的微生物油与食品组合。
31.制备选自医学食品、饮食补充物、婴儿配方和药品的产品的方法,其中该产品补充了花生四烯酸,该方法包括将权利要求19的方法生产的微生物油与该产品组合。
32.制备补充了花生四烯酸的动物饲料的方法,包括将权利要求19的方法生产的微生物油与动物饲料组合。
33.给包含花生四烯酸的动物饲料补充饲料营养物质的方法,包括将权利要求26的动物饲料与包含饲料营养物质的酵母生物物质组合。
34.权利要求33的方法,其中该饲料营养物质选自蛋白、脂质、碳水化合物、维生素、矿物质和核酸。
35.提供富含花生四烯酸的人、动物或水产养殖生物饮食补充物的方法,包括提供权利要求19的方法生产的含有花生四烯酸的微生物油,该微生物油是可被人或动物消耗或使用的形式。
36.用于生产花生四烯酸的重组生产宿主解脂耶氏酵母Y2047,ATCC保藏号为ATCC______。
Applications Claiming Priority (2)
| Application Number | Priority Date | Filing Date | Title |
|---|---|---|---|
| US62481204P | 2004-11-04 | 2004-11-04 | |
| US60/624,812 | 2004-11-04 |
Publications (1)
| Publication Number | Publication Date |
|---|---|
| CN101437952A true CN101437952A (zh) | 2009-05-20 |
Family
ID=36337096
Family Applications (4)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CNA2005800457798A Pending CN101437951A (zh) | 2004-11-04 | 2005-11-03 | 解脂耶氏酵母的二十二碳六烯酸生产菌株 |
| CN200580045823.5A Expired - Fee Related CN101111601B (zh) | 2004-11-04 | 2005-11-03 | 解脂耶氏酵母的高二十碳五烯酸生产菌株 |
| CN2005800459172A Expired - Fee Related CN101437953B (zh) | 2004-11-04 | 2005-11-03 | 用于改变含油生物的多不饱和脂肪酸和油含量的二酰基甘油酰基转移酶 |
| CNA2005800459100A Pending CN101437952A (zh) | 2004-11-04 | 2005-11-03 | 解脂耶氏酵母的高花生四烯酸酸生产菌株 |
Family Applications Before (3)
| Application Number | Title | Priority Date | Filing Date |
|---|---|---|---|
| CNA2005800457798A Pending CN101437951A (zh) | 2004-11-04 | 2005-11-03 | 解脂耶氏酵母的二十二碳六烯酸生产菌株 |
| CN200580045823.5A Expired - Fee Related CN101111601B (zh) | 2004-11-04 | 2005-11-03 | 解脂耶氏酵母的高二十碳五烯酸生产菌株 |
| CN2005800459172A Expired - Fee Related CN101437953B (zh) | 2004-11-04 | 2005-11-03 | 用于改变含油生物的多不饱和脂肪酸和油含量的二酰基甘油酰基转移酶 |
Country Status (9)
| Country | Link |
|---|---|
| US (8) | US7588931B2 (zh) |
| EP (5) | EP2458000B1 (zh) |
| JP (3) | JP5080979B2 (zh) |
| KR (3) | KR20070085669A (zh) |
| CN (4) | CN101437951A (zh) |
| CA (3) | CA2584719C (zh) |
| DK (4) | DK1807527T3 (zh) |
| NO (3) | NO20072517L (zh) |
| WO (3) | WO2006052871A2 (zh) |
Cited By (3)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN104099254A (zh) * | 2014-07-11 | 2014-10-15 | 无锡超科食品有限公司 | 一株产多不饱和脂肪酸的菌株及其筛选方法 |
| CN110499259A (zh) * | 2019-07-22 | 2019-11-26 | 浙江工业大学 | 一种解酯耶氏酵母yw100-1及其应用 |
| CN111685223A (zh) * | 2019-03-13 | 2020-09-22 | 上海吉态来生物技术有限公司 | 含有解脂亚罗酵母的饲料、其制备方法及其应用 |
Families Citing this family (165)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US8313911B2 (en) * | 2003-05-07 | 2012-11-20 | E I Du Pont De Nemours And Company | Production of polyunsaturated fatty acids in oleaginous yeasts |
| US11952581B2 (en) | 2003-08-01 | 2024-04-09 | Basf Plant Science Gmbh | Process for the production of polyunsaturated fatty acids in transgenic organisms |
| EP2172536B1 (de) | 2003-08-01 | 2016-05-04 | BASF Plant Science GmbH | Verfahren zur Herstellung mehrfach ungesättigter Fettsäuren in transgenen Organismen |
| CN1930277B (zh) | 2004-02-27 | 2011-02-02 | 巴斯福植物科学有限公司 | 在转基因植物中产生多不饱和脂肪酸的方法 |
| BRPI0510132A (pt) | 2004-04-22 | 2007-10-02 | Commw Scient Ind Res Org | sìntese de ácidos graxos poliinsaturados de cadeia longa por células recombinantes |
| CN103451246B (zh) | 2004-04-22 | 2018-02-16 | 联邦科学技术研究组织 | 用重组细胞合成长链多不饱和脂肪酸 |
| CA2568689A1 (en) * | 2004-06-04 | 2005-12-15 | Fluxome Sciences A/S | Metabolically engineered cells for the production of polyunsaturated fatty acids |
| US20060094102A1 (en) * | 2004-11-04 | 2006-05-04 | Zhixiong Xue | Ammonium transporter promoter for gene expression in oleaginous yeast |
| US7879591B2 (en) | 2004-11-04 | 2011-02-01 | E.I. Du Pont De Nemours And Company | High eicosapentaenoic acid producing strains of Yarrowia lipolytica |
| US7588931B2 (en) | 2004-11-04 | 2009-09-15 | E. I. Du Pont De Nemours And Company | High arachidonic acid producing strains of Yarrowia lipolytica |
| GB0503657D0 (en) | 2005-02-22 | 2005-03-30 | Fluxome Sciences As | Metabolically engineered cells for the production of resveratrol or an oligomeric or glycosidically-bound derivative thereof |
| US7470532B2 (en) * | 2005-10-19 | 2008-12-30 | E.I. Du Pont De Nemours And Company | Mortierella alpina C16/18 fatty acid elongase |
| US20070118929A1 (en) | 2005-11-23 | 2007-05-24 | Damude Howard G | Delta-9 elongases and their use in making polyunsaturated fatty acids |
| AR059376A1 (es) * | 2006-02-21 | 2008-03-26 | Basf Plant Science Gmbh | Procedimiento para la produccion de acidos grasos poliinsaturados |
| US7465793B2 (en) | 2006-04-20 | 2008-12-16 | E.I. Du Pont De Nemours And Company | Synthetic Δ17 desaturase derived from Phytophthora ramourum and its use in making polyunsaturated fatty acids |
| US7943823B2 (en) | 2006-04-28 | 2011-05-17 | E.I. Du Pont De Nemours And Company | Delta-8 desaturase and its use in making polyunsaturated fatty acids |
| AU2007243282A1 (en) * | 2006-04-28 | 2007-11-08 | Resolvyx Pharmaceuticals, Inc. | Compositions and methods for the treatment of cardiovascular disease |
| US7678560B2 (en) | 2006-05-17 | 2010-03-16 | E.I. Du Pont De Nemours And Company | Δ 5 desaturase and its use in making polyunsaturated fatty acids |
| US7695950B2 (en) | 2006-05-17 | 2010-04-13 | E. I. Du Pont De Nemours And Company | Δ5 desaturase and its use in making polyunsaturated fatty acids |
| GB0614442D0 (en) | 2006-07-20 | 2006-08-30 | Fluxome Sciences As | Metabolically engineered cells for the production of pinosylvin |
| US9029111B2 (en) | 2006-08-24 | 2015-05-12 | Basf Plant Science Gmbh | Isolation and characterization of a novel pythium omega 3 desaturase with specificity to all omega 6 fatty acids longer than 18 carbon chains |
| BRPI0716075A2 (pt) | 2006-08-29 | 2013-08-06 | Commw Scient Ind Res Org | sÍntese de Ácidos graxos |
| US20080095711A1 (en) * | 2006-08-31 | 2008-04-24 | Falck John R | Modulators of Pulmonary Hypertension |
| CA2663768A1 (en) | 2006-10-23 | 2008-05-29 | E. I. Du Pont De Nemours And Company | Delta-8 desaturases and their use in making polyunsaturated fatty acids |
| US7709239B2 (en) * | 2006-12-07 | 2010-05-04 | E.I. Du Pont De Nemours And Company | Mutant Δ8 desaturase genes engineered by targeted mutagenesis and their use in making polyunsaturated fatty acids |
| US8846374B2 (en) * | 2006-12-12 | 2014-09-30 | E I Du Pont De Nemours And Company | Carotenoid production in a recombinant oleaginous yeast |
| US7923223B2 (en) | 2006-12-20 | 2011-04-12 | E. I. Du Pont De Nemours And Company | Δ-9 desaturase and its use in making polyunsaturated fatty acids |
| CA2675207A1 (en) | 2007-02-12 | 2008-07-21 | E. I. Du Pont De Nemours And Company | Production of arachidonic acid in oilseed plants |
| US7790156B2 (en) * | 2007-04-10 | 2010-09-07 | E. I. Du Pont De Nemours And Company | Δ-8 desaturases and their use in making polyunsaturated fatty acids |
| US8119860B2 (en) | 2007-04-16 | 2012-02-21 | E. I. Du Pont De Nemours And Company | Delta-9 elongases and their use in making polyunsaturated fatty acids |
| US8957280B2 (en) | 2007-05-03 | 2015-02-17 | E. I. Du Pont De Nemours And Company | Delta-5 desaturases and their use in making polyunsaturated fatty acids |
| AU2008255877B2 (en) * | 2007-05-25 | 2013-10-31 | Suntory Holdings Limited | Novel lysophosphatidic acid acyltransferase genes |
| MY154965A (en) | 2007-06-01 | 2015-08-28 | Solazyme Inc | Production of oil in microorganisms |
| CN101578366B (zh) * | 2007-06-18 | 2012-05-30 | 三得利控股株式会社 | 甘油-3-磷酸酰基转移酶(gpat)同源物及其应用 |
| EP2176433B1 (de) * | 2007-07-31 | 2011-10-12 | BASF Plant Science GmbH | Desaturasen und verfahren zur herstellung mehrfach ungesättigter fettsäuren in transgenen organismen |
| JP5711968B2 (ja) * | 2007-10-03 | 2015-05-07 | イー・アイ・デュポン・ドウ・ヌムール・アンド・カンパニーE.I.Du Pont De Nemours And Company | 高エイコサペンタエン酸を生成するためのヤロウィア・リポリティカ(YarrowiaLipolytica)の最適化株 |
| WO2009046248A1 (en) * | 2007-10-03 | 2009-04-09 | E. I. Du Pont De Nemours And Company | Peroxisome biogenesis factor protein (pex) disruptions for altering the content of polyunsaturated fatty acids and the total lipid content in oleaginous eukaryotic organisms |
| US8343753B2 (en) * | 2007-11-01 | 2013-01-01 | Wake Forest University School Of Medicine | Compositions, methods, and kits for polyunsaturated fatty acids from microalgae |
| US7879384B2 (en) | 2007-11-08 | 2011-02-01 | Kraft Foods Global Brands Llc | Structured glycerol esters useful as edible moisture barriers |
| US8486478B2 (en) | 2007-11-08 | 2013-07-16 | International Great Brands LLC | Structured lipid compositions |
| US8206772B2 (en) | 2007-11-08 | 2012-06-26 | Kraft Foods Global Brands Llc | Structured lipid compositions and methods of formulation thereof |
| WO2009126548A2 (en) * | 2008-04-07 | 2009-10-15 | Rich Products Corporation | Method for preparing edible aquatic animals for storage |
| US20090291479A1 (en) * | 2008-05-23 | 2009-11-26 | E. I. Du Pont De Nemours And Company | Manipulation of acyl-coa binding protein expression for altered lipid production in microbial hosts |
| US8168858B2 (en) | 2008-06-20 | 2012-05-01 | E. I. Du Pont De Nemours And Company | Delta-9 fatty acid elongase genes and their use in making polyunsaturated fatty acids |
| WO2010004784A1 (ja) * | 2008-07-10 | 2010-01-14 | 株式会社J-オイルミルズ | 飲食品の呈味向上剤 |
| DE112009002048T5 (de) * | 2008-08-26 | 2012-01-26 | Basf Plant Science Gmbh | Nukleinsäure, die Desaturasen kodieren, und modifiziertes Planzenöl |
| BRPI0913138A2 (pt) * | 2008-08-29 | 2016-05-31 | Du Pont | célula hospedeira eucariótica oleaginosa transgênica, célula de levedura eucariótica oleaginosa transgênica, óleo ou lipídeos, método para aumentar o teor de lipídeo total de uma célula hospedeira eucariótica oleaginosa e método para aumentar o teor total de ácidos graxos poliinsaturados no óleo microbiano |
| US20100297296A1 (en) * | 2008-10-14 | 2010-11-25 | Solazyme, Inc. | Healthier Baked Goods Containing Microalgae |
| US20100303989A1 (en) * | 2008-10-14 | 2010-12-02 | Solazyme, Inc. | Microalgal Flour |
| CN107858204B (zh) * | 2008-11-18 | 2021-10-29 | 联邦科学技术研究组织 | 产生ω-3脂肪酸的酶和方法 |
| MX2011005630A (es) * | 2008-11-28 | 2011-09-28 | Solazyme Inc | Producción de aceites específicos en microorganismos heterótroficos recombinantes. |
| BRPI0917722A2 (pt) | 2008-12-18 | 2017-05-30 | Du Pont | organismo transgênico e método para manipular o teor de malonatos em um organismo transgênico |
| AU2010225738B2 (en) | 2009-03-18 | 2015-01-29 | Suntory Holdings Limited | Novel acetyl-CoA carboxylase |
| KR20110139743A (ko) * | 2009-03-19 | 2011-12-29 | 로스 엔젤레스 바이오메디칼 리서치 인스티튜트 엣 하버-유씨엘에이 메디칼 센터 | 모균증 및 다른 진균 질환의 치료를 위한 백신 조성물 및 방법 |
| US8207363B2 (en) * | 2009-03-19 | 2012-06-26 | Martek Biosciences Corporation | Thraustochytrids, fatty acid compositions, and methods of making and uses thereof |
| CA3004441C (en) | 2009-03-26 | 2019-07-02 | Suntory Holdings Limited | Mortierella alpina lysophospholipid acyltransferase |
| EP2443248B1 (en) * | 2009-06-16 | 2017-12-27 | E. I. du Pont de Nemours and Company | IMPROVEMENT OF LONG CHAIN OMEGA-3 AND OMEGA-6 POLYUNSATURATED FATTY ACID BIOSYNTHESIS BY EXPRESSION OF ACYL-CoA LYSOPHOSPHOLIPID ACYLTRANSFERASES |
| WO2010147907A1 (en) * | 2009-06-16 | 2010-12-23 | E. I. Du Pont De Nemours And Company | High eicosapentaenoic acid oils from improved optimized strains of yarrowia lipolytica |
| US8524485B2 (en) * | 2009-06-16 | 2013-09-03 | E I Du Pont De Nemours And Company | Long chain omega-3 and omega-6 polyunsaturated fatty acid biosynthesis by expression of acyl-CoA lysophospholipid acyltransferases |
| CN102803289B (zh) * | 2009-06-16 | 2015-07-22 | 纳幕尔杜邦公司 | 用于高水平生产二十碳五烯酸的改善的优化的解脂耶氏酵母菌株 |
| KR101399242B1 (ko) | 2009-09-18 | 2014-05-27 | 산토리 홀딩스 가부시키가이샤 | 글리세롤-3-인산 아실기 전이 효소 |
| CA2784711A1 (en) | 2009-12-24 | 2011-06-30 | E.I. Du Pont De Nemours And Company | Plant membrane bound o-acyl transferase (mboat) family protein sequences and their uses for altering fatty acid compositions |
| US20110178105A1 (en) | 2010-01-15 | 2011-07-21 | E.I. Du Pont De Nemours And Company | Clinical benefits of eicosapentaenoic acid in humans |
| BR112012017831B8 (pt) | 2010-01-19 | 2021-05-25 | Dsm Ip Assets Bv | óleo microbiano, alimento animal e biomassa compreendendo o referido óleo microbiano |
| JP5890687B2 (ja) | 2010-02-03 | 2016-03-22 | サントリーホールディングス株式会社 | グリセロール−3−リン酸アシル基転移酵素ホモログとその利用 |
| CN103282474A (zh) | 2010-04-22 | 2013-09-04 | 纳幕尔杜邦公司 | 从微生物生物质中获得包含多不饱和脂肪酸的组合物的方法 |
| GB201008826D0 (en) | 2010-05-26 | 2010-07-14 | Fluxome Sciences As | Production of metabolites |
| MX339639B (es) | 2010-05-28 | 2016-06-02 | Solazyme Inc * | Aceites especificos producidos a partir de microorganismos heterotrofos recombinantes. |
| AU2011262568B2 (en) | 2010-06-09 | 2015-01-29 | Fermentalg | Compositions comprising eicosapentaenoic acid suitable for high purification |
| WO2012021686A1 (en) | 2010-08-11 | 2012-02-16 | E. I. Du Pont De Nemours And Company | Improved aquaculture meat products |
| WO2012021703A1 (en) | 2010-08-11 | 2012-02-16 | E. I. Du Pont De Nemours And Company | A sustainable aquaculture feeding strategy |
| US20120040076A1 (en) | 2010-08-11 | 2012-02-16 | E. I. Du Pont De Nemours And Company | Aquaculture feed compositions |
| BR112013004351A2 (pt) * | 2010-08-26 | 2016-05-31 | Du Pont | célula hospedeira microbiana recombinante e método para a produção de um óleo microbiano compreendendo ácido eicosapentaenócio. |
| JP2013535988A (ja) | 2010-08-26 | 2013-09-19 | イー・アイ・デュポン・ドウ・ヌムール・アンド・カンパニー | 変異hpggモチーフおよびhdashモチーフδ5デサチュラーゼおよび多価不飽和脂肪酸の製造におけるそれらの使用 |
| BR112013004356A2 (pt) | 2010-08-26 | 2017-06-27 | Du Pont | polinucleotídeo isolado, polipeptídeo mutante, constructo recombinante, célula transformada, método para produção de um ácido graxo poli-insaturado, óleo microbiano e célula hospedeira microbiana recombinante. |
| CN101979622B (zh) * | 2010-09-13 | 2012-10-10 | 北京凯泰新世纪生物技术有限公司 | 一种脂肪酶催化合成鱼油乙酯的方法 |
| SG10201509035WA (en) | 2010-11-03 | 2015-12-30 | Solazyme Inc | Microbial Oils With Lowered Pour Points, Dielectric Fluids Produced Therefrom, And Related Methods |
| AU2011353002A1 (en) | 2010-12-30 | 2013-05-30 | E. I. Du Pont De Nemours And Company | Use of Saccharomyces cerevisiae SUC2 gene in Yarrowia lipolytica for sucrose utilization |
| US8865429B2 (en) | 2010-12-30 | 2014-10-21 | E I Du Pont De Nemours And Company | Increased oil content by increasing Yap1 transcription factor activity in oleaginous yeasts |
| MX344012B (es) | 2011-02-02 | 2016-12-02 | Terravia Holdings Inc | Aceites adaptados producidos a partir de microorganismos oleaginosos recombinantes. |
| US20130040340A1 (en) | 2011-02-07 | 2013-02-14 | E. I. Du Pont De Nemours And Company | Production of alcohol esters in situ using alcohols and fatty acids produced by microorganisms |
| EP2673063A1 (en) | 2011-02-11 | 2013-12-18 | E.I. Du Pont De Nemours And Company | Purification of triglyceride oil from microbial sources using short path distillation |
| CA3026436A1 (en) | 2011-02-11 | 2012-08-16 | E. I. Du Pont De Nemours And Company | Method for obtaining a lipid-containing composition from microbial biomass |
| EP2691518A1 (en) | 2011-03-31 | 2014-02-05 | E.I. Du Pont De Nemours And Company | Yarrowia diacylglycerol acyltransferase promoter regions for gene expression in yeast |
| US20120247066A1 (en) | 2011-04-01 | 2012-10-04 | Ice House America, Llc | Ice bagging apparatus and methods |
| WO2012135777A1 (en) | 2011-04-01 | 2012-10-04 | E. I. Du Pont De Nemours And Company | Yarrowia esterase/lipase promoter regions for gene expression in yeast |
| US8735094B2 (en) | 2011-04-05 | 2014-05-27 | E I Du Pont De Nemours And Company | Yarrowia n-alkane-hydroxylating cytochrome P450 promoter regions for gene expression in yeast |
| US8609369B2 (en) | 2011-04-07 | 2013-12-17 | E I Du Pont De Nemours And Company | Yarrowia peroxisomal 2,4-dienoyl-CoA reductase promoter regions for gene expression in yeast |
| CN103608450A (zh) | 2011-05-06 | 2014-02-26 | 索拉兹米公司 | 基因工程改造的代谢木糖的微生物 |
| CA2833400A1 (en) * | 2011-05-26 | 2012-11-29 | E.I. Dupont De Nemours And Company | Expression of caleosin in recombinant oleaginous microorganisms to increase oil content therein |
| CN102373229B (zh) * | 2011-06-27 | 2013-09-11 | 中国农业科学院油料作物研究所 | 油脂酵母δ12和δ15双功能脂肪酸去饱和酶基因及其克隆方法 |
| CN102559710B (zh) * | 2011-06-27 | 2013-10-30 | 中国农业科学院油料作物研究所 | 等鞭金藻δ4-脂肪酸去饱和酶基因及其克隆方法 |
| EP2734626B1 (en) * | 2011-07-21 | 2020-12-16 | DSM IP Assets B.V. | Microbial oils enriched in polyunsaturated fatty acids |
| MX342216B (es) | 2011-07-21 | 2016-09-20 | Dsm Ip Assets Bv | Composiciones de acidos grasos. |
| WO2013059649A1 (en) | 2011-10-19 | 2013-04-25 | Massachusetts Institute Of Technology | Engineered microbes and methods for microbial oil production |
| JP5685783B2 (ja) * | 2012-01-19 | 2015-03-18 | 国立大学法人名古屋大学 | 新規ヤロウィア属微生物、並びにそれを用いた油分解剤及び油分解除去方法 |
| EP3550025A1 (en) | 2012-04-18 | 2019-10-09 | Corbion Biotech, Inc. | Tailored oils |
| US9719114B2 (en) | 2012-04-18 | 2017-08-01 | Terravia Holdings, Inc. | Tailored oils |
| ES2636487T3 (es) | 2012-06-15 | 2017-10-05 | Commonwealth Scientific And Industrial Research Organisation | Producción de ácidos grasos poliinsaturados de cadena larga en células vegetales |
| DK2861727T3 (en) | 2012-06-19 | 2018-06-14 | Du Pont | MUTERED ACYL-COA: LYSOPHOSPHATIDYLCHOLINE ACYL TRANSFERASES |
| SG11201501013PA (en) | 2012-08-10 | 2015-04-29 | Opx Biotechnologies Inc | Microorganisms and methods for the production of fatty acids and fatty acid derived products |
| EP2914725A4 (en) * | 2012-10-30 | 2016-04-20 | Agres Ltd | NOVEL POLYNUCLEOTIDES WITH ACYLTRANSFERASE ACTIVITY, POLYPEPTIDES AND METHODS OF USE |
| JP6064573B2 (ja) * | 2012-12-12 | 2017-01-25 | 栗田工業株式会社 | 飼料用添加物及び混合飼料 |
| WO2014100061A1 (en) | 2012-12-21 | 2014-06-26 | E. I. Du Pont De Nemours And Company | Down-regulation of a polynucleotide encoding a sou2 sorbitol utilization protein to modify lipid production in microbial cells |
| CA2893409A1 (en) | 2012-12-21 | 2014-06-26 | E. I. Du Pont De Nemours And Company | Recombinant microbial cells that produce at least 28% eicosapentaenoic acid as dry cell weight |
| PT2934505T (pt) | 2012-12-24 | 2021-07-02 | Qualitas Health Inc | Formulação de ácido eicosapentaenoico (epa) |
| US10098371B2 (en) | 2013-01-28 | 2018-10-16 | Solazyme Roquette Nutritionals, LLC | Microalgal flour |
| US9567615B2 (en) | 2013-01-29 | 2017-02-14 | Terravia Holdings, Inc. | Variant thioesterases and methods of use |
| US9816079B2 (en) | 2013-01-29 | 2017-11-14 | Terravia Holdings, Inc. | Variant thioesterases and methods of use |
| US9290749B2 (en) | 2013-03-15 | 2016-03-22 | Solazyme, Inc. | Thioesterases and cells for production of tailored oils |
| WO2014146026A1 (en) | 2013-03-15 | 2014-09-18 | Opx Biotechnologies, Inc. | Bioproduction of chemicals |
| US9783836B2 (en) | 2013-03-15 | 2017-10-10 | Terravia Holdings, Inc. | Thioesterases and cells for production of tailored oils |
| WO2014176515A2 (en) | 2013-04-26 | 2014-10-30 | Solazyme, Inc. | Low polyunsaturated fatty acid oils and uses thereof |
| US11408013B2 (en) | 2013-07-19 | 2022-08-09 | Cargill, Incorporated | Microorganisms and methods for the production of fatty acids and fatty acid derived products |
| JP6603658B2 (ja) | 2013-07-19 | 2019-11-06 | カーギル インコーポレイテッド | 脂肪酸及び脂肪酸誘導体の製造のための微生物及び方法 |
| FR3009619B1 (fr) | 2013-08-07 | 2017-12-29 | Roquette Freres | Compositions de biomasse de microalgues riches en proteines de qualite sensorielle optimisee |
| EP3030648B1 (en) | 2013-08-08 | 2020-01-08 | Knipbio, Inc. | Methylotrophs for aquaculture and animal feed |
| WO2015025920A1 (ja) * | 2013-08-22 | 2015-02-26 | 協和発酵バイオ株式会社 | アラキドン酸生産ポリケチドシンターゼ及びその利用 |
| AU2014331605A1 (en) | 2013-10-04 | 2016-05-12 | Corbion Biotech, Inc. | Tailored oils |
| ES2875965T3 (es) | 2013-12-04 | 2021-11-11 | Nippon Suisan Kaisha Ltd | Aceite microbiano que contiene ácido dihomo-gamma-linolénico y biomasa microbiana que contiene ácido dihomo-gamma-linolénico |
| EA037817B1 (ru) | 2013-12-18 | 2021-05-25 | Коммонвелт Сайнтифик Энд Индастриэл Рисерч Организэйшн | Экстрагированный растительный липид, содержащий длинноцепочечные полиненасыщенные жирные кислоты |
| EP3102683B1 (en) | 2014-01-31 | 2019-10-23 | DSM IP Assets B.V. | Promoters suitable for heterologous gene expression in yeast |
| AU2015217064B2 (en) | 2014-02-12 | 2017-05-04 | OmniGen Research, L.L.C. | Composition and method for promoting reduction of heat stress in animals |
| CN113151319A (zh) * | 2014-02-24 | 2021-07-23 | 银杏生物制品公司 | 二酰基甘油酰基转移酶(dga1)多核苷酸,和通过异源dga1的超量表达增加酵母细胞脂质生产的方法 |
| WO2015149026A1 (en) | 2014-03-28 | 2015-10-01 | Solazyme, Inc. | Lauric ester compositions |
| US10443047B2 (en) * | 2014-05-01 | 2019-10-15 | Novogy, Inc. | Increasing cellular lipid production by increasing the activity of diacylglycerol acyltransferase and decreasing the activity of triacylglycerol lipase |
| US10260077B2 (en) | 2014-05-29 | 2019-04-16 | Novogy, Inc. | Increasing lipid production in oleaginous yeast |
| EP3160482A4 (en) | 2014-06-27 | 2018-02-14 | Commonwealth Scientific and Industrial Research Organisation | Lipid comprising docosapentaenoic acid |
| BR112017000414A2 (pt) | 2014-07-10 | 2017-11-07 | Terravia Holdings Inc | genes cetoacil-acp sintase e utilizações dos mesmos |
| US9765368B2 (en) | 2014-07-24 | 2017-09-19 | Terravia Holdings, Inc. | Variant thioesterases and methods of use |
| EP2993228B1 (en) | 2014-09-02 | 2019-10-09 | Cargill, Incorporated | Production of fatty acid esters |
| EP3194582A2 (en) | 2014-09-18 | 2017-07-26 | TerraVia Holdings, Inc. | Acyl-acp thioesterases and mutants thereof |
| FR3028527A1 (fr) | 2014-11-13 | 2016-05-20 | Pivert | Identification de facteurs de transcription de yarrowia lipolytica |
| WO2016075314A1 (fr) | 2014-11-13 | 2016-05-19 | Institut National De La Recherche Agronomique | Identification de facteurs de transcription de yarrowia lipolytica affectant la production de proteines |
| CN107208088B (zh) * | 2014-12-10 | 2022-02-11 | 银杏生物制品公司 | 酵母中的油酸生产 |
| WO2016092071A1 (en) | 2014-12-12 | 2016-06-16 | Dsm Ip Assets B.V. | Feed supplement material for use in aquaculture feed |
| CA2987508A1 (en) | 2015-05-28 | 2016-12-01 | Evolva Sa | Biosynthesis of phenylpropanoids and phenylpropanoid derivatives |
| CN105176848B (zh) * | 2015-08-19 | 2017-05-17 | 江南大学 | 一株过表达3‑磷酸甘油脱氢酶基因的高山被孢霉、其构建方法及应用 |
| WO2017044832A1 (en) | 2015-09-09 | 2017-03-16 | Omnigen Research, Llc | A composition and/or combination for aquaculture |
| AU2016333440A1 (en) | 2015-10-01 | 2017-06-15 | Dsm Ip Assets B.V. | Supplement material for use in pet food |
| JP7011590B2 (ja) | 2015-10-12 | 2022-02-10 | イー・アイ・デュポン・ドウ・ヌムール・アンド・カンパニー | 細胞内での遺伝子組換えおよび相同組換えの増加のための保護dna鋳型および使用方法 |
| EP3387134B1 (en) | 2015-12-11 | 2020-10-14 | Danisco US Inc. | Methods and compositions for enhanced nuclease-mediated genome modification and reduced off-target site effects |
| EP3708665A1 (en) | 2015-12-18 | 2020-09-16 | Danisco US Inc. | Methods and compositions for t-rna based guide rna expression |
| CN106434416B (zh) * | 2016-07-28 | 2019-08-30 | 中国农业科学院油料作物研究所 | 一种产二十碳五烯酸的菌株及其应用 |
| JP2020506702A (ja) | 2017-02-02 | 2020-03-05 | カーギル インコーポレイテッド | C6−c10脂肪酸誘導体を生成する遺伝子組み換え細胞 |
| CN107345211A (zh) * | 2017-04-27 | 2017-11-14 | 广州弘宝元生物科技有限公司 | 引入外源多肽的活细胞脂质体及其应用 |
| US11560583B2 (en) | 2017-06-01 | 2023-01-24 | Knipbio, Inc. | Heterologous carotenoid production in microorganisms |
| CN109234216B (zh) * | 2017-07-10 | 2022-05-20 | 上海医药工业研究院 | 一种生产鲨烯的基因工程菌及其方法 |
| CN108467841B (zh) * | 2018-03-23 | 2021-08-24 | 辽宁大学 | 蛭石固定化耐低温降解多环芳烃混合菌颗粒及其制备方法和应用 |
| CN110358692B (zh) | 2018-04-09 | 2021-07-27 | 中国科学院青岛生物能源与过程研究所 | 生产神经酸的重组酵母菌株及其应用 |
| WO2019209241A1 (en) | 2018-04-23 | 2019-10-31 | Dupont Nutrition Biosciences Aps | Increasing export of 2' fucosyllactose from microbial cells through the expression of a heterologous nucleic acid |
| CN110373437B (zh) * | 2018-12-11 | 2022-09-27 | 山东理工大学 | 一种产十八碳四烯酸卷枝毛霉细胞工厂的构建及其发酵技术 |
| CN109929870B (zh) * | 2019-02-20 | 2021-03-16 | 天津大学 | 糖代谢与脂质代谢协同提高解脂耶氏酵母合成脂肪酸衍生物的产量的应用 |
| CN109837256B (zh) * | 2019-03-21 | 2020-12-01 | 江南大学 | 一种二酰甘油酰基转移酶1及其在生产甘油三酯中的应用 |
| CN110229832A (zh) * | 2019-06-16 | 2019-09-13 | 山东理工大学 | 一种提高耶式解脂酵母生物量和细胞壁产量的菌株和方法 |
| CN110923261B (zh) * | 2019-12-20 | 2021-12-17 | 江南大学 | 一种提高酿酒酵母细胞膜脂肪酸c20:0和/或c22:0含量的方法 |
| WO2021130078A1 (en) | 2019-12-23 | 2021-07-01 | Dsm Ip Assets B.V. | Aquaculture feed |
| CN112538438B (zh) * | 2020-12-11 | 2023-04-04 | 南京工业大学 | 一株高产油酸的重组解脂耶氏酵母菌及其构建方法和应用 |
| WO2022129592A1 (en) | 2020-12-17 | 2022-06-23 | Dsm Ip Assets B.V. | Aquaculture feed |
| WO2022178302A1 (en) * | 2021-02-18 | 2022-08-25 | Zero Acre Farms Inc. | Systems and methods of making oil from microorganisms |
| AU2022231106A1 (en) * | 2021-03-03 | 2023-10-12 | Nourish Ingredients Pty Ltd | Production of phospholipids in microbes and uses thereof |
| CN113388533B (zh) * | 2021-06-11 | 2022-09-20 | 东北农业大学 | 一株发酵性能良好且具有产香功能的汉逊德巴利酵母菌及其筛选方法 |
| WO2023105498A1 (en) * | 2021-12-10 | 2023-06-15 | Phycoil Biotechnology International, Inc. | Heterotrophic production of essential long-chain polyunsaturated lipids (lcpufa) in auxenochlorella protothecoides |
| WO2023148187A1 (en) | 2022-02-02 | 2023-08-10 | Dsm Ip Assets B.V. | Yarrowia production process |
| CN115029257B (zh) * | 2022-05-05 | 2023-09-26 | 南京工业大学 | 产β-胡萝卜素的重组解脂耶氏酵母及其构建方法和应用 |
Family Cites Families (118)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| US2454404A (en) | 1942-10-03 | 1948-11-23 | American Cyanamid Co | Preparation of piperazine |
| US2454504A (en) | 1946-01-29 | 1948-11-23 | Franz F Ehrenhaft | Nonintermittent cinematographic projector |
| NZ190194A (en) | 1978-05-08 | 1982-02-23 | Cpc International Inc | Production of oil by fermenting yeast in fatty acids |
| US4670285A (en) | 1982-08-06 | 1987-06-02 | The University Of Toronto Innovations Foundation | Infant formula |
| US5071764A (en) | 1983-10-06 | 1991-12-10 | Pfizer Inc. | Process for integrative transformation of yarrowia lipolytica |
| US4880741A (en) | 1983-10-06 | 1989-11-14 | Pfizer Inc. | Process for transformation of Yarrowia lipolytica |
| GB8407195D0 (en) | 1984-03-20 | 1984-04-26 | Cadbury Schweppes Plc | Microbial desaturase enzyme inhibitors |
| US4792523A (en) | 1984-08-31 | 1988-12-20 | Cetus Corporation | 3'Expression enhancing fragments and method |
| DD227448A1 (de) | 1984-10-18 | 1985-09-18 | Akad Wissenschaften Ddr | Verfahren zur herstellung von zitronensaeure auf mikrobiellem wege |
| JPH075484B2 (ja) | 1985-03-06 | 1995-01-25 | 帝人株式会社 | 血液脳関門通過性医薬品組成物 |
| US5107065A (en) | 1986-03-28 | 1992-04-21 | Calgene, Inc. | Anti-sense regulation of gene expression in plant cells |
| DE3783396T2 (de) * | 1986-07-08 | 1993-07-29 | Suntory Ltd | Verfahren zur herstellung von bishomo-gamma-linolensaeure und eicosapentaensaeure. |
| JP2527340B2 (ja) | 1986-12-15 | 1996-08-21 | アプライド バイオシステムズ インコーポレイテッド | ロ―ダミン染料の5−及び6−スクシニミジルカルボキシレ―ト異性体 |
| US5246841A (en) * | 1986-12-26 | 1993-09-21 | Sagami Chemical Research Center | Microbial process for production of eicosapentaenoic acid |
| DD259637A1 (de) | 1987-04-09 | 1988-08-31 | Adw Ddr | Verfahren zur herstellung von isozitratlyase in rekombinanten mikroorganismen |
| SU1454852A1 (ru) | 1987-05-19 | 1989-01-30 | Институт Микробиологии Им.Августа Кирхенштейна | Штамм дрожжей JaRRoWIa LIроLYтIса-продуцент липазы и питательна среда дл его культивировани |
| JP2582622B2 (ja) | 1987-10-27 | 1997-02-19 | 日東化学工業株式会社 | 糸状菌による高度不飽和脂肪酸の製造法 |
| DD267999B5 (de) | 1988-01-06 | 2000-08-10 | Akad Wissenschaften Ddr | Verfahren zur herstellung von 2-oxoglutarsaeure durch hefen |
| US5340742A (en) | 1988-09-07 | 1994-08-23 | Omegatech Inc. | Process for growing thraustochytrium and schizochytrium using non-chloride salts to produce a microfloral biomass having omega-3-highly unsaturated fatty acids |
| DD275480A1 (de) | 1988-09-15 | 1990-01-24 | Ve Forschungszentrum Biotechno | Verfahren zur herstellung von zitronensaeure durch aerobe submerse kultivierung von hefen |
| US5057419A (en) | 1988-09-22 | 1991-10-15 | Rutgers University | Genetically engineered plasmid and organisms for the production of specialized oils |
| DD279267A1 (de) | 1989-01-05 | 1990-05-30 | Akad Wissenschaften Ddr | Verfahren zur herstellung mikrobieller lipase |
| PL160027B1 (pl) | 1989-03-23 | 1993-01-29 | Akad Rolnicza | Sposób jednoczesnego otrzymywania kwasu cytrynowego i kwasu izocytrynowego PL |
| DD285372A5 (de) | 1989-06-29 | 1990-12-12 | Ingenieurtechnik,Dd | Verfahren zur immobilisierung von mikroorganismen bzw. coimmobilisierung von mikroorganismen und enzymen |
| DD285370A5 (de) | 1989-06-29 | 1990-12-12 | Ingenieurtechnik Im Stammbetrieb Veb Kombinat Suesswaren,Dd | Verfahren zur immobilisierung von mikroorganismen bzw. coimmobilisierung von mikroorganismen und enzymen |
| US5366860A (en) | 1989-09-29 | 1994-11-22 | Applied Biosystems, Inc. | Spectrally resolvable rhodamine dyes for nucleic acid sequence determination |
| JP3007119B2 (ja) | 1990-01-12 | 2000-02-07 | 紀夫 村田 | グリセロール―3―リン酸アシルトランスフェラーゼをコードするdna鎖 |
| ATE241007T1 (de) | 1990-03-16 | 2003-06-15 | Calgene Llc | Dnas, die für pflanzliche desaturasen kodieren und deren anwendungen |
| US5244921A (en) * | 1990-03-21 | 1993-09-14 | Martek Corporation | Eicosapentaenoic acids and methods for their production |
| DE69103753T3 (de) | 1990-05-25 | 2008-01-24 | E.I. Dupont De Nemours And Co., Wilmington | Nukleotidsequenz des gens für stearoyl-acp-desaturase aus soja. |
| US5658767A (en) | 1991-01-24 | 1997-08-19 | Martek Corporation | Arachidonic acid and methods for the production and use thereof |
| US5246842A (en) | 1991-10-30 | 1993-09-21 | The United States Of America As Represented By The Secretary Of Agriculture | Production of eicosapentaenoic acid from filamentous fungi utilizing lactose as a primary carbon source |
| AU675923B2 (en) | 1991-12-04 | 1997-02-27 | E.I. Du Pont De Nemours And Company | Fatty acid desaturase genes from plants |
| US5246642A (en) * | 1992-02-04 | 1993-09-21 | Slaughter Jr Gibbs M | Method for resurfacing fiberglass boat hulls |
| JPH05308978A (ja) | 1992-05-11 | 1993-11-22 | Onoda Cement Co Ltd | 藻類によるドコサヘキサエン酸の製造方法 |
| DE578388T1 (de) | 1992-06-25 | 1995-03-16 | Int Flavors & Fragrances Inc | Verfahren zur fermentativen Herstellung von 10-Hydroxy-C18-Carbonsäure und Gamma-Dodekalaktonderivaten. |
| IT1256043B (it) | 1992-08-12 | 1995-11-21 | Ceppo mutante di yarrowia lipolytica e procedimento per la produzione di acido citrico | |
| WO1994011516A1 (en) | 1992-11-17 | 1994-05-26 | E.I. Du Pont De Nemours And Company | Genes for microsomal delta-12 fatty acid desaturases and related enzymes from plants |
| US6093568A (en) | 1994-04-06 | 2000-07-25 | Calgene, Inc. | Plant lysophosphatidic acid acyltransferases |
| US5910630A (en) | 1994-04-06 | 1999-06-08 | Davies; Huw Maelor | Plant lysophosphatidic acid acyltransferases |
| WO1996033263A1 (fr) | 1995-04-17 | 1996-10-24 | JAPAN, represented by DIRECTOR-GENERAL OF AGENCY OF INDUSTRIAL SCIENCE AND TECHNOLOGY | Nouveaux micro-organismes capables de produire des acides gras hautement insatures et procede de production d'acides gras hautement insatures utilisant ces micro-organismes |
| FR2734843A1 (fr) | 1995-06-02 | 1996-12-06 | Centre Nat Rech Scient | Nouveau procede de bioconversion microbienne. |
| EP0770683A1 (en) | 1995-10-04 | 1997-05-02 | Mitsubishi Chemical Corporation | Method for producing erythritol |
| JPH09252790A (ja) | 1996-03-21 | 1997-09-30 | Japan Energy Corp | 発酵法によるピルビン酸の製造方法 |
| DK0894142T4 (da) * | 1996-03-28 | 2014-02-24 | Dsm Ip Assets Bv | Mikrobiel olie der omfatter en polyumættet fedtsyre og proces til produktion af olie fra pasteuriseret og granuleret biomasse. |
| AU2887897A (en) | 1996-05-21 | 1997-12-09 | Novo Nordisk A/S | Novel yeast promoters suitable for expression cloning in yeast and heterologous expression of proteins in yeast |
| EE04063B1 (et) | 1996-07-23 | 2003-06-16 | Nagase Biochemicals, Ltd. | Meetod dokosaheksaeenhappe ning dokosapentaeenhappe valmistamiseks |
| JP3792309B2 (ja) * | 1996-08-30 | 2006-07-05 | サントリー株式会社 | 不飽和脂肪酸含有油脂の製造方法 |
| RU2090611C1 (ru) | 1996-09-27 | 1997-09-20 | Финогенова Татьяна Васильевна | Штамм дрожжей yarrowia lipolytica - продуцент лимонной кислоты, способ получения лимонной кислоты и способ выделения цитрата натрия |
| PL190181B1 (pl) * | 1996-10-11 | 2005-11-30 | Scarista Ltd | Zastosowanie oleju zawierającego kwas eikozapentaenowy (KEP) i/lub kwas stearydonowy (KS) oraz preparat farmaceutyczny zawierający olej |
| US6432684B1 (en) * | 1997-04-11 | 2002-08-13 | Abbott Laboratories | Human desaturase gene and uses thereof |
| US6075183A (en) | 1997-04-11 | 2000-06-13 | Abbott Laboratories | Methods and compositions for synthesis of long chain poly-unsaturated fatty acids in plants |
| US5968809A (en) | 1997-04-11 | 1999-10-19 | Abbot Laboratories | Methods and compositions for synthesis of long chain poly-unsaturated fatty acids |
| US5972664A (en) | 1997-04-11 | 1999-10-26 | Abbott Laboratories | Methods and compositions for synthesis of long chain poly-unsaturated fatty acids |
| JP2002503447A (ja) | 1997-12-04 | 2002-02-05 | スミスクライン・ビーチャム・コーポレイション | 発現可能なアンチセンス配列を用いる条件発現変異細胞の取得方法 |
| US6344548B1 (en) | 1998-06-24 | 2002-02-05 | The Regents Of The University Of California | Diacylglycerol o-acyltransferase |
| US20030167483A1 (en) | 1998-06-24 | 2003-09-04 | Farese Robert V. | Diacylglycerol O-acyltransferase |
| US7135617B2 (en) | 1998-07-02 | 2006-11-14 | Calgene Llc | Diacylglycerol acyl transferase proteins |
| EP1098962B1 (en) | 1998-07-02 | 2009-09-09 | Calgene LLC | Diacylglycerol acyl transferase proteins |
| US6677145B2 (en) | 1998-09-02 | 2004-01-13 | Abbott Laboratories | Elongase genes and uses thereof |
| US6403349B1 (en) | 1998-09-02 | 2002-06-11 | Abbott Laboratories | Elongase gene and uses thereof |
| US6100077A (en) | 1998-10-01 | 2000-08-08 | The Trustees Of Columbia University In The City Of New York | Isolation of a gene encoding diacylglycerol acyltransferase |
| EP0997533A1 (de) | 1998-10-24 | 2000-05-03 | Haarmann & Reimer Gmbh | Verfahren zur Gewinnung von Gamma-Decalacton |
| FR2785911B1 (fr) | 1998-11-18 | 2001-01-26 | Agronomique Inst Nat Rech | Gene codant pour une acyltransferase de colza, et ses utilisations |
| AU1837600A (en) | 1998-12-02 | 2000-06-19 | E.I. Du Pont De Nemours And Company | Plant diacylglycerol acyltransferases |
| CA2791961A1 (en) | 1998-12-07 | 2000-06-15 | Washington State University Research Foundation | Desaturases and methods of using them for synthesis of polyunsaturated fatty acids |
| CN1352690A (zh) * | 1998-12-17 | 2002-06-05 | 加拿大国家研究局 | 来源于植物的二酯酰甘油酰基转移酶基因 |
| WO2000049156A2 (en) | 1999-02-22 | 2000-08-24 | E.I. Du Pont De Nemours And Company | Lysophosphatidic acid acetyltransferases |
| EP1165803B1 (en) | 1999-04-01 | 2007-03-07 | BASF Plant Science GmbH | Enzymes of the biosynthetic pathway for the production of triacylglycerol and recombinant dna molecules encoding these enzymes |
| KR19990046733A (ko) | 1999-04-20 | 1999-07-05 | 류성구 | 미생물슈도모나스에의한도코사핵사노인산(dha)의제조방법 |
| ES2280261T3 (es) | 1999-11-12 | 2007-09-16 | Basf Plant Science Gmbh | Utilizacion de una clase de enzimas y de sus genes codificantes para aumentar el contenido de aceite de organismos transgenicos. |
| AU2001239244B2 (en) | 2000-02-09 | 2006-06-08 | Basf Aktiengesellschaft | Novel elongase gene and method for producing multiple-unsaturated fatty acids |
| WO2001083773A1 (fr) | 2000-04-28 | 2001-11-08 | Laboratoires Mayoly Spindler | Clonage et expression d'une lipase extracellulaire acidoresistante de yarrowia lipolytica |
| EP1283266A4 (en) | 2000-05-19 | 2005-01-05 | Kaneka Corp | TRANSFORMANT AND PROCESS FOR PRODUCING POLYESTER USING THE TRANSFORMANT |
| US6552250B1 (en) | 2000-06-14 | 2003-04-22 | Her Majesty The Queen In Right Of Canada As Represented By The Minister Of Agriculture And Agri-Food | Diacylglycerol O-acyltransferase |
| DE10102337A1 (de) * | 2001-01-19 | 2002-07-25 | Basf Plant Science Gmbh | Verfahren zur Herstellung mehrfach ungesättigter Fettsäuren, neue Biosynthesegene sowie neue pflanzliche Expressionskonstrukte |
| US6635451B2 (en) * | 2001-01-25 | 2003-10-21 | Abbott Laboratories | Desaturase genes and uses thereof |
| GB2377455A (en) | 2001-02-09 | 2003-01-15 | Univ Hull | Method of culturing crypthecodinium cohnii |
| US7045326B2 (en) | 2001-02-23 | 2006-05-16 | The Regents Of The University Of California | Mono- and diacylglycerol acyltransferases and methods of use thereof |
| GB0107510D0 (en) | 2001-03-26 | 2001-05-16 | Univ Bristol | New elongase gene and a process for the production of -9-polyunsaturated fatty acids |
| JP2002296554A (ja) | 2001-03-30 | 2002-10-09 | Minebea Co Ltd | ファラデー回転子 |
| US7045683B2 (en) * | 2001-05-04 | 2006-05-16 | Abbott Laboratories | Δ4-desaturase genes and uses thereof |
| US20060206960A1 (en) | 2001-09-21 | 2006-09-14 | Jitao Zou | Higher plant cytosolic er-based glycerol-3-phosphate acyltransferase genes |
| US7211656B2 (en) * | 2002-01-30 | 2007-05-01 | Abbott Laboratories | Desaturase genes, enzymes encoded thereby, and uses thereof |
| DE10219203A1 (de) * | 2002-04-29 | 2003-11-13 | Basf Plant Science Gmbh | Verfahren zur Herstellung mehrfach ungesättigter Fettsäuren in Pflanzen |
| KR101138631B1 (ko) | 2002-05-22 | 2012-04-26 | 몬산토 테크놀로지 엘엘씨 | 진균 기원의 지방산 데사투라제 |
| US7470527B2 (en) * | 2002-06-19 | 2008-12-30 | Dsm Ip Assets B.V. | Preparation of microbial oil |
| AU2003259682A1 (en) | 2002-07-31 | 2004-02-16 | Monsanto Technology, Llc | Diacylglycerol acyltransferase nucleic acid sequences and associated products |
| BR0317304A (pt) | 2002-12-19 | 2005-11-08 | Univ Bristol | Processo para a produção de compostos, sequência de ácido nucleico isolada, sequência de aminoácidos, construção gênica, vetor, e, organismo |
| JP4280158B2 (ja) | 2002-12-27 | 2009-06-17 | 富士フイルム株式会社 | ドコサヘキサエン酸生産能を有する微生物及びその利用 |
| US20040172682A1 (en) * | 2003-02-12 | 2004-09-02 | Kinney Anthony J. | Production of very long chain polyunsaturated fatty acids in oilseed plants |
| US7537920B2 (en) | 2003-02-27 | 2009-05-26 | Basf Plant Science Gmbh | Method for the production of polyunsaturated fatty acids |
| WO2004087902A2 (de) * | 2003-03-31 | 2004-10-14 | University Of Bristol | Neue pflanzliche acyltransferasen spezifisch für langkettige mehrfach ungesättigte fettsäuren |
| US7238482B2 (en) | 2003-05-07 | 2007-07-03 | E. I. Du Pont De Nemours And Company | Production of polyunsaturated fatty acids in oleaginous yeasts |
| US7125672B2 (en) * | 2003-05-07 | 2006-10-24 | E. I. Du Pont De Nemours And Company | Codon-optimized genes for the production of polyunsaturated fatty acids in oleaginous yeasts |
| US7214491B2 (en) * | 2003-05-07 | 2007-05-08 | E. I. Du Pont De Nemours And Company | Δ-12 desaturase gene suitable for altering levels of polyunsaturated fatty acids in oleaginous yeasts |
| DE602004022806D1 (de) | 2003-06-04 | 2009-10-08 | Nestec Sa | Getränk zur gewichtskontrolle |
| KR101180600B1 (ko) | 2003-06-04 | 2012-09-06 | 세키스이가가쿠 고교가부시키가이샤 | 경화성 수지 조성물의 제조 방법 |
| AU2003903058A0 (en) | 2003-06-18 | 2003-07-03 | Phytopharm Australia Ltd | A process and apparatus for the modification of plant extracts |
| US7259255B2 (en) | 2003-06-25 | 2007-08-21 | E. I. Du Pont De Nemours And Company | Glyceraldehyde-3-phosphate dehydrogenase and phosphoglycerate mutase promoters for gene expression in oleaginous yeast |
| US7459546B2 (en) * | 2003-06-25 | 2008-12-02 | E.I. Du Pont De Nemours And Company | Glyceraldehyde-3-phosphate dehydrogenase and phosphoglycerate mutase regulatory sequences for gene expression in oleaginous yeast |
| US7267976B2 (en) | 2003-07-02 | 2007-09-11 | E.I. Du Pont De Nemours And Company | Acyltransferases for alteration of polyunsaturated fatty acids and oil content in oleaginous yeasts |
| EP2172536B1 (de) * | 2003-08-01 | 2016-05-04 | BASF Plant Science GmbH | Verfahren zur Herstellung mehrfach ungesättigter Fettsäuren in transgenen Organismen |
| WO2005047480A2 (en) | 2003-11-12 | 2005-05-26 | E.I. Dupont De Nemours And Company | Delta-15 desaturases suitable for altering levels of polyunsaturated fatty acids in oleaginous plants and yeast |
| US7504259B2 (en) * | 2003-11-12 | 2009-03-17 | E. I. Du Pont De Nemours And Company | Δ12 desaturases suitable for altering levels of polyunsaturated fatty acids in oleaginous yeast |
| WO2005049805A2 (en) | 2003-11-14 | 2005-06-02 | E.I. Dupont De Nemours And Company | Fructose-bisphosphate aldolase regulatory sequences for gene expression in oleaginous yeast |
| US7264408B2 (en) | 2004-04-28 | 2007-09-04 | Finisar Corporation | Modular optical device package |
| WO2006012326A1 (en) * | 2004-06-25 | 2006-02-02 | E.I. Dupont De Nemours And Company | Delta-8 desaturase and its use in making polyunsaturated fatty acids |
| US7264949B2 (en) * | 2004-09-15 | 2007-09-04 | E.I. Du Pont De Nemours And Company | Glycerol-3-phosphate o-acyltransferase promoter for gene expression in oleaginous yeast |
| US7198937B2 (en) | 2004-11-04 | 2007-04-03 | E. I. Du Pont De Nemours And Company | Mortierella alpina diacylglycerol acyltransferase for alteration of polyunsaturated fatty acids and oil content in oleaginous organisms |
| US8685679B2 (en) | 2004-11-04 | 2014-04-01 | E I Du Pont De Nemours And Company | Acyltransferase regulation to increase the percent of polyunsaturated fatty acids in total lipids and oils of oleaginous organisms |
| US7189559B2 (en) | 2004-11-04 | 2007-03-13 | E. I. Du Pont De Nemours And Company | Mortierella alpina lysophosphatidic acid acyltransferase homolog for alteration of polyunsaturated fatty acids and oil content in oleaginous organisms |
| US7192762B2 (en) | 2004-11-04 | 2007-03-20 | E. I. Du Pont De Nemours And Company | Mortierella alpina glycerol-3-phosphate o-acyltransferase for alteration of polyunsaturated fatty acids and oil content in oleaginous organisms |
| US7588931B2 (en) | 2004-11-04 | 2009-09-15 | E. I. Du Pont De Nemours And Company | High arachidonic acid producing strains of Yarrowia lipolytica |
| US7273746B2 (en) | 2004-11-04 | 2007-09-25 | E.I. Dupont De Nemours And Company | Diacylglycerol acyltransferases for alteration of polyunsaturated fatty acids and oil content in oleaginous organisms |
| US20060094102A1 (en) | 2004-11-04 | 2006-05-04 | Zhixiong Xue | Ammonium transporter promoter for gene expression in oleaginous yeast |
| EP1828395A1 (en) | 2004-12-14 | 2007-09-05 | Avestha Gengraine Technologies PVT Ltd | Recombinant production docosahexaenoic acid (dha) in yeast |
| US7470532B2 (en) | 2005-10-19 | 2008-12-30 | E.I. Du Pont De Nemours And Company | Mortierella alpina C16/18 fatty acid elongase |
-
2005
- 2005-11-01 US US11/264,784 patent/US7588931B2/en not_active Expired - Fee Related
- 2005-11-01 US US11/264,737 patent/US7550286B2/en not_active Expired - Fee Related
- 2005-11-02 US US11/265,761 patent/US7932077B2/en not_active Expired - Fee Related
- 2005-11-03 CN CNA2005800457798A patent/CN101437951A/zh active Pending
- 2005-11-03 CN CN200580045823.5A patent/CN101111601B/zh not_active Expired - Fee Related
- 2005-11-03 JP JP2007540127A patent/JP5080979B2/ja not_active Expired - Fee Related
- 2005-11-03 JP JP2007540126A patent/JP5129574B2/ja not_active Expired - Fee Related
- 2005-11-03 KR KR1020077012480A patent/KR20070085669A/ko not_active Application Discontinuation
- 2005-11-03 EP EP12152258.5A patent/EP2458000B1/en not_active Expired - Fee Related
- 2005-11-03 KR KR1020077012441A patent/KR20070085649A/ko not_active Application Discontinuation
- 2005-11-03 CA CA2584719A patent/CA2584719C/en not_active Expired - Fee Related
- 2005-11-03 WO PCT/US2005/040256 patent/WO2006052871A2/en active Application Filing
- 2005-11-03 DK DK05851406.8T patent/DK1807527T3/da active
- 2005-11-03 WO PCT/US2005/040306 patent/WO2006055322A2/en active Application Filing
- 2005-11-03 JP JP2007540139A patent/JP5139806B2/ja not_active Expired - Fee Related
- 2005-11-03 CA CA2585235A patent/CA2585235C/en not_active Expired - Fee Related
- 2005-11-03 EP EP05818267A patent/EP1807526A4/en not_active Withdrawn
- 2005-11-03 CN CN2005800459172A patent/CN101437953B/zh not_active Expired - Fee Related
- 2005-11-03 EP EP05851406.8A patent/EP1807527B1/en not_active Expired - Fee Related
- 2005-11-03 DK DK05818385.6T patent/DK1809756T3/en active
- 2005-11-03 KR KR1020077012473A patent/KR20070085665A/ko not_active Application Discontinuation
- 2005-11-03 EP EP13161757.3A patent/EP2649887B1/en not_active Expired - Fee Related
- 2005-11-03 WO PCT/US2005/040255 patent/WO2006052870A2/en active Application Filing
- 2005-11-03 CA CA2585178A patent/CA2585178C/en not_active Expired - Fee Related
- 2005-11-03 DK DK13161757.3T patent/DK2649887T3/en active
- 2005-11-03 DK DK12152258.5T patent/DK2458000T3/da active
- 2005-11-03 EP EP05818385.6A patent/EP1809756B1/en not_active Expired - Fee Related
- 2005-11-03 CN CNA2005800459100A patent/CN101437952A/zh active Pending
-
2007
- 2007-05-15 NO NO20072517A patent/NO20072517L/no not_active Application Discontinuation
- 2007-05-16 NO NO20072516A patent/NO20072516L/no not_active Application Discontinuation
- 2007-05-21 NO NO20072575A patent/NO20072575L/no not_active Application Discontinuation
-
2009
- 2009-05-14 US US12/466,072 patent/US8685682B2/en not_active Expired - Fee Related
- 2009-07-30 US US12/512,507 patent/US20100022647A1/en not_active Abandoned
-
2010
- 2010-09-29 US US12/893,469 patent/US8518674B2/en active Active
-
2012
- 2012-11-13 US US13/675,144 patent/US20130123361A1/en not_active Abandoned
-
2013
- 2013-07-17 US US13/943,992 patent/US8815566B2/en active Active
Cited By (4)
| Publication number | Priority date | Publication date | Assignee | Title |
|---|---|---|---|---|
| CN104099254A (zh) * | 2014-07-11 | 2014-10-15 | 无锡超科食品有限公司 | 一株产多不饱和脂肪酸的菌株及其筛选方法 |
| CN111685223A (zh) * | 2019-03-13 | 2020-09-22 | 上海吉态来生物技术有限公司 | 含有解脂亚罗酵母的饲料、其制备方法及其应用 |
| CN111685223B (zh) * | 2019-03-13 | 2024-02-02 | 吉态来博(北京)生物科技发展有限公司 | 含有解脂亚罗酵母的饲料、其制备方法及其应用 |
| CN110499259A (zh) * | 2019-07-22 | 2019-11-26 | 浙江工业大学 | 一种解酯耶氏酵母yw100-1及其应用 |
Also Published As
Similar Documents
| Publication | Publication Date | Title |
|---|---|---|
| CN101111601B (zh) | 解脂耶氏酵母的高二十碳五烯酸生产菌株 | |
| JP2016101172A (ja) | 改良されたヤロウイア・リポリティカ(yarrowialipolytica)最適化株による高エイコサペンタエン酸油 | |
| JP5893555B2 (ja) | エイコサペンタエン酸を高産生するよう改良されたヤロウイア・リポリティカ(Yarrowia lipolytica)最適化株 | |
| JP2012529913A6 (ja) | エイコサペンタエン酸を高産生するよう改良されたヤロウイア・リポリティカ(Yarrowia lipolytica)最適化株 |
Legal Events
| Date | Code | Title | Description |
|---|---|---|---|
| C06 | Publication | ||
| PB01 | Publication | ||
| C10 | Entry into substantive examination | ||
| SE01 | Entry into force of request for substantive examination | ||
| C02 | Deemed withdrawal of patent application after publication (patent law 2001) | ||
| WD01 | Invention patent application deemed withdrawn after publication |
Open date: 20090520 |